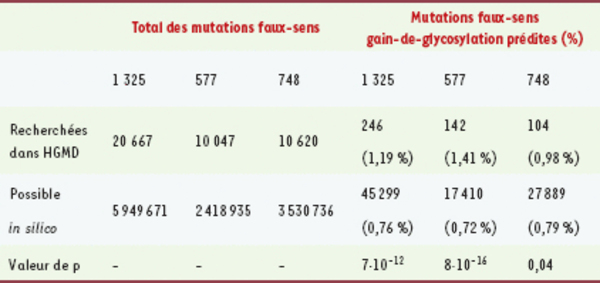
Tableau I.
Un total de 20 667 mutations faux-sens pathogènes comprises dans 1 325 gènes ont été analysées dans la HGMD (Human Gene Mutation Database) (mise à jour de mai 2004). Parmi ces gènes, 577 codent pour des protéines qui sont prédites pour entrer dans le réticulum endoplasmique et ainsi être potentiellement glycosylées (fondé sur l’identification, in silico, des peptides signaux ou d’ancrages) avec une probabilité combinée supérieure à 0,75. Les 748 gènes restants sont ceux qui ne sont pas prédits pour entrer dans le réticulum endoplasmique. De ces groupes de 1 325, 577 et 748 gènes, les mutations dans 158, 77 et 81 gènes, respectivement, causent potentiellement des gains de glycosylation. Pour chacun de ces trois groupes de gènes, le nombre de mutations faux-sens pathogènes documentées, entraînant un gain de glycosylation prédit a été comparé avec le nombre total de mutations faux-sens possibles (créées in silico) pouvant créer un site consensus de glycosylation. Une statistique de χ2 a ensuite été utilisée pour comparer les proportions observées avec celles attendues (valeurs de p indiquées) (adapté de [16]).
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.