Figure 3.
Télécharger l'image originale
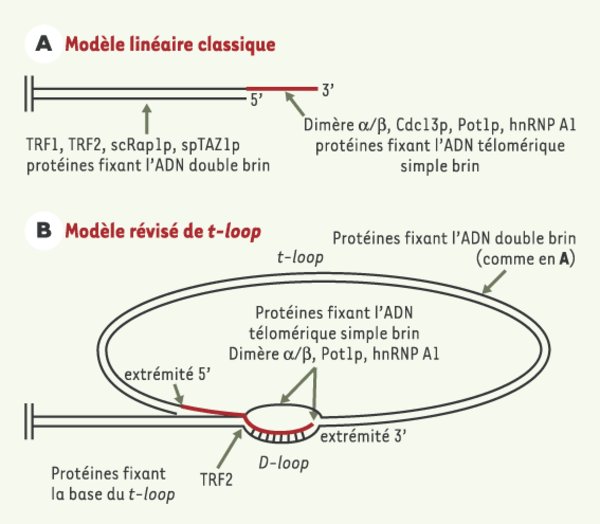
Modèles de structure des télomères. A. Modèle classique d’une extrémité chromosomique avec une courte extension en 3’ (en rouge) et un complexe protéique protégeant l’ADN chromosomique (modèle linéaire). B. Les télomères des mammifères se terminent par de larges boucles, les t-loops (telomere loops). Le double brin télomérique se replie sur lui-même, l’extension 3’ simple brin s’insère dans la portion double brin et engendre la D-loop (displacement loop). La protéine TRF2 se fixe à la base du D-loop in vitro. Les t-loops, en association avec un complexe protéique terminal, pourraient permettre de différencier les extrémités des chromosomes des cassures double brin dans l’ADN, et éviter ainsi la détection des télomères par la machinerie de réparation des cassures d’ADN. Les régions simple brin du t-loop et les extensions en 3’ sont indiquées en rouge. TRF1, 2 : telomeric repeat binding factor 1, 2 ; scRap1p : protéine télomérique RAP1 de Saccharomyces cerevisiae ; Cdc13p : telomere binding protein Cdc13p ; Pot1p : protection of telomeres 1 ; hnRNPA1 : heterogeneous ribonucleoprotein A1.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




