| Numéro |
Med Sci (Paris)
Volume 33, Numéro 2, Février 2017
|
|
|---|---|---|
| Page(s) | 151 - 158 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/20173302010 | |
| Publié en ligne | 27 février 2017 | |
Séquences provenant d’éléments génétiques mobiles, face cachée du génome humain
DNA sequences from mobile genetic elements, a hidden half of the human genome
1
GeNeuro Innovation, Bioparc Laënnec, 60, avenue Rockefeller, 69008
Lyon, France
2
GeNeuro, 18, chemin des Aulx, 1228 Plan-Les-Ouates, Genève, Suisse
3
Université Lyon-1, Faculté de Médecine Laënnec, 69008
Lyon, France
*
julie.medina@geneuro.com
**
hp@geneuro.com
Les données actuelles estiment que les éléments génétiques mobiles représentent plus de la moitié du génome humain. La littérature est en perpétuelle évolution à ce sujet compte tenu de l’évolution du séquençage et des techniques d’analyse. Cet article a pour objectif de donner des bases sur ce sujet et d’en montrer la complexité au vu des différentes classifications et appellations retrouvées dans les articles scientifiques. La catégorie des rétrotransposons et notamment celle des rétrovirus endogènes sera plus particulièrement développée afin d’appréhender, dans une seconde revue, leur activation et leur implication dans des fonctions physiologiques et/ou des mécanismes pathologiques, notamment ceux associés au développement de maladies telles que la sclérose en plaques (SEP) ou la sclérose latérale amyotrophique (SLA).
Abstract
Current data estimate that mobile genetic elements represent more than one-half of the human genome. The literature is constantly updating data following the evolution of sequencing techniques and of algorithms for genome analyses. This review aims to provide an overview of the topic showing the complexity given by the various designations and classifications found in scientific papers. A particular focus is made on retrotransposons, including Endogenous RetroViruses (ERV), to introduce a second article focusing on their activation and their involvement in physiological functions and/or pathological mechanisms associated with diseases like multiple sclerosis (MS) or amyotrophic lateral sclerosis (ALS).
© 2017 médecine/sciences – Inserm
Le génome des eucaryotes est constitué d’un ensemble de séquences d’ADN dont certaines ont une origine particulière, que l’on nomme « éléments transposables » ou encore « éléments génétique mobiles ». Ces éléments ont été découverts dans les années 1980 par Barbara McClintock, dont le travail de génétique sur les grains de maïs a été récompensé par l’attribution du Prix Nobel [1]. Ils sont présents en proportion variable selon les espèces. Initialement on estimait qu’ils représentaient environ 45 % du génome humain. Un article publié en 2016 parle désormais de 2/3 de notre génome [2]. Leur détection est particulièrement complexe, ce qui explique que leur proportion au sein des génomes a été largement sous-estimée et que les données évoluent au fur et à mesure des années, avec l’amélioration des techniques et des logiciels permettant de les mettre en évidence.
On distingue deux types d’éléments transposables : les éléments qui peuvent se transposer via un intermédiaire ADN et un mécanisme de « coupé-collé » (ce sont les transposons ou éléments de classe II, ils représentent environ 3 % du génome humain) et ceux qui utilisent un ARN et un mécanisme de « copié-collé » (ce sont les rétrotransposons ou éléments de classe I qui représentent environ 42 % du génome humain) [3]. Ces pourcentages évoluent avec le séquençage de nouvelles régions du génome, auparavant inaccessibles comme notamment les télomères.
Parmi les rétrotransposons (Tableau I), il existe des rétrovirus endogènes (ERV pour endogenous retrovirus) pour lesquels la littérature actuelle présente beaucoup de données incomplètes et des classifications ou nomenclatures différentes, ce qui génère certaines ambiguïtés. Ces incohérences sont liées à des analyses fondées sur des gènes différents ou à des données divergentes qui ont été acquises plus récemment.
Les rétrotransposons du génome humain. HERV : human endogenous retrovirus ; MaLR : mammalian apparent LTR retrotransposon ; LINE : long interspersed elements ; SINE : short interspersed elements ; SVA : SINE-VNTR (variable-number-of-tandem-repeats)-Alus ; séquences Alu : séquences de type SINE caractérisées par un site de reconnaissance unique pour l’enzyme de restriction AluI.
Cette revue abordera certains aspects encore méconnus et présentera les classifications ou nomenclatures existantes des ERV humains (HERV pour human-ERV) parmi les séquences rétrovirales. D’ailleurs, l’utilisation du terme « HERV » indique que l’on fait référence aux séquences des ERV retrouvées dans le génome humain, au sein d’une famille endogène phylogéniquement commune à plusieurs genres et espèces, ce qui est le cas pour la famille HERV de type W (HERV-W) ou ERV-W [4].
Les rétrotransposons
Parmi les rétrotransposons, on distingue ceux qui ont des longues répétitions terminales (LTR) à leurs extrémités et ceux qui en sont dépourvus (environ 80 % des rétrotransposons) [4]. Les LTR sont en général dupliqués aux deux extrémités des séquences codantes et comprennent des régions promotrices et régulatrices de la transcription. Les LTR sont divisés en 3 régions : U3 (pour « unique » en 3’ de l’ARN), R (pour « répétée » aux deux extrémités de l’ARN) et U5 (pour « unique » en 5’ de l’ARN). L’ARN transcrit débutera et se terminera à la région R. Ces rétroéléments représentent au moins 43 % du génome humain [4].
Rétroélements sans LTR
Les séquences LINE (long interspersed elements) codent 2 protéines : ORF1p (pour open reading frame protein 1, une protéine de liaison à l’ARN) et ORF2p (pour open reading frame protein 2, une protéine ayant des activités de transcriptase inverse et endonucléasique). Ces deux protéines se lient à l’ARN pour former un complexe ribonucléoprotéique qui transite dans le noyau où l’endonucléase permet le clivage de l’ADN cellulaire ainsi que la création d’amorces pour la transcription inverse de l’ARN en ADN qui peut ensuite s’intégrer dans les chromosomes.
Les séquences SINE (short interspersed elements) correspondent à un groupe d’éléments sans LTR qui n’ont pas la capacité de coder des protéines mais qui détournent la machinerie des éléments LINE pour compléter leur transposition. La plupart de ces éléments SINE sont des éléments Alu (1,1 millions de sites d’insertion) ainsi appelés parce que la plupart d’entre eux contiennent un site de reconnaissance unique pour l’enzyme de restriction AluI.
Les SVA (SINE-VNTR [variable-number-of-tandem-repeats]-Alus) sont des éléments SINE qui présentent des répétitions d’hexamères, une séquence Alu, un nombre variable de répétitions en tandem ainsi qu’une région d’analogie à un rétrovirus endogène (HERV-K) appelée SINE-R [5]. Les SVA sont des rétroéléments qui ont été identifiés récemment. Ils ont été mis en cause dans plusieurs maladies génétiques humaines [5].
Rétroéléments à LTR
Les rétrovirus endogènes (endogenous retrovirus ou ERV) sont présents sous forme de séquences intégrées dans l’ADN chromosomique à la suite d’insertion dans la lignée germinale, contrairement aux rétrovirus exogènes qui eux sont classiquement des agents infectieux environnementaux. En effet, les génomes rétroviraux sont des ARN qui peuvent être rétro-transcrits en ADN par une transcriptase inverse que code leur gène pol, puis intégrés dans l’ADN des cellules à l’aide d’une autre enzyme rétrovirale appelée intégrase. On parle alors de génome proviral ou de provirus. Les rétrovirus endogènes sont rarement infectieux, mais certains ERV infectieux ont été identifiés chez des mammifères [6, 7]. Comme illustré dans la Figure 1 , ils correspondent pour la plupart à des « vestiges génétiques » d’anciens virus exogènes dont des séquences ont colonisé notre génome et ont été transmises verticalement, parfois avec de nouvelles rétrotranspositions ou des réarrangements somatiques, voire des réinfections fréquentes de la lignée germinale, par un virus exogène de l’environnement au cours de certaines périodes de l’évolution [8, 9]. Cela aboutit à ce que toutes les cellules d’un nouvel organisme possèdent un certain nombre de copies du génome rétroviral dans leur ADN. On parle « d’endogénisation » [10], contrairement aux rétrovirus infectieux acquis dans l’environnement (comme le virus de l’immunodéficience humaine ou VIH) qui ne peuvent intégrer un génome proviral que dans l’ADN des cellules qu’ils sont capables d’infecter et non de l’ensemble des cellules de l’organisme. Encore aujourd’hui, il existe des rétrovirus en cours d’endogénisation : c’est le cas d’un rétrovirus présent chez le koala (KoRV) [11] provenant de la souche exogène proche d’un rétrovirus de leucémie du gibbon (gibbon ape leukemia virus, GalV) [11–13]. Ce phénomène est même envisagé pour le VIH chez l’homme [14].
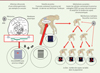 |
Figure 1. Illustration du mode d’acquisition des ERV (endogenous retrovirus). LTR : longues répétitions terminales. |
Selon les données actuelles, les plus anciens HERV seraient des familles HERV-S/L apparues il y a 150 millions d’années, et HERV-F/H apparues il y a 70 millions d’années, intégrées avant la séparation entre les singes et les lémuriens, tandis que la famille HERV-K représente la famille la plus récemment intégrée, il y a moins de 6 millions d’années [15].
Depuis leur endogénisation, la plupart des HERV ont été progressivement altérés par l’accumulation de mutations, d’insertions ou de délétions, donnant les pseudo-éléments. Il existe aussi un nombre très important de séquences « LTR solitaires » ou « solo LTR » résultant de recombinaisons homologues qui ont excisé les gènes structuraux internes. Par exemple, le génome humain contient environ 50 copies de la famille HERV9 et entre 3 000 et 4 000 LTR apparentés à HERV9 [15]. On estime en fait que 85 % des séquences HERV correspondent à des LTR solitaires [15], mais il semble aussi que les logiciels d’assemblage des séquences puissent eux-mêmes aligner des séquences flanquantes répétées, comme les LTR, puis exclure les séquences internes qui sont mises à part et non assemblées, voire réassemblées avec celles d’autres sites d’insertion [16, 17].
Les HERV sont généralement rendus silencieux par contrôle épigénétique comme la méthylation de leurs promoteurs et de leurs gènes. Cependant, de rares séquences rétrovirales endogènes ont gardé la capacité de coder des protéines fonctionnelles. La transcription de ces séquences est possible quand ces gènes rétroviraux sont dé-réprimés et activés spécifiquement dans certains tissus et phénotypes cellulaires [18], mais cela aboutit rarement à une traduction en protéines. Certaines protéines issues de l’expression anormale de séquences rétrovirales endogènes humaines sont néanmoins associées à des maladies complexes, notamment des maladies neurodégénératives comme la sclérose latérale amyotrophique (SLA) via la protéine d’enveloppe de HERV-K, ainsi que des maladies auto-immunes comme la sclérose en plaques (SEP) ou des syndromes neuropsychiatriques associant une immuno-inflammation comme la schizophrénie ou la maladie bipolaire via la protéine d’enveloppe de HERV-W [19]. La famille HERV-E est également associée à différentes maladies avec une production de protéines endogènes codées par des séquences qui sont normalement sous contrôle épigénétique [20].
On estime actuellement que 8 % des séquences du génome humain sont des rétrotransposons à LTR, contre 3 % pour les séquences codant l’ensemble des protéines humaines [21]. Généralement, les articles simplifient en disant que ces 8 % du génome correspondent à des HERV [22, 23].
Certaines publications présentent au moins 31 familles d’HERV [21], d’autres une cinquantaine [24] et certaines vont jusqu’à 200 [15]. Actuellement, le nombre de copies d’HERV de différentes familles dans le génome humain n’est pas homogène dans la littérature. Cela peut souvent s’expliquer par la prise en compte, ou non, d’entités particulières comme les LTR solitaires (Tableau II).
Nombre de copies des familles HERV-W et HERV-K (HML-2) dans le génome humain. HERV-W : human endogenous retrovirus type W (les éléments de cette famille endogène ont un site d’amorçage de la transcriptase inverse dont la séquence nucléotidique est complémentaire de l’anticodon de l’ARNt tryptophane dont le symbole est « W ») ; HERV-K : human endogenous retrovirus type K (les éléments de cette famille endogène ont un site d’amorçage de la transcriptase inverse dont la séquence nucléotidique est complémentaire de l’anticodon de l’ARNt lysine dont le symbole est « K ») ; HML2 : human mouse mammary tumor virus like-2 (élément prototype de la famille HERV-K) ; LTR : longues répétitions terminales ; MSRV : multiple sclerosis-associated retrovirus.
• Les rétrovirus endogènes dits « fixés »
Ils représentent des insertions chromosomiques stables dans l’ensemble de la population. Ces HERV ont un nombre variable de gènes structuraux (gag, pro, pol et env) et de LTR (normalement deux, aux extrémités 5’ et 3’) résultant de réarrangements somatiques divers et comprenant de fréquentes mutations interdisant leur transcription ou leur traduction en protéines quand ils sont transcrits sous forme d’ARN défectifs. Néanmoins, certaines copies complètes ou incomplètes ont gardé les gènes rétroviraux avec des cadres de lecture ouverts et peuvent exprimer des protéines, voire des virions, y compris dans le génome humain [25]. Même partiellement codantes, des copies peuvent aussi se complémenter : ce phénomène aussi appelé « transcomplémentation » peut ainsi générer des virions complets à partir des ARN exprimés par différents provirus incomplets.
• Les rétrovirus endogènes dits « non fixés »
De telles copies endogènes, déjà bien connues dans d’autres espèces, ont été récemment découvertes chez l’homme avec l’étude de la famille HERV-K dont le nombre de copies s’avère varier au sein de la population humaine [9]. En plus des copies anciennement fixées, on distingue désormais les copies plus récemment intégrées dont la présence et le nombre sont variables d’un individu à un autre ; elles sont plus susceptibles d’exprimer des ARN, voire des protéines, dans des contextes particuliers. Ainsi, dans son matériel génétique, chaque individu contient des insertions rétrovirales endogènes distribuées inégalement dans la population. Dans la population humaine, ces copies non-fixées ont été détectées dans un pourcentage variable d’individus [9].
• Les MaLR (mammalian apparent LTR retrotransposon)
Ce sont des rétrotransposons à LTR qui présentent des délétions internes, notamment au niveau des gènes gag et pol [4, 15].
Les structures génomiques de ces différents éléments sont représentées dans la Figure 2 .
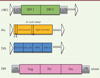 |
Figure 2. Structure génomique des rétroélements. ORF : open reading frame (cadre de lecture ouvert) ; LTR : long terminal repeat (longue répétition flanquante) ; VNTR : variable number of tandem repeats (nombre variable de répétitions en tandem) ; SINE-R : short interspersed elements (éléments courts intercalés) ; UTR : untranslated region (région non codante) ; Gag : antigène de groupe (matrice + capside + nucléocapside) ; Pol : gène codant pour les enzyme rétrovirales (transcriptase inverse + Rnase H + protéase + intégrase ) ; Env : enveloppe ; Alu-like : séquence de type Alu (contenant un site de restriction AluI) ; ERV : endogenous retrovirus ; LINE1 : long interspersed repetitive element 1 ; SVA : SINE-VNTR-Alus ; UTR : untranslated regions (adapté de [40]). |
Classifications des rétrovirus
Les séquences rétrovirales sont répertoriées selon plusieurs nomenclatures qui se fondent sur des critères de classement différents allant des pathologies qui leur sont associées à l’analyse en microscopie électronique des particules rétrovirales produites par les différents rétrovirus et aux homologies de leurs séquences nucléotidiques. Plusieurs articles font ouvertement part d’un besoin d’uniformité dans la nomenclature et le classement des rétrovirus endogènes [22, 24]. Notamment, avec les classements fondés sur des alignements nucléotidiques, les homologies entre les familles rétrovirales peuvent varier selon les gènes qui sont alignés. Nous allons dans cette partie de la revue décrire les classifications qui ont été proposées jusqu’à présent.
Classification en fonction de critères pathologiques
Historiquement les rétrovirus étaient considérés selon trois catégories liées à leur pathogénicité :
-
les Oncovirinae causant des tumeurs et des leucémies chez les volailles et les mammifères, comme le virus aviaire du sarcome de Rous et le virus de la tumeur mammaire murine (MMTV) ;
-
les Lentivirinae causant des maladies à évolution lente comme le Visna du mouton (atteinte neurologique progressive) ;
-
les Spumavirinae causant des effets cytopathiques avec des images de « mousse » produite par les cellules infectées, sans pathologie clinique avérée [26].
Classification en fonction de leur morphologie
Une classification des rétrovirus a été établie en fonction du mode de formation et de la morphologie des particules virales, d’après des observations en microscopie électronique : le type A avec des virions strictement intracellulaires dépourvus d’enveloppe ; les types B, C ou D pour des virus enveloppés dont les particules sont extracellulaires mais dont les conditions d’assemblage et de bourgeonnement dans les cellules infectées, la taille et la forme varient [27]. À cela a été ajouté un type « lentivirus » à la suite des études réalisées sur le VIH montrant des caractéristiques qui ne pouvaient s’accorder avec les critères des catégories précédentes mais qui se sont révélées communes avec les autres Lentivirinae.
Classification en fonction de leur génome
Quand les analyses de séquences nucléotidiques des génomes sont devenues disponibles, la classification des ERV s’est alors fondée sur les homologies génétiques de leurs séquences provirales chromosomiques avec celles des rétrovirus animaux et humains. Ainsi, l’ICTV (international committee on taxonomy of viruses) 1 propose une classification qui est fondée sur ces séquences nucléotidiques, divisant la famille des Retroviridae en 2 sous-familles qui regroupent 7 genres :
-
Les Orthovirinae qui contiennent 6 genres : alpha, bêta, gamma, delta, epsilon, et lenti-rétrovirus.
-
Les Spumavirinae qui correspondent au genre Spumavirus.
D’autres classifications se réfèrent aux alignements de séquences des gènes pol et notamment de la transcriptase inverse. On distingue ainsi les rétrovirus de classe I, regroupant les gamma- et epsilon-rétrovirus ; ceux de classe II regroupant les lentivirus et les alpha, bêta et delta-rétrovirus ; ceux de classe III correspondant aux spuma-rétrovirus [28] ; la classe IV rassemble les autres éléments éventuels [29].
En se fondant sur la composition du génome viral, il est parfois proposé de regrouper les rétrovirus à génome simple (alpha, bêta, gamma et epsilon) et ceux qui ont un génome complexe (delta, lenti- et spuma-rétrovirus) [30]. Les génomes simples contiennent des gènes codant les protéines structurales et les polyprotéines Gag, Pro, Pol et Env, alors que les génomes complexes produisent des protéines additionnelles et des ARN avec des fonctions régulant notamment la virulence.
Cependant, cette classification s’avère inexacte notamment pour l’ensemble du génome de certaines familles d’ERV, comme celle du baboon endogenous virus (BaEV) et celle d’HERV-W. En effet, HERV-W est souvent présenté comme appartenant aux gamma-rétrovirus quand les alignements se réfèrent au seul gène pol. Or, HERV-W a un génome hybride, avec des origines phylogéniques différentes pour les gènes gag-pol d’une part et pour le gène env d’autre part. On retrouve la même phylogénie hybride dans le BaEV dont il est le plus proche. Ce génome est vraisemblablement issu d’une recombinaison entre un gamma-rétrovirus proche du virus de la leucémie murine (MLV) pour les gènes gag, pro et pol et un bêta-rétrovirus proche du virus simien Mason-Pfizer (MPMV) pour le gène env. Comme présenté dans la Figure 3 , les alignements de séquences de l’ORF (open reading frame) gag-pol (Figure 3A) classent les familles HERV-W et BaEV dans les gamma-rétrovirus, les rétrovirus de type C et les Oncovirinae, alors que les alignements de séquences de l’ORF env (Figure 3B) les rangent dans les bêta-rétrovirus, les rétrovirus de type D, et encore les Oncovirinae.
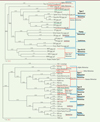 |
Figure 3. Alignements des séquences gag-pol (A) et env (B) HERV-W avec les autres Retroviridae. L’alignement par homologie de séquence (logiciel « Mac Vector », méthode « neighbor joining ; best tree ») utilise la séquence d’enveloppe d’un virus d’un autre genre (virus herpès simplex de type 1, HSV-1) pour produire un repère « éloigné » requis pour comparer ces séquences rétrovirales. Les différentes classifications pour les génomes rétroviraux discutées dans le texte sont indiquées dans les différents groupes encadrés par les parenthèses en rouge (classification selon les homologies de séquence du gène pol) et en bleu (classification selon la morphologie et/ou selon le type de pathologie associée). Les groupes d’alignement pour les lentivirus et les foamy virus ou spumavirus (virus spumeux) coïncident et sont dans le même cadre bleu. Les génomes hybrides, qui se retrouvent « classés » différemment selon les alignements des gènes gag-pol ou env (voir texte), sont soulignés en vert. HSV-1 : virus Herpes simplex de type 1 ; ALV : virus de la leucose aviaire ; RSV : virus du sarcome de Rous ; HTLV : virus T-lymphotropique humain ; GALV : gibbon ape leukemia virus (leucémie du singe gibbon) ; Koala retrovirus : rétrovirus du koala ; SFFV : spleen focus-forming virus (virus formant des foyers dans la rate) ; MoMLV : virus Moloney de la leucémie murine (variant du MLV) ; MLV : murine leukemia virus (leucémie murine) ; Rauscher MLV : virus Rauscher de la leucémie murine (variant du MLV) ; HERV-W : human endogenous retrovirus type W (rétrovirus endogène humain de type W) ; BaEV : baboon endogenous virus (rétrovirus endogène du babouin) ; MPMV : Mason-Pfizer monkey virus (virus Mason-Pfizer du singe) ; HERV-K113 : human endogenous retrovirus type K, K113 copy (rétrovirus endogène humain de type K, copie K113) ; JSRV : Jaagsiekte retrovirus (virus de l’adénomatose pulmonaire du mouton) ; MMTV : mouse mammary tumor virus (tumeur mammaire murine) ; HIV : human immunodeficiency virus (virus de l’immunodéficience humaine) ; SIV : simian immunodeficiency virus (virus de l’immunodéficience simienne) ; CAEV : caprine arthritis-encephalitis virus (virus de l’arthrite et de l’encéphalite de la chèvre) ; MVV : Maedi-Visna virus (virus du Maëdi-Visna, causant des atteintes pulmonaires, nerveuses, articulaires ou mammaires chez le mouton) ; FFV : feline foamy virus (virus « spumeux » félin) ; BFV : bovine foamy virus (virus « spumeux » bovin) ; SFV : simian foamy virus (virus « spumeux » simien) ; PERV : porcine endogenous retrovirus (rétrovirus endogène porcin). |
Cet exemple montre les limites des classifications actuelles qui se focalisent sur des homologies fondées sur un seul gène. Ainsi, les rétrovirus ayant généré des recombinaisons inter-familles qui se sont maintenues au cours de l’évolution se retrouvent classés de façon inappropriée avec des rétrovirus qui ont, eux, une phylogénie homogène. Ces génomes hybrides devraient constituer une catégorie à part et les études d’homologie devraient prendre en compte tous les gènes afin de prévenir l’ignorance de ces particularités dont la traduction biologique peut être très divergente des autres rétrovirus avec lesquels ils sont classés actuellement.
Classification selon la région PBS (primer binding site)
Une classification plus spécifique des HERV est fondée sur la séquence du PBS (primer binding site) qui est présent en aval du LTR5’. Ce site consiste en un anticodon reconnu par un ARNt (ARN de transfert), spécifique d’acide aminé, qui sert d’amorce à la transcriptase inverse pour la réplication du génome rétroviral. Par exemple, la famille HERV, dont les membres présentent un PBS avec une séquence homologue à un anticodon pour le site spécifique de l’ARNt de la lysine (K), est appelée HERV-K. Cette classification est aussi considérée comme inadéquate puisque des provirus appartenant au même groupe phylogénique peuvent avoir des régions PBS distinctes [24].
Conclusion
Afin de clarifier la nomenclature et la classification des HERV qui restent actuellement imparfaites, Mayer et al. ont proposé une nouvelle nomenclature. Elle prendrait la forme :
ERV + symbole alphabétique + numéro de membre unique.
Ainsi : ERVK-6 pour rétrovirus endogène du groupe K, membre 6 [31].
Cependant, comme les exemples des familles BaEV et HERV-W le démontrent, les catégories définies sur une base ou une autre ne peuvent être fiables que si elles représentent l’ensemble des gènes de ces éléments rétroviraux. Il est néanmoins intéressant de constater que, parmi les différents systèmes de classification que nous avons exposés, la seule catégorie « stable » pour ces éléments est celle qui se rapporte aux Oncovirinae, Lentivirinae et Spumavirinae.
La difficulté à classifier et à nommer les ERV reflète donc leur longue histoire de transmission inter-espèces, d’amplification intranucléaire ou par réinfections multiples de la lignée germinale, de même que les influences environnementales ayant agi sur leur sélection au cours de l’évolution. Il est aujourd’hui nécessaire de s’accorder sur des appellations et sur une classification globale et fiable [32]. Cependant, de nombreux aspects encore méconnus et des données critiques non encore identifiées pourraient rendre cet objectif irréalisable à ce jour. La poursuite de l’étude de ces éléments du génome humain et des autres espèces est donc indispensable pour atteindre un niveau de connaissance suffisant à la définition de caractéristiques stables pour une classification selon des critères qui ne deviendront pas rapidement caduques.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article, mais déclarent avoir des liens durables avec l’entreprise GeNeuro-Innovation, 60, avenue Rockefeller, 69008 Lyon, France.
Références
- Ravindran S. Barbara McClintock and the discovery of jumping genes. Proc Natl Acad Sci USA 2012 ; 109 : 20198–20199. [CrossRef] [Google Scholar]
- Hancks DC, Kazazian HH Roles for retrotransposon insertions in human disease. Mob DNA 2016 ; 7 : 9. [Google Scholar]
- Pace JK, Feschotte C The evolutionary history of human DNA transposons : evidence for intense activity in the primate lineage. Genome Res 2007 ; 17 : 422–432. [CrossRef] [PubMed] [Google Scholar]
- Kassiotis G, Stoye JP Immune responses to endogenous retroelements : taking the bad with the good. Nat Rev Immunol 2016 ; 16 : 207–219. [CrossRef] [PubMed] [Google Scholar]
- Ostertag EM, Goodier JL, Zhang Y, et al. SVA Elements are nonautonomous retrotransposons that cause disease in humans. Am J Hum Genet 2003 ; 73 : 1444–1451. [CrossRef] [PubMed] [Google Scholar]
- Xu L, Tay CH, Huber BT, et al. Cloning of an infectious milk-borne mouse mammary tumor virus (MMTV) DNA from a mammary tumor that developed in an endogenous MMTV-free wild mouse. Virology 2000 ; 273 : 325–332. [CrossRef] [PubMed] [Google Scholar]
- Black SG, Arnaud F, Burghardt RC, et al. Viral particles of endogenous betaretroviruses are released in the sheep uterus and infect the conceptus trophectoderm in a transspecies embryo transfer model. J Virol 2010 ; 84 : 9078–9085. [CrossRef] [PubMed] [Google Scholar]
- Belshaw R, Katzourakis A, Paces J, et al. High copy number in human endogenous retrovirus families is associated with copying mechanisms in addition to reinfection. Mol Biol Evol 2005 ; 22 : 814–817. [CrossRef] [PubMed] [Google Scholar]
- Marchi E, Kanapin A, Magiorkinis G, et al. Unfixed endogenous retroviral insertions in the human population. J Virol 2014 ; 88 : 9529–9537. [CrossRef] [PubMed] [Google Scholar]
- Lemos de Matos A, de Sousa-Pereira P, Lissovsky AA, et al. Endogenization of mouse mammary tumor virus (MMTV)-like elements in genomes of pikas (Ochotona sp.). Virus Res 2015 ; 210 : 22–26. [CrossRef] [PubMed] [Google Scholar]
- Tarlinton RE, Meers J, Young PR Retroviral invasion of the koala genome. Nature 2006 ; 442 : 79–81. [CrossRef] [PubMed] [Google Scholar]
- Denner J, Young PR Koala retroviruses : characterization and impact on the life of koalas. Retrovirology 2013 ; 10 : 108. [CrossRef] [PubMed] [Google Scholar]
- Xu W, Eiden MV Koala retroviruses : evolution and disease dynamics. Annu Rev Virol 2015 ; 2 : 119–134. [CrossRef] [PubMed] [Google Scholar]
- Colson P, Ravaux I, Tamalet C, et al. HIV infection en route to endogenization : two cases. Clin Microbiol Infect 2014 ; 20 : 1280–1288. [Google Scholar]
- Katoh I, Kurata S Association of endogenous retroviruses and long terminal repeats with human disorders. Front Oncol 2013 ; 3 : 234. [Google Scholar]
- Chen G, Li R, Shi L, et al. Revealing the missing expressed genes beyond the human reference genome by RNA-Seq. BMC Genomics 2011 ; 12 : 590. [CrossRef] [PubMed] [Google Scholar]
- Chen G, Wang C, Shi L, et al. Comprehensively identifying and characterizing the missing gene sequences in human reference genome with integrated analytic approaches. Hum Genet 2013 ; 132 : 899–911. [CrossRef] [PubMed] [Google Scholar]
- Seifarth W, Frank O, Zeilfelder U, et al. Comprehensive analysis of human endogenous retrovirus transcriptional activity in human tissues with a retrovirus-specific microarray. J Virol 2005 ; 79 : 341–352. [CrossRef] [PubMed] [Google Scholar]
- Nath A, Küry P, Olival GS do, et al. First international workshop on human endogenous retroviruses and diseases, HERVs and disease 2015. Mob DNA 2015 ; 6 : 20. [Google Scholar]
- Le Dantec C, Vallet S, Brooks WH, et al. Human endogenous retrovirus group E and its involvement in diseases. Viruses 2015 ; 7 : 1238–1257. [CrossRef] [PubMed] [Google Scholar]
- Trela M, Nelson PN, Rylance PB The role of molecular mimicry and other factors in the association of human endogenous retroviruses and autoimmunity. APMIS 2016 ; 124 : 88–104. [Google Scholar]
- Vargiu L, Rodriguez-Tomé P, Sperber GO, et al. Classification and characterization of human endogenous retroviruses; mosaic forms are common. Retrovirology 2016 ; 13 : 7. [CrossRef] [PubMed] [Google Scholar]
- Li F, Karlsson H Expression and regulation of human endogenous retrovirus W elements. APMIS 2016 ; 124 : 52–66. [Google Scholar]
- Escalera-Zamudio M, Greenwood AD On the classification and evolution of endogenous retrovirus : human endogenous retroviruses may not be human after all. APMIS 2016 ; 124 : 44–51. [Google Scholar]
- Dube D, Contreras-Galindo R, He S, et al. Genomic flexibility of human endogenous retrovirus type K. J Virol 2014 ; 88 : 9673–9682. [CrossRef] [PubMed] [Google Scholar]
- Weiss RA The discovery of endogenous retroviruses. Retrovirology 2006 ; 3 : 3–67. [CrossRef] [PubMed] [Google Scholar]
- Cloyd MW. Human retroviruses. In: Baron S (ed). Medical microbiology, 4th ed, chapter 62. Galveston (TX) : University of Texas Medical Branch, 1996. [Google Scholar]
- Henzy JE, Gifford RJ, Johnson WE, et al. A novel recombinant retrovirus in the genomes of modern birds combines features of avian and mammalian retroviruses. J Virol 2014 ; 88 : 2398–2405. [CrossRef] [PubMed] [Google Scholar]
- Chong AY, Kojima KK, Jurka J, et al. Evolution and gene capture in ancient endogenous retroviruses - insights from the crocodilian genomes. Retrovirology 2014 ; 11 : 71. [CrossRef] [PubMed] [Google Scholar]
- Jern P, Sperber GO, Blomberg J Use of endogenous retroviral sequences (ERVs) and structural markers for retroviral phylogenetic inference and taxonomy. Retrovirology 2005 ; 2 : 50. [CrossRef] [PubMed] [Google Scholar]
- Mayer J, Blomberg J, Seal RL A revised nomenclature for transcribed human endogenous retroviral loci. Mob DNA 2011 ; 2 : 7. [Google Scholar]
- Blomberg J, Benachenhou F, Blikstad V, et al. Classification and nomenclature of endogenous retroviral sequences (ERVs): problems and recommendations. Gene 2009 ; 448 : 115–123. [Google Scholar]
- Pavlícek A, Paces J, Elleder D, et al. Processed pseudogenes of human endogenous retroviruses generated by LINEs : their integration, stability, and distribution. Genome Res 2002 ; 12 : 391–399. [CrossRef] [PubMed] [Google Scholar]
- Li F, Nellåker C, Yolken RH, et al. A systematic evaluation of expression of HERV-W elements; influence of genomic context, viral structure and orientation. BMC Genomics 2011 ; 12 : 22. [CrossRef] [PubMed] [Google Scholar]
- Subramanian RP, Wildschutte JH, Russo C, et al. Identification, characterization, and comparative genomic distribution of the HERV-K (HML-2) group of human endogenous retroviruses. Retrovirology 2011 ; 8 : 90. [CrossRef] [PubMed] [Google Scholar]
- Macfarlane C, Simmonds P Allelic variation of HERV-K(HML-2) endogenous retroviral elements in human populations. J Mol Evol 2004 ; 59 : 642–656. [Google Scholar]
- Wildschutte JH, Ram D, Subramanian R, et al. The distribution of insertionally polymorphic endogenous retroviruses in breast cancer patients and cancer-free controls. Retrovirology 2014 ; 11 : 62. [CrossRef] [PubMed] [Google Scholar]
- Garcia-Montojo M, Dominguez-Mozo M, Arias-Leal A, et al. The DNA copy number of human endogenous retrovirus-W (MSRV-type) is increased in multiple sclerosis patients and is influenced by gender and disease severity. PLoS One 2013 ; 8 : e53623. [Google Scholar]
- Wildschutte JH, Williams ZH, Montesion M, et al. Discovery of unfixed endogenous retrovirus insertions in diverse human populations. Proc Natl Acad Sci USA 2016 ; 113 : E2326–E2334. [CrossRef] [Google Scholar]
- Xing J, Witherspoon DJ, Jorde LB Mobile element biology : new possibilities with high-throughput sequencing. Trends Genet 2013 ; 29 : 280–289. [Google Scholar]
- Jern P, Sperber GO, Blomberg J Divergent patterns of recent retroviral integrations in the human and chimpanzee genomes: probable transmissions between other primates and chimpanzees. J Virol 2006 ; 80 : 1367–1375. [CrossRef] [PubMed] [Google Scholar]
Liste des tableaux
Les rétrotransposons du génome humain. HERV : human endogenous retrovirus ; MaLR : mammalian apparent LTR retrotransposon ; LINE : long interspersed elements ; SINE : short interspersed elements ; SVA : SINE-VNTR (variable-number-of-tandem-repeats)-Alus ; séquences Alu : séquences de type SINE caractérisées par un site de reconnaissance unique pour l’enzyme de restriction AluI.
Nombre de copies des familles HERV-W et HERV-K (HML-2) dans le génome humain. HERV-W : human endogenous retrovirus type W (les éléments de cette famille endogène ont un site d’amorçage de la transcriptase inverse dont la séquence nucléotidique est complémentaire de l’anticodon de l’ARNt tryptophane dont le symbole est « W ») ; HERV-K : human endogenous retrovirus type K (les éléments de cette famille endogène ont un site d’amorçage de la transcriptase inverse dont la séquence nucléotidique est complémentaire de l’anticodon de l’ARNt lysine dont le symbole est « K ») ; HML2 : human mouse mammary tumor virus like-2 (élément prototype de la famille HERV-K) ; LTR : longues répétitions terminales ; MSRV : multiple sclerosis-associated retrovirus.
Liste des figures
 |
Figure 1. Illustration du mode d’acquisition des ERV (endogenous retrovirus). LTR : longues répétitions terminales. |
| Dans le texte | |
 |
Figure 2. Structure génomique des rétroélements. ORF : open reading frame (cadre de lecture ouvert) ; LTR : long terminal repeat (longue répétition flanquante) ; VNTR : variable number of tandem repeats (nombre variable de répétitions en tandem) ; SINE-R : short interspersed elements (éléments courts intercalés) ; UTR : untranslated region (région non codante) ; Gag : antigène de groupe (matrice + capside + nucléocapside) ; Pol : gène codant pour les enzyme rétrovirales (transcriptase inverse + Rnase H + protéase + intégrase ) ; Env : enveloppe ; Alu-like : séquence de type Alu (contenant un site de restriction AluI) ; ERV : endogenous retrovirus ; LINE1 : long interspersed repetitive element 1 ; SVA : SINE-VNTR-Alus ; UTR : untranslated regions (adapté de [40]). |
| Dans le texte | |
 |
Figure 3. Alignements des séquences gag-pol (A) et env (B) HERV-W avec les autres Retroviridae. L’alignement par homologie de séquence (logiciel « Mac Vector », méthode « neighbor joining ; best tree ») utilise la séquence d’enveloppe d’un virus d’un autre genre (virus herpès simplex de type 1, HSV-1) pour produire un repère « éloigné » requis pour comparer ces séquences rétrovirales. Les différentes classifications pour les génomes rétroviraux discutées dans le texte sont indiquées dans les différents groupes encadrés par les parenthèses en rouge (classification selon les homologies de séquence du gène pol) et en bleu (classification selon la morphologie et/ou selon le type de pathologie associée). Les groupes d’alignement pour les lentivirus et les foamy virus ou spumavirus (virus spumeux) coïncident et sont dans le même cadre bleu. Les génomes hybrides, qui se retrouvent « classés » différemment selon les alignements des gènes gag-pol ou env (voir texte), sont soulignés en vert. HSV-1 : virus Herpes simplex de type 1 ; ALV : virus de la leucose aviaire ; RSV : virus du sarcome de Rous ; HTLV : virus T-lymphotropique humain ; GALV : gibbon ape leukemia virus (leucémie du singe gibbon) ; Koala retrovirus : rétrovirus du koala ; SFFV : spleen focus-forming virus (virus formant des foyers dans la rate) ; MoMLV : virus Moloney de la leucémie murine (variant du MLV) ; MLV : murine leukemia virus (leucémie murine) ; Rauscher MLV : virus Rauscher de la leucémie murine (variant du MLV) ; HERV-W : human endogenous retrovirus type W (rétrovirus endogène humain de type W) ; BaEV : baboon endogenous virus (rétrovirus endogène du babouin) ; MPMV : Mason-Pfizer monkey virus (virus Mason-Pfizer du singe) ; HERV-K113 : human endogenous retrovirus type K, K113 copy (rétrovirus endogène humain de type K, copie K113) ; JSRV : Jaagsiekte retrovirus (virus de l’adénomatose pulmonaire du mouton) ; MMTV : mouse mammary tumor virus (tumeur mammaire murine) ; HIV : human immunodeficiency virus (virus de l’immunodéficience humaine) ; SIV : simian immunodeficiency virus (virus de l’immunodéficience simienne) ; CAEV : caprine arthritis-encephalitis virus (virus de l’arthrite et de l’encéphalite de la chèvre) ; MVV : Maedi-Visna virus (virus du Maëdi-Visna, causant des atteintes pulmonaires, nerveuses, articulaires ou mammaires chez le mouton) ; FFV : feline foamy virus (virus « spumeux » félin) ; BFV : bovine foamy virus (virus « spumeux » bovin) ; SFV : simian foamy virus (virus « spumeux » simien) ; PERV : porcine endogenous retrovirus (rétrovirus endogène porcin). |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




