| Numéro |
Med Sci (Paris)
Volume 40, Numéro 10, Octobre 2024
Les microbes, l’Anthropocène et nous
|
|
|---|---|---|
| Page(s) | 757 - 765 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/2024121 | |
| Publié en ligne | 25 octobre 2024 | |
Le microbiome humain à l’épreuve de l’anthropocène
De la corrélation à la causalité et à l’intervention
The human microbiome proofed by the Anthropocene: from correlation to causality and intervention
1
Institut Pasteur et Collège de France Paris France
2
Université Paris-Saclay, INRAE, MetaGenoPolis, AgroParisTech, MICALIS Jouy-en-Josas France
*
philippe.sansonetti@pasteur.fr
Si les effets délétères des activités humaines sur la biodiversité du monde végétal et animal et sur le climat sont un fait acquis, leur impact sur la biodiversité microbienne doit être urgemment considéré, particulièrement sur le microbiome humain. La révolution métagénomique permet une large analyse et un suivi spatio-temporels jusqu’à présent inenvisageables. Une réduction de la richesse et de la diversité des microbiotes humains, en particulier intestinaux, est maintenant avérée, surtout dans les aires industrialisées de la planète. Utilisation inconsidérée des antibiotiques, changements drastiques des régimes alimentaires et éléments restant à déterminer de l’exposome environnemental sont le plus souvent incriminés. En découlent des situations de dysbioses caractérisées par une érosion du cœur d’espèces microbiennes communes à tous les individus et une prolifération de pathobiontes opportunistes, sans doute due à un affaiblissement de l’effet de barrière du microbiome. Le défi actuel est d’établir un lien de causalité entre ces dysbioses et des maladies en émergence épidémique, bien que non transmissibles, comme l’asthme, l’allergie, les maladies auto-immunes, l’obésité, le diabète et certains cancers. Modèles expérimentaux et études cliniques contrôlées prospectives et interventionnelles sont indispensables pour consolider cette causalité, d’autant que dans le déchiffrage des altérations de la symbiose homme-microbiome se profile un nouveau chapitre de la médecine : la « médecine microbienne »
Abstract
The deleterious effects of human activities on biodiversity in the vegetal and animal world, and on climate changes are now well-established facts. However, little is yet known on the impact of human activities on microbial diversity on the planet and more specifically on the human microbiota Large implementation of metagenomics allows exaustive microbial cataloguing with broad spatio-temporal resolution of human microbiota. A reduction in bacterial richness and diversity in the human microbiota, particularly in the intestinal tract, is now established and particularly obvious in the most industrialized regions of the planet. Massive, uncontrolled use of antibiotics, drastic changes in traditional food habits and some elements of the “global exposome” that remain to identify are usually considered as stressors accounting for this situation of “missing microbes”. As a consequence, a dysbiotic situation develops, a “dysbiosis” being characterized by the erosion of the central core of shared bacterial species across individuals and the development of opportunistic “pathobionts” in response to a weaker barrier capacity of these impoverished microbiota. The current challenge is to establish a causality link between the extension of these dysbiotic situations and the steady emergence of epidemic, non-communicable diseases such as asthma, allergy, obesity, diabetes, autoimmune diseases and some cancers. Experimental animal models combined with controlled, prospective clinical interventions are in demand to consolidate causality links, with the understanding that in the deciphering of the mechanisms of alteration of the human-microbiome symbiosis resides a novel exciting chapter of medicine: “microbial medicine”.
© 2024 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l’utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l’utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Vignette (© Philippe Sansonetti).
Le mode de vie moderne affecte la richesse et la diversité du microbiote1 intestinal humain. Cette perte de richesse et de diversité a pour possibles conséquences un accroissement du risque infectieux par affaiblissement de son effet de barrière contre les agents pathogènes et une participation à l’augmentation soutenue d’incidence de maladies « post-modernes, épidémiques, non transmissibles », selon la terminologie de l’Organisation mondiale de la santé (OMS)2. Cette inquiétante corrélation entre microbiologie et maladies chroniques – surtout maladies auto-immunes et allergiques – fut initialement proposée dans une revue fondatrice de Jean-François Bach [1]3, qui la rapporta à la réduction massive des maladies infectieuses au cours du xxe siècle sous l’action combinée de la mise en œuvre des mesures d’hygiène individuelle et collective – particulièrement celle des eaux et des aliments – et l’introduction des vaccins et des antibiotiques. On parla alors « d’hypothèse hygiéniste ». La réduction brutale d’une pression infectieuse multimillénaire était ainsi suspectée de rompre les règles d’engagement de la réponse immunitaire aux microbes et aux allergènes. Cependant, si le contrôle des maladies infectieuses demeure une hypothèse légitime, le concept s’est enrichi d’une hypothèse complémentaire, celle des « microbes manquants » (missing microbes) largement portée par Martin Blaser (Rutgers University, New Jersey, États-Unis) [2]. Cette hypothèse attribue un rôle majeur à l’appauvrissement du microbiome, dès la naissance, altérant ses multiples fonctions dans la maturation et le maintien de l’homéostasie de l’immunité, élargies à d’autres domaines, comme la nutrition, le métabolisme, le développement et la régulation de processus oncogènes. Ainsi, dans une vision du microbiome senseur, intégrateur et effecteur de stress causés par le mode de vie et l’environnement, les stress nouveaux, caractéristiques de la transition vers la modernité – l’anthropocène –, conduisent à une rupture de l’homéostasie de la symbiose microbiote-hôte, porteuse de nouvelles maladies [3].
Au-delà d’une perte de diversité de l’écosystème microbien humain liée au mésusage des antibiotiques, sur laquelle nous reviendrons, cette vision globale porte vers une réponse à l’exposome, tel que défini par l’épidémiologiste environnemental Rémy Slama, dans ses cours au Collège de France4.
Les altérations du microbiote moderne – les dysbioses – correspondent essentiellement à une perte de richesse et de diversité d’espèces appartenant au microbiote humain « ancestral » reflétant un « décalage évolutif » homme-microbiome potentiellement pathogène. La relation dysbiose-pathologie, la diversité de ses étiologies, de ses altérations, l’établissement des liens de causalité et son éventuelle réparation – le thème du « réensauvagement » (ou rewilding) – font l’objet de ce second article de la série thématique sur biodiversité microbienne, santé et maladie.
Sur quel référentiel de normalité définir une dysbiose ?
Même en ne considérant que les bactéries du microbiote intestinal, définir une dysbiose demeure complexe car, pour ce faire, il faudrait disposer d’un référentiel de « normalité ». Or, les grandes études métagénomiques du microbiote humain en cours montrent une extraordinaire hétérogénéité de composition, chaque microbiote étant qualitativement et quantitativement unique, au point que l’on a pu le comparer à des empreintes digitales…
Le microbiote intestinal d’un adulte sain comporte en moyenne 160 espèces bactériennes au sein desquelles une vingtaine seulement sont conservées, représentant un noyau essentiel aux fonctions physiologiques de bases du microbiome [4].
À une échelle populationnelle, on considère que le « panmétagénome » comporte plusieurs milliers d’espèces bactériennes. Cette hétérogénéité répond à la dynamique individuelle de construction du microbiote, l’origine géographique et le régime alimentaire des individus étant des facteurs essentiels [5]. De grandes configurations écologiques conservées ont pu être observées, assemblées sous le concept d’entérotypes, distinguant notamment les sujets ayant une alimentation de base riche en fibres [6]. Plus récemment, sur la base de larges échantillons longitudinaux, de nouvelles structures ont été proposées, combinant rapport entre certains genres dominants et fonctions métaboliques des microbiomes respectifs. C’est le concept d’entérobranches [7]. Cependant la structure de l’ensemble d’un microbiote individuel demeure multidimensionnelle et très dispersée, laissant de côté un nombre sans doute important d’espèces participant activement à la symbiose. Il a récemment été proposé, par Liping Zhao (Rutgers University, New Jersey, États-Unis), de réduire la diversité en considérant un angle plus fonctionnel et en réunissant en « guildes » des espèces bactériennes différentes mais associées en réseaux restreints contribuant à des fonctions – surtout métaboliques – similaires [8]. Sur ces bases, peut-on définir une dysbiose et toutes les dysbioses sont-elles pathogènes ? Peut-on approcher autrement le « microbiote ancestral » ?
L’idéal serait de pouvoir se référer à des microbiotes intestinaux humains « anciens », au minimum antérieurs à l’introduction des sulfamides et des antibiotiques. L’archéomicrobiologie, consistant à recueillir des échantillons fécaux historiquement datés et de les soumettre au séquençage de nouvelle génération (NGS) de leur ADN fossile, a permis l’analyse génomique des agents pathogènes responsables des grandes épidémies passées, comme la peste. De nombreuses difficultés s’opposent cependant à la caractérisation des écosystèmes complexes, dont l’hétérogénéité en qualité de l’ADN fossile, la contamination par des ADN de bactéries telluriques contemporaines, et le choix des échantillons contrôles. Réflexion et adaptations techniques sont néanmoins en cours [9].
Des approches utilisant des échantillons issus d’êtres vivants contemporains existent, comme la comparaison par métagénomique de la diversification phylogénique des taxons5 du microbiote fécal de grands primates et d’êtres humains qui a clairement montré la conservation d’un cœur microbien ancestral lors de la transition des grands primates vers les humanoïdes. En regard, cette étude a aussi montré que cette transition s’était accompagnée de la perte de centaines de taxons diversifiés à l’occasion de l’établissement de la co-évolution chez les hominidés. Cette perte atteignait 44 % en Afrique, et 54 % lorsque la comparaison était faite avec des données issues de sujets vivant dans des régions industrialisées, révélant une perte de diversité de 10 % entre populations vivant dans des régions plus ou moins touchées par le mode de vie moderne [10].
La comparaison par métataxonomique 16S, ou métagénomique du microbiote fécal de populations vivant dans des zones radicalement différentes en regard de leur état de développement socio-économique, représente une autre approche. Une étude pionnière publiée en 2012, comparant par métataxonomique 16S des échantillons fécaux de sujets de tous âges, vivant aux États-Unis, au Malawi et dans des communautés amérindiennes d’Amazonie, avait montré une diminution de diversité en taxons bactériens – particulièrement chez les enfants – entre les sujets nord-américains et les autres, la réduction de diversité atteignant 50 % entre nord-américains et amérindiens [11]. Une étude plus récente focalisée sur une population amérindienne isolée du monde extérieur depuis l’implantation de ses pionniers il y a 11 000 ans, a aussi montré une extraordinaire diversité des microbiotes fécal et buccal, renforçant l’idée d’une réduction de diversité du microbiote des populations des régions industrialisées. Étonnamment, malgré l’absence de consommation d’antibiotiques, le résistome – c’est-à-dire la gamme de gènes potentiels de résistance aux antibiotiques – de ces microbiotes était très riche… [12].
Il convient donc de mettre en place un réseau global, méthodologiquement standardisé, assurant une surveillance prospective de l’évolution de la richesse et de la diversité du microbiote intestinal dans des populations représentatives de différents modes de vie. Les populations en transition économique seront particulièrement intéressantes. Au-delà des études individuelles, on considère de plus les études globales sur les eaux usées en amont des stations d’épuration, qui sont un reflet du « microbiote urbain » [13]. Cette approche menée prospectivement, couplée à l’identification et au dosage de médicaments, permet d’établir des corrélations entre consommation médicamenteuse et variation qualitative et quantitative des « microbiotes urbains ». Quels sont les « stresseurs » environnementaux responsables de cette réduction de diversité microbienne dans les populations des régions industrialisées ? Ceci n’est qu’un angle d’approches plus globales visant à définir « l’exposome occidental » et son impact sur les écosystèmes humains, animaux et végétaux. En milieu urbain, des approches se développent en parallèle afin de fournir des données prospectives sur la flore aérienne, en intérieur et en extérieur, avec la difficulté d’analyse métagénomique fiable d’échantillons de très faible biomasse [14].
On voit aujourd’hui très légitimement se développer une approche « une santé » (One health) pour laquelle des progrès sont indispensables, non tant dans les technologies « omiques » que dans le traitement standardisé des « big data » générées par ces approches. Audelà, une approche prospective globale sera indispensable pour suivre l’impact des changements climatiques sur les écosystèmes microbiens en rapport avec la santé humaine.
L’ensemble des données actuelles confirme cette perte globale de diversité dans le contexte de l’anthropocène. Sur ce fond global, il convient de définir plus précisément ce que l’on peut considérer – maintenant à l’échelle individuelle – comme une dysbiose afin de préparer l’étape ultérieure de démonstration de causalité en termes de pathogénicité.
La surconsommation d’antibiotiques : paradigme de stresseur environnemental « anthropique » des microbiotes humains et au-delà
Il est de plus en plus évident que la surconsommation d’antibiotiques, ainsi que d’autres médicaments à effet antibactérien caché, comme certains psychotropes [15], non seulement mettent en tension les écosystèmes microbiens par sélection et dissémination de souches antibiorésistantes et de gènes d’antibiorésistance, comme récemment rappelé par l’OMS6, mais peuvent aussi altérer richesse et diversité des écosystèmes microbiens dans leur globalité. En effet, au-delà de leur consommation encore excessive en médecines humaine et vétérinaire, les antibiotiques sont de plus en plus largement utilisés – hors Union Européenne – en élevages, surtout porcin et aviaire, et en aquaculture, dans un but de prophylaxie anti-infectieuse, mais aussi comme facteurs de croissance. Chaque année, plus de 300 000 tonnes estimées d’antibiotiques sont déversés intacts dans l’environnement, dont plus de 50 % ne sont pas justifiés par des indications thérapeutiques médicales ou vétérinaires. Les antibiotiques sont devenus des polluants environnementaux retrouvés dans 65 % des fleuves et des rivières de la planète [16]. Le défi qui s’impose est donc de sauver en même temps les antibiotiques et les écosystèmes microbiens, particulièrement ceux associés à l’homme.
Marqueurs définissant une dysbiose « anthropique »
Il est difficile d’offrir une définition claire d’une dysbiose. De plus, toute altération en richesse et en diversité d’un microbiote n’est pas nécessairement pathogène. On peut néanmoins avancer un cadre global associant une perte de richesse et de diversité microbiennes, touchant en particulier le noyau commun d’espèces physiologiquement essentielles à la symbiose, même si les frontières en demeurent imprécises. C’est le cas de la disparition d’espèces reconnues pour être des déterminants clés de la santé de l’écosystème ou de son hôte. On peut citer Faecalibacterium prautznitzii, dont l’absence fut très tôt corrélée à la survenue de maladies inflammatoires chroniques de l’intestin (MICI) comme la maladie de Crohn [17]. Il en est de même pour Akkermansia muciniphila, largement considéré comme un déterminant de la qualité fonctionnelle de la barrière intestinale [18].
Ces déficits en richesse et diversité microbiennes sont souvent associés à la prolifération incontrôlée de pathobiontes, des espèces microbiennes opportunistes, non pathogènes tant qu’elles demeurent intégrées dans un écosystème sain, mais devenant pathogènes lorsque la dégradation de leur écosystème hôte en libère le potentiel de croissance et de virulence [19]. Les pathobiontes peuvent être classés en plusieurs catégories.
Les pathobiontes « épidémiques », comme Clostridium difficile, dont la croissance, après contamination, est stimulée par la dégradation du microbiote intestinal par un traitement antibiotique [20].
Les pathobiontes « résidents » qui sont des hôtes naturels du microbiote intestinal humain, jouant, par exemple, un rôle dans la maturation du système immunitaire, mais dont la croissance incontrôlée dans le contexte d’une dysbiose devient pathogène. C’est le cas de la bactérie filamenteuse segmentée (SFB), essentielle au développement de l’immunité innée du nourrisson par la maturation des lymphocytes Th17 [21], mais susceptible de causer une inflammation intestinale en cas de dysbiose permettant sa croissance incontrôlée [22]. Escherichia coli, et les entérobactéries en général, demeurent minoritaires dans le microbiote intestinal quoi qu’y jouant sans doute un rôle important dans la stimulation physiologique de l’immunité innée, mais leur prolifération à la surface muqueuse est clairement établie dans la maladie de Crohn [23]. Certains clones (ou pathovars) d’E. coli, comme les souches productrices de Cdt (cytolethal distending toxin), affectent la prolifération épithéliale intestinale et se rapprochent de vrais pathogènes, montrant que le concept de pathobiontes peut recouvrir un large éventail de situations entre commensalisme et pathogénicité [24]. Dans cette même catégorie de pathobiontes résidents, on peut, par exemple, ranger les clones de Bacteroides fragilis producteurs de cytotoxine [25] et Streptococcus gallolyticus [26].
Les pathobiontes « ectopiques » sont des espèces éventuellement pathogènes de la flore orale (périodontopathies) qui se délocalisent dans le microbiote colique et y proliférent, surtout à la surface muqueuse, dans un contexte dysbiotique. La présence de ces espèces, telles Fusobacterium nucleatum, Porphyromonas gingivalis et Parvimonas micra, est corrélée à la survenue de colites inflammatoires [27] et de cancer colorectal (CRC) [28, 29]. Il est possible que ces bactéries orales agissent en synergie pour stimuler le processus oncogène [30]. La microbiologie cellulaire, qui a permis, au fil des dernières décennies, de déchiffrer le dialogue moléculaire entre bactéries pathogènes, cellules et tissus de l’hôte, doit maintenant s’appliquer aux pathobiontes, voire, plus largement, aux espèces clés de la symbiose hôte-microbiome. La difficulté réside à plusieurs niveaux : le stade de la pathologie concernée auquel intervient le pathobionte n’est pas toujours évident ; les phénotypes fonctionnels à rechercher ne sont pas nécessairement aussi clairs que pour un agent pathogène « vrai » (adhérence, invasion, mort cellulaire, régulation d’une cascade inflammatoire, etc.). Ce peuvent être en effet des voies métaboliques [31] ou des altérations épigénétiques [32], dont le criblage peut être très lourd. Les cibles cellulaires, enfin, peuvent être difficiles à déterminer. Il s’agit là d’une science multidisciplinaire qui est indispensable pour établir le lien de causalité incontournable entre pathobionte/ dysbiose et maladie.
Dysbioses et causalité des maladies : les postulats de Koch revisités
L’établissement d’un lien de causalité entre dysbiose et maladie est au cœur de l’analyse des conséquences de l’altération des microbiotes humains sur la santé. Cette démarche demande une réactualisation des « postulats de Koch »7 [33] établis dans le but de confirmer la pathogénicité d’une espèce microbienne unique. Le déchiffrage du mode d’action des pathobiontes s’en rapproche, mais en tant qu’opportunistes, leur pathogénicité ne se conçoit que dans leur synergie avec d’autres pathobiontes et leur interaction complexe avec le microbiote dégradé auxquels ils appartiennent. Ce lien de causalité doit être, quoiqu’il en soit, établi à l’échelle d’un écosystème microbien complexe en rapport avec la perte de certaines espèces et l’acquisition de pathobiontes. C’est ce qui a été baptisé « postulats écologiques de Koch » [34].
Afin de consolider ces postulats revisités, de nouvelles approches expérimentales sont requises, qu’il s’agisse de modèles cellulaires ou ex vivo, sur des organoïdes intestinaux, l’intégralité de la flore étant cependant difficile à y maintenir. Il peut aussi s’agir de modèles in vivo, essentiellement chez la souris – en particulier axénique8 – permettant une recolonisation par des échantillons fécaux humains dysbiotiques en regard de contrôles. L’expérience pionnière dans ce contexte fut la démonstration, en 2006, par le groupe de Jeff Gordon (Washington University School of Medicine, St. Louis, États-Unis), que la transplantation de selles de souris obèses déficientes en leptine (souris ob-/ob-) à des souris de corpulence normale, entraînait chez ces dernières une obésité [35]. Cette expérience prouva que le microbiome seul pouvait significativement engager la survenue d’un désordre physiologique majeur, s’expliquant, dans ce cas, par une capacité accrue du microbiome des souris obèses de « récolter » plus d’énergie de leur ration alimentaire que celui d’une souris de corpulence normale. Une plus grande représentation de clostridiales, bactéries productrices d’acides gras à chaînes courtes (SCFA) à haute valeur énergétique à partir des polyosides alimentaires complexes, a été considérée comme décisive. Un contexte pro-inflammatoire est aussi envisagé, l’obésité étant associée à une inflammation chronique de bas grade induite par une endotoxémie9 directement liée à la dysbiose [36]. Ce domaine d’interface entre microbiome et métabolisme est maintenant bien établi [37]. En complément de ces études expérimentales, les études interventionnelles visant à la restauration d’un microbiome non pathogène, qu’il s’agisse de complémentation partielle d’espèces manquantes ou de transfert fécal, fait partie intégrante de l’établissement d’un lien de causalité.
À ce stade, un bilan de la relation entre dégradation du microbiome humain et maladies chroniques non-transmissibles post-modernes s’impose, assorti d’exemples pour lesquels un lien de corrélation – plus rarement de causalité – a été établi (Tableau I). À la lumière de ces exemples, on envisagera comment le concept de probiotiques peut être rationnellement réévalué pour la prophylaxie et le traitement de ces maladies.
Pour beaucoup de maladies largement « post-modernes » (citées dans le Tableau I), des études ont récemment apporté des résultats renforçant l’évidence d’un lien de causalité entre dysbiose et maladie. C’est particulièrement le cas chez l’adulte, pour les maladies inflammatoires, métaboliques et le cancer. Les évidences reposent beaucoup sur des modèles expérimentaux établis chez la souris [38]. En plusieurs circonstances, des études cliniques interventionnelles par « thérapie microbienne » sont en projet ou déjà en cours. Il est clair que le succès clinique de telles interventions représentera l’ultime étape pour remplir l’intégralité des « postulats écologiques de Koch » tels que revisités.
Il semble cependant important de développer un thème : celui des dysbioses du nourrisson. Ceci pour plusieurs raisons. Le microbiote du nourrisson est vulnérable en ce qu’il est exposé en pleine période de construction, d’où le risque de perturbation des étapes clés des successions écologiques qui assurent sa complète maturation. De plus, toutes les agressions que peut subir ce microbiote dans cette période ultra-sensible où des phases essentielles de développement sont encore en cours, porte le risque de retards développementaux ou de fragilité future face à des maladies.
La survenue d’une dysbiose intestinale chez le nouveauné et le nourrisson – définie comme une réduction de diversité associée à la perte de taxons bénéfiques et/ ou à l’acquisition de pathobiontes – offre le cadre le plus solide pour interroger le rôle causal de ces dysbioses [39]. Une étude rétrospective impliquant près de 15 000 enfants aux États-Unis, a montré, suite à la prise d’au moins un antibiotique au cours des deux premières années de vie, un accroissement significatif de la survenue d’asthme précoce, de rhinite allergique, de dermatite atopique, de maladie cœliaque, de surpoids, voire d’obésité, mais aussi de syndromes d’hyper-activité et manque d’attention (ADHD) [40]. Ce sujet sensible a fait l’objet d’une récente revue [41]. Une méta-analyse a pu montrer - y compris en fonction des antibiotiques utilisés - une gamme de profils dysbiotiques créés chez les nourrissons traités [42]. Des études plus ciblées ont montré une nette corrélation entre la prise d’antibiotiques dans les deux premières années de vie et une augmentation du risque d’obésité pédiatrique pouvant atteindre plus de 20 % [43].
Un lien de causalité fut suggéré très tôt par des expériences au cours desquelles les selles de souris recevant dès la naissance de faibles doses de pénicilline étaient transplantées à des souris axéniques soumises à un régime riche en graisses. En comparaison de souris axéniques transplantées avec une flore d’animaux n’ayant pas reçu de pénicilline et recevant le même régime riche en graisses, les premières développaient une obésité plus rapidement que les secondes, associée à une activation intestinale plus marquée de gènes métaboliques et inflammatoires. [44]. Ces observations consolident l’hypothèse du rôle important d’une perturbation de la dynamique des successions écologiques aboutissant à un microbiome eubiotique dans les deux premières années de vie vis-à-vis de perturbations de la maturation de l’immunité, du métabolisme et du développement psychomoteur post-natal de l’enfant.
Notons qu’à côté de l’usage des antibiotiques, un autre déterminant anthropique de dysbiose du nourrisson – particulièrement impliqué dans la survenue de l’asthme précoce – est l’accouchement par césarienne [45]. Cette situation particulière offre la possibilité de démontrer le lien de causalité entre dysbiose et pathologie, en réensemençant le nouveau-né avec un échantillon de flore vaginale maternelle [46], voire intestinale ou une combinaison des deux. Cette approche de complémentation récemment débutée va demander un certain recul pour apprécier son efficacité. On parle de plus en plus à ce sujet de « réensauvagement » (rewilding).
Si des expériences murines similaires à celles réalisées dans le contexte de l’obésité avaient montré que l’administration précoce d’antibiotiques entraînait, chez des lignées sensibles, une inflammation bronchique semblable à l’asthme précoce, des données humaines manquaient. Un travail décisif, fondé sur la cohorte CHILD au Canada, a montré, sur plusieurs centaines d’enfants, que l’existence d’une dysbiose dans la fenêtre des 100 premiers jours de vie était corrélée à la survenue d’un asthme du nourrisson. Les quatre types de bactéries intestinales manquant chez les nourrissons sujets à l’asthme étaient Faecalibacterium, Lachnospira, Veillonella et Rothia (FLVR). Afin de consolider le lien de causalité, des souris sensibles colonisées par les microbiotes dysbiotiques ont développé une inflammation bronchique, contrairement aux souris contrôles colonisées par les microbiotes eubiotiques. Plus encore, l’ajout aux microbiotes dysbiotiques des quatre genres manquants effaçait la survenue de l’inflammation bronchique [47].
Maladies associées à une dysbiose.
Perte de diversité du microbiote humain, dysbiose et lien de causalité des maladies « post-modernes »
Il n’est pas question ici de proposer une revue détaillée des approches prophylactiques et thérapeutiques innovantes que propose la « médecine microbienne » [48]. Tenons-nous en aux grands principes : il est clair que la situation globale d’appauvrissement du microbiote humain qui s’étend à l’allure que lui imposent les stresseurs alimentaires, médicamenteux, et plus largement environnementaux, représente un défi aussi gigantesque en matière de réversion que de prévenir l’émission de gaz à effets de serre. Il y a là, en effet, des évolutions sociales, économiques, anthropologiques, culturelles, éventuellement existentielles pour les populations des pays en transition économique, qui rend modeste sur l’établissement d’objectifs globaux de réduction des impacts anthropiques. Si l’on pouvait au minimum atteindre un plateau, on pourrait compter sur un certain degré de résilience humaine par rapport à la réduction globale de la diversité de son microbiote et se donner le temps nécessaire à l’éducation alimentaire, à la parcimonie dans l’usage des médicaments et à une meilleure connaissance des exposomes et de la manière de s’en protéger. C’est pour cela que l’éducation axée sur la reconnaissance du rôle critique de la santé des écosystèmes microbiens en rapport avec le vivant est essentielle10. Cette approche pédagogique touche au concept de prébiotiques et à l’importance des fibres dans l’équilibre alimentaire, en ce qu’elles favorisent la repopulation des microbiotes par des clostridiales producteurs, en présence de ces nutriments, d’acides gras à chaînes courtes [47], leur perte étant une caractéristique commune des dysbioses.
En complément de ces approches génériques, des approches plus ciblées de prévention de survenue de dysbioses responsables de maladies dans des conditions clairement établies est probablement le cadre actuellement le plus prometteur [49]. La dysbiose précoce du nourrisson, due à un accouchement par césarienne, a donné lieu à suffisamment d’études concernant le risque de survenue d’asthme précoce et d’obésité, et à des données claires concernant les espèces microbiennes manquantes et le lien de causalité de leur absence avec ces maladies pour justifier une complémentation précoce par un échantillon de microbiote vaginal et/ou intestinal en post-partum, ou par un cocktail des microbes manquants. Cette approche méritera d’être ajustée concernant la prévention de l’installation d’une dysbiose après prise d’antibiotiques [50].
Pour d’autres situations de dysbiose, particulièrement celles dont les conséquences sont dominées par une forte composante inflammatoire, il est clair que des espèces comme F. prauznizii et A. mucinophila sont en première ligne en tant que « probiotiques de nouvelle génération ».
L’approche du transfert fécal thérapeutique à partir de donneurs sains a largement fait ses preuves dans le traitement des colites ulcéro-membraneuses récidivantes à Clostridioides difficile, une situation typique de dysbiose post antibiotique. C’est clairement une situation où les « postulats écologiques de Koch » sont remplis [51]. Le transfert fécal thérapeutique tend à s’élargir dans ses indications [52]. Il pourra ainsi s’adresser au diabète de type 2 [53], ainsi qu’aux maladies inflammatoires chroniques de l’intestin [54]. L’apport du transfert fécal thérapeutique est également exploré dans des contextes d’impasses thérapeutiques, comme la maladie du greffon contre l’hôte, suite à une greffe de moelle osseuse dans le traitement des hémopathies malignes11. De grandes séries sont maintenant nécessaires pour consolider cette approche qui n’est pas dénuée de difficultés, exigeant des efforts de standardisation des procédures et de présentation galénique, de gestion du risque et de considérations éthiques.
La « médecine microbienne » est en réalité la médecine de l’humain-microbien. La dysbiose a longtemps été considérée comme une altération du microbiome au sens strict, mais force est de constater que, dans les conditions cliniques nombreuses où la dysbiose a été décrite (Tableau I), lui sont très souvent associés une perméabilité intestinale, une inflammation de niveau variable et un stress oxydant. Plus que la seule altération du microbiome, ces quatre leviers signent ainsi une altération de la symbiose hôte-microbes [55, 56]. Ces quatre leviers peuvent de plus établir un cercle vicieux, si bien que si le système global est poussé au-delà de la robustesse qui lui permet de rester résilient, il pourra atteindre un point de bascule et s’installer durablement dans un état stable alternatif de symbiose altérée suite à ce que les théoriciens des systèmes complexes appellent une « transition critique » [57, 58]. Ceci aura des conséquences en matière de diagnostic et de pronostic, rendant ainsi pertinent d’associer aux paramètres du microbiote dysbiotique, des paramètres de l’hôte : niveau d’inflammation, de perméabilité intestinale et de stress oxydant [59]. En matière de prévention et de thérapie, il faudra concevoir une combinaison d’actifs pouvant agir conjointement sur les leviers de la symbiose altérée (Figure 1). Par exemple, une étude a récemment montré le potentiel d’une combinaison d’actifs alimentaires à se montrer aussi efficace qu’une molécule antidépresseur à restaurer un comportement normal dans un modèle de souris stressées [60]. Un essai clinique est en cours afin de valider chez des patients dépressifs cette approche typique du potentiel de la « médecine microbienne ».
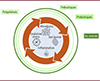 |
Figure 1. Exemple d’actifs alimentaires pouvant être combinés pour une action préventive ou thérapeutique visant à restaurer une symbiose microbiote-hôte fonctionnelle. |
Conclusion
Une « médecine microbienne » ou « médecine de l’humain microbien » est née. Comme toute révolution naissante, elle porte sa part de doutes et d’excès de confiance dans le concept. C’est ainsi qu’ont souvent évolué les recherches dans le domaine biomédical. Cette nouvelle médecine a du sens car elle repose sur l’observation de dysfonctions d’une symbiose animal/homme-microbes fruit d’une co-évolution millénaire. Elle est en fait une partie de la « médecine environnementale », maintenant largement admise, où le microbiome en tant qu’environnement tient une place de choix. C’est la conjonction d’études expérimentales et d’approches cliniques interventionnelles rigoureusement réalisées et interprétées, loin des surévaluations médiatiques incontrôlées, qui permettra de définir les contours exacts de cette nouvelle discipline et des attentes raisonnables que l’on peut en avoir. C’est aussi une médecine moderne, de « big data » où l’intelligence artificielle joue déjà son plein rôle. Moderne enfin, car elle s’inscrit au cœur des préoccupations écologiques de notre siècle et dans ce cadre « une seule santé » qui en est une composante incontournable.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Mise à jour dans : https://ourworldindata.org/grapher/burden-of-disease
Références
- Bach JF. The effect of infections on susceptibility to autoimmune and allergic diseases. N Engl J Med 2002 ; 347 : 911–20. [CrossRef] [PubMed] [Google Scholar]
- Blaser MJ. Missing microbes : how the overuse of antibiotics is fueling our modern plagues. New York : Henry Holt and Company, 2014 : 273 p. [Google Scholar]
- Anwar H, Iftikhar A, Muzaffar H, et al. Biodiversity of Gut Microbiota: Impact of Various Host and Environmental Factors. Biomed Res Int 2021 ; 2021 : 5575245. [CrossRef] [PubMed] [Google Scholar]
- Lloyd-Price J, Abu-Ali G, Huttenhower C. The healthy human microbiome. Genome Med 2016 ; 8 : 51. [CrossRef] [PubMed] [Google Scholar]
- Rinninella E, Raoul P, Cintoni M, et al. What is the Healthy Gut Microbiota Composition? A Changing Ecosystem across Age, Environment, Diet, and Diseases. Microorganisms 2019 ; 7. [Google Scholar]
- Costea PI, Hildebrand F, Arumugam M, et al. Enterotypes in the landscape of gut microbial community composition. Nat Microbiol 2018 ; 3 : 8–16. [Google Scholar]
- Tap J, Lejzerowicz F, Cotillard A, et al. Global branches and local states of the human gut microbiome define associations with environmental and intrinsic factors. Nat Commun 2023 ; 14 : 3310. [CrossRef] [PubMed] [Google Scholar]
- Wu G, Zhao N, Zhang C, et al. Guild-based analysis for understanding gut microbiome in human health and diseases. Genome Med 2021 ; 13 : 22. [CrossRef] [PubMed] [Google Scholar]
- Amato KR, Jeyakumar T, Poinar H, Gros P. Shifting Climates, Foods, and Diseases: The Human Microbiome through Evolution. Bioessays 2019 ; 41 : e1900034. [CrossRef] [PubMed] [Google Scholar]
- Sanders JG, Sprockett DD, Li Y, et al. Widespread extinctions of co-diversified primate gut bacterial symbionts from humans. Nat Microbiol 2023 ; 8 : 1039–50. [CrossRef] [PubMed] [Google Scholar]
- Yatsunenko T, Rey FE, Manary MJ, et al. Human gut microbiome viewed across age and geography. Nature 2012 ; 486 : 222–7. [CrossRef] [PubMed] [Google Scholar]
- Clemente JC, Pehrsson EC, Blaser MJ, et al. The microbiome of uncontacted Amerindians. Sci Adv 2015 ; 1 : e1500183. [CrossRef] [PubMed] [Google Scholar]
- Marechal V, Maday Y, Wallet C, et al. Wastewater-based epidemiology: Retrospective, current status, and future prospects. Anaesth Crit Care Pain Med 2023 ; 42 : 101251. [CrossRef] [PubMed] [Google Scholar]
- Luhung I, Uchida A, Lim SBY, et al. Experimental parameters defining ultra-low biomass bioaerosol analysis. NPJ Biofilms Microbiomes 2021 ; 7 : 37. [CrossRef] [PubMed] [Google Scholar]
- Maier L, Pruteanu M, Kuhn M, et al. Extensive impact of non-antibiotic drugs on human gut bacteria. Nature 2018 ; 555 : 623–8. [CrossRef] [PubMed] [Google Scholar]
- Wilkinson JL, Boxal ABA, Kolpin DW. A Novel Method to Characterise Levels of Pharmaceutical Pollution in Large-Scale Aquatic Monitoring Campaigns. Appl Sci 2019 ; 9. [Google Scholar]
- Sokol H, Pigneur B, Watterlot L, et al. Faecalibacterium prausnitzii is an anti-inflammatory commensal bacterium identified by gut microbiota analysis of Crohn disease patients. Proc Natl Acad Sci U S A 2008 ; 105 : 16731–6. [CrossRef] [PubMed] [Google Scholar]
- Cani PD, Depommier C, Derrien M, et al. Akkermansia muciniphila: paradigm for next-generation beneficial microorganisms. Nat Rev Gastroenterol Hepatol 2022 ; 19 : 625–37. [CrossRef] [PubMed] [Google Scholar]
- Caballero-Flores G, Pickard JM, Nunez G. Microbiota-mediated colonization resistance: mechanisms and regulation. Nat Rev Microbiol 2023 ; 21 : 347–60. [CrossRef] [PubMed] [Google Scholar]
- Crobach MJT, Vernon JJ, Loo VG, et al. Understanding Clostridium difficile Colonization. Clin Microbiol Rev 2018 ; 31. [Google Scholar]
- Gaboriau-Routhiau V, Rakotobe S, Lecuyer E, et al. The key role of segmented filamentous bacteria in the coordinated maturation of gut helper T cell responses. Immunity 2009 ; 31 : 677–89. [CrossRef] [PubMed] [Google Scholar]
- Stepankova R, Powrie F, Kofronova O, et al. Segmented filamentous bacteria in a defined bacterial cocktail induce intestinal inflammation in SCID mice reconstituted with CD45RBhigh CD4+ T cells. Inflamm Bowel Dis 2007 ; 13 : 1202–11. [CrossRef] [PubMed] [Google Scholar]
- Mirsepasi-Lauridsen HC, Vallance BA, Krogfelt KA, Petersen AM. Escherichia coli Pathobionts Associated with Inflammatory Bowel Disease. Clin Microbiol Rev 2019 ; 32. [Google Scholar]
- De Rycke J, Milon A, Oswald E. Necrotoxic Escherichia coli (NTEC): two emerging categories of human and animal pathogens. Vet Res 1999 ; 30 : 221–33. [PubMed] [Google Scholar]
- Valguarnera E, Wardenburg JB. Good Gone Bad: One Toxin Away From Disease for Bacteroides fragilis. J Mol Biol 2020 ; 432 : 765–85. [CrossRef] [PubMed] [Google Scholar]
- Pasquereau-Kotula E, Martins M, Aymeric L, Dramsi S. Significance of Streptococcus gallolyticus subsp. gallolyticus Association With Colorectal Cancer. Front Microbiol 2018 ; 9 : 614. [CrossRef] [PubMed] [Google Scholar]
- Dinakaran V, Mandape SN, Shuba K, et al. Identification of Specific Oral and Gut Pathogens in Full Thickness Colon of Colitis Patients: Implications for Colon Motility. Front Microbiol 2018 ; 9 : 3220. [Google Scholar]
- Koliarakis I, Messaritakis I, Nikolouzakis TK, et al. Oral Bacteria and Intestinal Dysbiosis in Colorectal Cancer. Int J Mol Sci 2019 ; 20. [PubMed] [Google Scholar]
- Rubinstein MR, Baik JE, Lagana SM, et al. Fusobacterium nucleatum promotes colorectal cancer by inducing Wnt/beta-catenin modulator Annexin A1. EMBO Rep 2019 ; 20. [Google Scholar]
- Flynn KJ, Baxter NT, Schloss PD. Metabolic and Community Synergy of Oral Bacteria in Colorectal Cancer. mSphere 2016 ; 1. [Google Scholar]
- Araujo JR, Tazi A, Burlen-Defranoux O, et al. Fermentation Products of Commensal Bacteria Alter Enterocyte Lipid Metabolism. Cell Host Microbe 2020 ; 27 : 358–75 e7. [CrossRef] [PubMed] [Google Scholar]
- Bergsten E, Mestivier D, Donnadieu F, et al. Parvimonas micra, an oral pathobiont associated with colorectal cancer, epigenetically reprograms human colonocytes. Gut Microbes 2023 ; 15 : 2265138. [CrossRef] [PubMed] [Google Scholar]
- [Die Atiologie der Tuberculose. Facsimile of the original contribution by Robert Koch in “Berliner Klinische Wochenschrift” 10 April 1882. Fortschr Med 1982 ; 100 : 539. [PubMed] [Google Scholar]
- Vonaesch P, Anderson M, Sansonetti PJ. Pathogens, microbiome and the host: emergence of the ecological Koch’s postulates. FEMS Microbiol Rev 2018 ; 42 : 273–92. [CrossRef] [PubMed] [Google Scholar]
- Turnbaugh PJ, Ley RE, Mahowald MA, et al. An obesity-associated gut microbiome with increased capacity for energy harvest. Nature 2006 ; 444 : 1027–31. [CrossRef] [PubMed] [Google Scholar]
- Kawai T, Autieri MV, Scalia R. Adipose tissue inflammation and metabolic dysfunction in obesity. Am J Physiol Cell Physiol 2021 ; 320 : C375–C91. [CrossRef] [PubMed] [Google Scholar]
- Fan Y, Pedersen O. Gut microbiota in human metabolic health and disease. Nat Rev Microbiol 2021 ; 19 : 55–71. [CrossRef] [PubMed] [Google Scholar]
- Al-Asmakh M, Zadjali F. Use of Germ-Free Animal Models in Microbiota-Related Research. J Microbiol Biotechnol 2015 ; 25 : 1583–8. [CrossRef] [PubMed] [Google Scholar]
- Parkin K, Christophersen CT, Verhasselt V, et al. Risk Factors for Gut Dysbiosis in Early Life. Microorganisms 2021 ; 9. [PubMed] [Google Scholar]
- Aversa Z, Atkinson EJ, Schafer MJ, et al. Association of Infant Antibiotic Exposure With Childhood Health Outcomes. Mayo Clin Proc 2021 ; 96 : 66–77. [CrossRef] [PubMed] [Google Scholar]
- Renz H, Skevaki C. Early life microbial exposures and allergy risks: opportunities for prevention. Nat Rev Immunol 2021 ; 21 : 177–91. [CrossRef] [PubMed] [Google Scholar]
- McDonnell L, Gilkes A, Ashworth M, et al. Association between antibiotics and gut microbiome dysbiosis in children: systematic review and meta-analysis. Gut Microbes 2021 ; 13 : 1–18. [CrossRef] [PubMed] [Google Scholar]
- Scott FI, Horton DB, Mamtani R, et al. Administration of Antibiotics to Children Before Age 2 Years Increases Risk for Childhood Obesity. Gastroenterology 2016 ; 151 : 120–9 e5. [CrossRef] [PubMed] [Google Scholar]
- Cox LM, Yamanishi S, Sohn J, et al. Altering the intestinal microbiota during a critical developmental window has lasting metabolic consequences. Cell 2014 ; 158 : 705–21. [CrossRef] [PubMed] [Google Scholar]
- Dominguez-Bello MG, Costello EK, Contreras M, et al. Delivery mode shapes the acquisition and structure of the initial microbiota across multiple body habitats in newborns. Proc Natl Acad Sci U S A 2010 ; 107 : 11971–5. [CrossRef] [PubMed] [Google Scholar]
- Dominguez-Bello MG, De Jesus-Laboy KM, Shen N, et al. Partial restoration of the microbiota of cesarean-born infants via vaginal microbial transfer. Nat Med 2016 ; 22 : 250–3. [CrossRef] [PubMed] [Google Scholar]
- Arrieta MC, Stiemsma LT, Dimitriu PA, et al. Early infancy microbial and metabolic alterations affect risk of childhood asthma. Sci Transl Med 2015 ; 7 : 307ra152. [CrossRef] [PubMed] [Google Scholar]
- Gebrayel P, Nicco C, Al Khodor S, et al. Microbiota medicine: towards clinical revolution. J Transl Med 2022 ; 20 : 111. [CrossRef] [PubMed] [Google Scholar]
- Korpela K, de Vos WM. Infant gut microbiota restoration: state of the art. Gut Microbes 2022 ; 14 : 2118811. [CrossRef] [PubMed] [Google Scholar]
- Korpela K, Helve O, Kolho KL, et al. Maternal Fecal Microbiota Transplantation in Cesarean-Born Infants Rapidly Restores Normal Gut Microbial Development: A Proof-of-Concept Study. Cell 2020 ; 183 : 324–34 e5. [CrossRef] [PubMed] [Google Scholar]
- van Nood E, Vrieze A, Nieuwdorp M, et al. Duodenal infusion of donor feces for recurrent Clostridium difficile. N Engl J Med 2013 ; 368 : 407–15. [Google Scholar]
- Hanssen NMJ, de Vos WM, Nieuwdorp M. Fecal microbiota transplantation in human metabolic diseases: From a murky past to a bright future? Cell Metab 2021 ; 33 : 1098–110. [CrossRef] [PubMed] [Google Scholar]
- Vrieze A et al. Transfer of intestinal microbiota from lean donors increases insulin sensitivity in individuals with metabolic syndrome. Gastroenterology. 2012 ; 143 :913–916 [CrossRef] [PubMed] [Google Scholar]
- Sokol H, Landman C, Seksik P, et al. Fecal microbiota transplantation to maintain remission in Crohn’s disease: a pilot randomized controlled study. Microbiome 2020 ; 8 : 12. [CrossRef] [PubMed] [Google Scholar]
- Doré J, Ortega Ugalde S. Human-microbes symbiosis in health and disease, on earth and beyond planetary boundaries. Frontiers in Astronomy and Space Sciences 2023 ; 10. [Google Scholar]
- Malard F, Dore J, Gaugler B, Mohty M. Introduction to host microbiome symbiosis in health and disease. Mucosal Immunol 2021 ; 14 : 547–54. [CrossRef] [PubMed] [Google Scholar]
- van de Guchte M, Blottiere HM, Dore J. Humans as holobionts: implications for prevention and therapy. Microbiome 2018 ; 6 : 81. [CrossRef] [PubMed] [Google Scholar]
- Van de Guchte M, Burz SD, Cadiou J, et al. Alternative stable states in the intestinal ecosystem: proof of concept in a rat model and a perspective of therapeutic implications. Microbiome 2020 ; 8 : 153. [CrossRef] [PubMed] [Google Scholar]
- van de Guchte M, Mondot S, Dore J. Dynamic Properties of the Intestinal Ecosystem Call for Combination Therapies, Targeting Inflammation and Microbiota, in Ulcerative Colitis. Gastroenterology 2021 ; 161 : 1969–81 e12. [CrossRef] [PubMed] [Google Scholar]
- Faucher P, Dries A, Mousset PY, et al. Synergistic effects of Lacticaseibacillus rhamnosus GG, glutamine, and curcumin on chronic unpredictable mild stress-induced depression in a mouse model. Benef Microbes 2022 ; 13 : 253–64. [CrossRef] [PubMed] [Google Scholar]
- Mangin I, Bonnet R, Seksik P, et al. Molecular inventory of faecal microflora in patients with Crohn’s disease. FEMS Microbiol Ecol 2004 ; 50 : 25–36. [CrossRef] [PubMed] [Google Scholar]
- Lepage P, Hasler R, Spehlmann ME, et al. Twin study indicates loss of interaction between microbiota and mucosa of patients with ulcerative colitis. Gastroenterology 2011 ; 141 : 227–36. [CrossRef] [PubMed] [Google Scholar]
- Hartstra AV, Bouter KE, Backhed F, Nieuwdorp M. Insights into the role of the microbiome in obesity and type 2 diabetes. Diabetes Care 2015 ; 38 : 159–65. [CrossRef] [PubMed] [Google Scholar]
- Burcelin R, Serino M, Chabo C, et al. Gut microbiota and diabetes: from pathogenesis to therapeutic perspective. Acta Diabetol 2011 ; 48 : 257–73. [CrossRef] [PubMed] [Google Scholar]
- Aron-Wisnewsky J, Gaborit B, Dutour A, Clement K. Gut microbiota and non-alcoholic fatty liver disease: new insights. Clin Microbiol Infect 2013 ; 19 : 338–48. [CrossRef] [PubMed] [Google Scholar]
- Giongo A, Gano KA, Crabb DB, et al. Toward defining the autoimmune microbiome for type 1 diabetes. ISME J 2011 ; 5 : 82–91. [CrossRef] [PubMed] [Google Scholar]
- Nadal I, Donant E, Ribes-Koninckx C, et al. Imbalance in the composition of the duodenal microbiota of children with coeliac disease. J Med Microbiol 2007 ; 56 : 1669–74. [CrossRef] [PubMed] [Google Scholar]
- Sobhani I, Tap J, Roudot-Thoraval F, et al. Microbial dysbiosis in colorectal cancer (CRC) patients. PLoS One 2011 ; 6 : e16393. [CrossRef] [PubMed] [Google Scholar]
- Abrahamsson TR, Jakobsson HE, Andersson AF, et al. Low diversity of the gut microbiota in infants with atopic eczema. J Allergy Clin Immunol 2012 ; 129 : 434–40, 40 e1-2. [CrossRef] [PubMed] [Google Scholar]
- Finegold SM, Molitoris D, Song Y, et al. Gastrointestinal microflora studies in late-onset autism. Clin Infect Dis 2002 ; 35 : S6–S16. [CrossRef] [PubMed] [Google Scholar]
- Cersosimo MG, Raina GB, Pecci C, et al. Gastrointestinal manifestations in Parkinson’s disease: prevalence and occurrence before motor symptoms. J Neurol 2013 ; 260 : 1332–8. [CrossRef] [PubMed] [Google Scholar]
- Wang Z, Klipfell E, Bennett BJ, et al. Gut flora metabolism of phosphatidylcholine promotes cardiovascular disease. Nature 2011 ; 472 : 57–63. [CrossRef] [PubMed] [Google Scholar]
Liste des tableaux
Liste des figures
 |
Figure 1. Exemple d’actifs alimentaires pouvant être combinés pour une action préventive ou thérapeutique visant à restaurer une symbiose microbiote-hôte fonctionnelle. |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




