| Issue |
Med Sci (Paris)
Volume 39, Number 3, Mars 2023
Néphrologie pédiatrique : de grandes avancées et un futur rempli d’espoir
|
|
|---|---|---|
| Page(s) | 297 - 300 | |
| Section | Forum | |
| DOI | https://doi.org/10.1051/medsci/2023021 | |
| Published online | 21 March 2023 | |
Naissance d’un gène
Chroniques génomiques
The birth of a gene
Biologiste, généticien et immunologiste, président d’Aprogène (Association pour la promotion de la Génomique), 13007 Marseille, France
Abstract
The availability of an extensive set of vertebrate genome sequences, together with large-scale transcriptome studies, has allowed the discovery of numerous non-canonical ORFs (usually quite short) with evidence of transcription, translation and functional involvement. Orthologs for these ORFs can be detected in many vertebrates, and the time of appearance of a functional mini-gene can be ascertained. Some of these have appeared quite recently in evolution and have already very specific expression patterns in humans.
© 2023 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.

Le séquençage intégral du génome de nombreuses espèces, et la disponibilité de données d’expression pour la plupart d’entre elles, permettent aujourd’hui de réexaminer le contenu et les éventuelles fonctions de cet ADN « non codant », autrefois appelé Junk DNA ou « ADN poubelle », qui constitue environ 98 % de notre génome [1] (→).
(→) Voir la Chronique génomique de B. Jordan, m/s n° 6-7, juin-juillet 2020, page 675
Un article récemment paru [2] va plus loin, en montrant l’émergence dans ces régions, de gènes fonctionnels, parfois apparus tout récemment en termes évolutifs. C’est un élément important pour la compréhension de notre patrimoine génétique, qui s’avère plus complexe qu’il n’y paraît à première vue.
Des ORF « non canoniques », mais transcrits et traduits
La recherche de gènes au sein de la séquence d’un génome s’est tout d’abord focalisée sur les cadres de lecture (ORF, pour open rading frames) dits « canoniques », répondant à des critères assez rigoureux : présence du codon d’initiation ATG, cadre de lecture ouvert sur au moins 100 (parfois 150) codons (soit 300 à 450 nucléotides) avant un signal de terminaison TAG, plus, éventuellement, des indications de conservation à travers les espèces ou des traces de sélection, comme une proportion élevée de mutations silencieuses (ne modifiant pas la séquence protéique). Grâce à la séquence du génome humain et aux études systématiques qu’elle a permise – notamment le projet ENCODE [3] –, on a rapidement constaté que la transcription en ARN ne se limitait pas aux ORF canoniques et qu’elle concernait une grande partie du génome, bien au-delà des 2 % correspondant aux gènes tels que définis jusque-là. Une partie des transcrits mis en évidence correspond à de tous petits ORF codant potentiellement pour de petites protéines longues de quelques dizaines d’acides aminés, parfois trouvés à l’intérieur de longs ARN non codants (LncRNA, pour long non-coding RNAs) ; leur codon d’initiation de la traduction est assez fréquemment GTG, contrairement aux ORF canoniques. Dans quelques cas, des petites protéines ont pu être identifiées comme, par exemple, la myoréguline (46 acides aminés) qui intervient dans la régulation du muscle squelettique [4], montrant ainsi que certaines de ces « microprotéines » jouent un rôle biologique. Une recherche systématique a récemment été menée sur l’ensemble du génome humain [5]. Brièvement, les auteurs ont procédé en sélectionnant les micro-ORF traduits par profilage de ribosomes1 et en repérant ceux qui sont fonctionnels, par des expériences d’inactivation grâce au système CRISPR et à la mesure de fitness comme récemment décrit [6] (→).
(→) Voir la Chronique génomique de B. Jordan, m/s n° 10, octobre 2022, page 839
Près de 500 petits ORF non canoniques, transcrits et traduits en protéines fonctionnelles ont ainsi été repérés. Les microprotéines résultantes ont fait l’objet d’études supplémentaires : liaison avec d’autres molécules (protéines ou acides nucléiques), localisation cellulaire, présentation par les lymphocytes T de peptides dérivés, etc. On a ainsi la preuve qu’au moins une partie de ces micro-ORF donnent naissance à de petites protéines qui jouent un rôle dans le fonctionnement cellulaire. L’article analysé ici [2] s’intéresse à la manière dont ces micro-ORF fonctionnels apparaissent au cours de l’évolution.
Les micro-ORF à travers l’arbre des vertébrés
Ce dernier travail est le fruit d’une collaboration entre une équipe grecque et une équipe irlandaise, avec comme objectif, la recherche, dans la lignée humaine, de petites protéines apparues à partir de séquences précédemment identifiées comme non codantes. Il s’appuie très fortement sur le catalogue de micro-ORF fonctionnels établi précédemment [5], et sur les données de séquence et d’expression aujourd’hui disponibles pour de nombreuses espèces : les auteurs ont exploré l’ensemble des vertébrés en considérant cent espèces, de l’homme à la lamproie (le plus ancien vertébré, remontant à environ 400 millions d’années), en passant par le chinchilla, l’éléphant, l’alligator et le fugu (ou poisson-globe). À partir d’un micro-ORF fonctionnel chez l’homme (transcrit, traduit et nécessaire à la croissance optimale des cellules), ils ont recherché des orthologues2 de cette séquence dans le génome d’espèces de plus en plus éloignées ; la Figure 1 présente schématiquement deux cas possibles. Cette figure se limite aux primates les plus proches de nous, mais en réalité la recherche a porté sur un large ensemble de vertébrés dont la Figure 2 présente une partie.
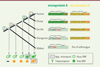 |
Figure 1. Schéma de l’apparition de microprotéines au cours de l’évolution des primates. Pour l’ORF A (en vert), la séquence est transcrite dans les cinq espèces, mais le cadre de lecture ouvert n’apparaît que chez le chimpanzé en raison d’une terminaison prématurée chez les quatre autres espèces ; pour l’ORF B (en jaune) la séquence est potentiellement codante dans quatre espèces (pas d’orthologue chez le gibbon), mais transcrite uniquement chez l’homme. La ligne ondulée au-dessus des séquences (qui en réalité couvrent plusieurs dizaines de nucléotides) figure la transcription (extrait partiel et modifié de la figure 1 de [2]). App. Microprot. : apparition de microprotéines. |
 |
Figure 2. Une partie de l’arbre phylogénique des vertébrés explorés dans cette étude. Il comporte en tout 100 espèces allant jusqu’à la lamproie, le plus ancien des vertébrés. L’époque de la divergence entre espèces (en millions d’années, mya) est indiquée à gauche (extrait partiel de la figure supplémentaire S3 de [2]). |
Les auteurs ont défini un jeu de 715 ORF fonctionnels chez l’homme (transcrits et traduits) d’après les données de Chen et al [5], en se restreignant aux séquences ne présentant pas de recouvrement avec des ORF canoniques et dont la transcription chez l’homme a été confirmée de manière indépendante [7]. Après analyse de leurs orthologues (éventuels) dans les 99 autres espèces, 155 d’entre eux s’avèrent « nouveaux », c’est-à-dire apparus (en tant que micro-ORF transcrits et traduits) au cours de l’évolution des vertébrés, pour la plupart bien avant l’apparition des primates. En fait, si les données de transcription sont disponibles pour toutes les espèces indiquées, l’étude fine de la traduction n’a vraiment été menée que chez l’homme [5]. Il n’est donc pas certain que tous les micro-ORF apparemment fonctionnels soient effectivement exprimés en protéines : leur date d’apparition peut donc être plus récente. Quoi qu’il en soit, les résultats indiquent que dix-neuf de ces nouvelles microprotéines sont apparues chez les primates, et deux dans l’espèce humaine, après la divergence homme/chimpanzé il y a près de sept millions d’années.
À quoi servent les (nouvelles) microprotéines ?
Le fait qu’une séquence soit transcrite et traduite ne prouve pas nécessairement qu’elle joue un rôle dans le fonctionnement de l’organisme, il pourrait s’agir d’une sorte de bruit de fond sans importance biologique. Mais avec le perfectionnement de méthodes dérivées du système CRISPR/Cas9, on dispose de techniques permettant d’inactiver de manière ciblée une série de gènes et de tester l’impact de chaque inactivation sur la viabilité au niveau cellulaire3. Cela a été testé sur deux lignées cellulaires, K562 (une lignée cellulaire tumorale), et des cellules souches pluripotentes induites (iPSC, pour induced pluripotent stem cells). Pour 44 des 155 microprotéines de novo révélées par cette étude, l’inactivation de l’ORF a un effet significatif sur la viabilité des cellules correspondantes. Cela ne signifie pas que les 111 autres soient inutiles, car leur inactivation aurait peut-être un effet dans d’autres types cellulaires. Mais, en tous cas, ces 44 microprotéines sont effectivement importantes pour le fonctionnement des cellules.
Un exemple : l’ORF CATP00001771233.1, long de 108 nucléotides et codant une microprotéine de 36 acides aminés. Il s’agit là d’un de ces « nouveaux gènes », transcrits et traduits chez l’homme ; on trouve des orthologues de ce gène dans de nombreuses espèces, dont la plus distante est l’armadillo (ou tatou), mais il n’est transcrit que chez l’homme et le chimpanzé : son apparition en tant que gène fonctionnel est donc récente du point de vue évolutif (Figure 2). Chez l’homme, cet ORF débute par un codon ATG, le codon d’initiation classique, mais chez les autres primates on trouve un codon GTG qui peut jouer le même rôle. Il est significatif de constater qu’aucun des algorithmes communément utilisés pour révéler la présence de gènes dans une séquence d’ADN ne le signale ; en comparaison avec les autres espèces, cet ORF n’est pas plus conservé que les zones d’ADN avoisinantes, et il ne présente pas non plus de signature de sélection4. La séquence protéique qu’il code n’a pas d’homologie significative avec les protéines répertoriées dans les protéomes de l’homme et de différents vertébrés. Il n’est donc pas étonnant que ce gène n’ait pas été détecté jusqu’ici ; pourtant, il est bel et bien transcrit, sa traduction est révélée par le profilage de ribosomes [5], et son inactivation inhibe la croissance dans les deux systèmes cellulaires testés (K562 et iPSC, voir plus haut). En recourant aux catalogues de séquences transcrites, comme GTEx (Genotype-Tissue Expression project)5, et à des données récemment publiées [7], on constate que cet ORF est principalement exprimé dans le tissu cardiaque, chez l’homme comme chez le chimpanzé. L’examen de résultats récents décrivant le translatome (ensemble des protéines produites) du tissu cardiaque humain confirme que la microprotéine codée est bien produite dans les cardiomyocytes [8]. On ignore encore sa fonction précise, qui fait sûrement l’objet d’études intensives, mais cet exemple montre comment un nouvel ORF fonctionnel peut être apparu récemment et avoir acquis un patron d’expression hautement spécifique. Ces recherches nous font bien assister à la naissance d’un gène……
Un nouveau domaine d’études
Ces travaux confirment ainsi l’existence de nombreux ORF non canoniques et jusqu’ici ignorés, mais bel et bien fonctionnels : au moins dix-neuf nouvelles microprotéines fonctionnelles apparues au niveau des primates, dont deux chez l’homme. L’article de Vakirlis et al. [2] montre aussi comment on peut suivre l’apparition de ces séquences, en tant que gènes exprimés, à travers l’arbre évolutif des vertébrés, soit sur plus de 400 millions d’années, et, en quelque sorte, assister à leur apparition. Tout ceci n’est possible que grâce à l’accumulation des séquences génomiques de très nombreux organismes : le Vertebrate Genomes Project [9] se fixe comme objectif le séquençage de tous les vertébrés (plus de 70 000 !) d’ici dix ans. La constitution de bases de données répertoriant les informations de transcription, et parfois de traduction, pour de plus en plus d’espèces, apporte des informations indispensables pour évaluer l’importance fonctionnelle des cadres de lecture détectés. Notre perception des mécanismes de l’évolution s’en trouve significativement modifiée. Les promoteurs du programme Génome humain n’imaginaient sans doute pas, il y a trente ans, que l’on irait aussi loin, et les détracteurs (qui furent nombreux) de ce projet « sans intérêt biologique » ont été superbement démentis – d’ailleurs, cela fait longtemps qu’ils ont changé d’avis tant les retombées du programme ont été évidentes dès les années 1990.
Liens d’intérêt
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Nous avons vu en détail un tel système dans une chronique récente [6].
Références
- Jordan B. Le Junk DNA n’est plus ce qu’il était. Med Sci (Paris) 2020; 36 : 675–7. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Vakirlis N, Vance Z, Duggan KM, McLysaght A. De novo birth of functional microproteins in the human lineage. Cell Rep 2022; 41 : 111808. [CrossRef] [PubMed] [Google Scholar]
- ENCODE Project Consortium, Snyder MP, Gingeras TR, Moore JE, et al. Perspectives on ENCODE. Nature 2020; 583 : 693–8. [CrossRef] [PubMed] [Google Scholar]
- Anderson DM, Anderson KM, Chang CL, et al. A micropeptide encoded by a putative long noncoding RNA regulates muscle performance. Cell 2015 ; 160 : 595–606. [CrossRef] [PubMed] [Google Scholar]
- Chen J, Brunner AD, Cogan JZ, et al. Pervasive functional translation of noncanonical human open reading frames. Science 2020; 367 : 1140–6. [Google Scholar]
- Jordan B. Scoop : les mutations « synonymes » ne sont pas neutres ! Med Sci (Paris) 2022; 38 : 839–41. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Hon CC, Ramilowski JA, Harshbarger J, et al. An atlas of human long non-coding RNAs with accurate 5’ ends. Nature 2017 ; 543 : 199–204. [CrossRef] [PubMed] [Google Scholar]
- Van Heesch S, Witte F, Schneider-Lunitz V, et al. The translational landscape of the human heart. Cell 2019 ; 178 : 242–260. [CrossRef] [PubMed] [Google Scholar]
- Rhie A, McCarthy SA, Fedrigo O, et al. Towards complete and error-free genome assemblies of all vertebrate species. Nature 2021; 592 : 737–46. [CrossRef] [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1. Schéma de l’apparition de microprotéines au cours de l’évolution des primates. Pour l’ORF A (en vert), la séquence est transcrite dans les cinq espèces, mais le cadre de lecture ouvert n’apparaît que chez le chimpanzé en raison d’une terminaison prématurée chez les quatre autres espèces ; pour l’ORF B (en jaune) la séquence est potentiellement codante dans quatre espèces (pas d’orthologue chez le gibbon), mais transcrite uniquement chez l’homme. La ligne ondulée au-dessus des séquences (qui en réalité couvrent plusieurs dizaines de nucléotides) figure la transcription (extrait partiel et modifié de la figure 1 de [2]). App. Microprot. : apparition de microprotéines. |
| Dans le texte | |
 |
Figure 2. Une partie de l’arbre phylogénique des vertébrés explorés dans cette étude. Il comporte en tout 100 espèces allant jusqu’à la lamproie, le plus ancien des vertébrés. L’époque de la divergence entre espèces (en millions d’années, mya) est indiquée à gauche (extrait partiel de la figure supplémentaire S3 de [2]). |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




