| Issue |
Med Sci (Paris)
Volume 38, Number 8-9, Août-Septembre 2022
Nos jeunes pousses ont du talent !
|
|
|---|---|---|
| Page(s) | 737 - 739 | |
| Section | Partenariat médecine/sciences - Écoles doctorales - Masters | |
| DOI | https://doi.org/10.1051/medsci/2022113 | |
| Published online | 12 September 2022 | |
L’analyse des mélanomes uvéaux primaires à l’aide de la technique de séquençage d’ARN de cellules uniques
Analysis of primary uveal melanomas using single cell RNA sequencing
Polytech Nice Sophia, Spécialité Génie biologique 5e année, Pharmacologie et Biotechnologies, Université Côte d’Azur, France
a eva.grandjean-closson@etu.univ-cotedazur.fr
b camille.heckmann@etu.univ-cotedazur.fr
c corentin.le-coz@etu.univ-cotedazur.fr
d isaline.louvet@etu.univ-cotedazur.fr
e matthieu.neri@etu.univ-cotedazur.fr
f Corine.Bertolotto@univ-cotedazur.fr
Les cancers sont hétérogènes. La variabilité entre différentes tumeurs d’un même patient ou entre plusieurs patients est appelée hétérogénéité inter-tumorale. Les tumeurs sont alors classées en sous-type oncogénique dont le pronostic et les thérapies peuvent être différents. On sait aussi maintenant que les tumeurs, individuellement, ne sont pas homogènes, c’est-à-dire qu’elles peuvent être composées de cellules génétiquement identiques mais ayant des profils moléculaires et phénotypiques différents. Cette hétérogénéité intratumorale peut résulter de surcroît d’une certaine plasticité des cellules cancéreuses en réponse à des changements de microenvironnement (hypoxie, carence en nutriments, etc.) ou d’exposition à un traitement et de leur interaction avec les cellules du stroma tumoral. Les cellules tumorales vont alors transiter dans un autre état cellulaire pour s’adapter au nouveau milieu pour survivre ou pour échapper à l’effet cytotoxique des médicaments, cette adaptation étant l’un des mécanismes pouvant conduire aux récidives.
Une meilleure compréhension des mécanismes de la plasticité des cellules et des différents états cellulaires composant une tumeur est un prérequis au développement de traitements efficaces et durables.
Hétérogénéité intratumorale du MU primaire
Parmi les patients pour lesquels le traitement du MU primaire a été efficace, 50 % vont développer des métastases majoritairement dans les 15 ans suivant l’intervention [1]. Ces observations suggèrent que des cellules se disséminent précocement à partir de la lésion primaire, restent probablement en dormance plusieurs mois voire plusieurs années, avant de se remettre à proliférer et à former des métastases [2, 3]. Cette hypothèse de dissémination précoce implique l’existence de différentes sous-populations cellulaires cancéreuses avec des comportements différents dans les MU primaires. En accord avec cette hypothèse, il est désormais bien établi que le MU peut être composé de cellules fusiformes associées à un bon pronostic, et/ou de cellules épithélioïdes associées à un mauvais pronostic. Le taux de survie à 15 ans est presque trois fois plus faible chez les patients atteints de tumeurs à cellules épithélioïdes que chez ceux atteints de tumeurs à cellules fusiformes. Ces morphologies cellulaires distinctes pourraient être associées à des signatures géniques différentes.
Identification de différents états transcriptionnels et de nouvelles signatures moléculaires dans le mélanome uvéal
Trois équipes ont récemment analysé le transcriptome de MU à l’échelle de la cellule unique et ont révélé l’existence d’une hétérogénéité intratumorale au sein de certains MU [4-7]. L’étude de Durante et al. a montré la complexité du microenvironnement tumoral, et s’est concentrée sur l’étude de l’infiltrat immunitaire immunosuppresseur, comprenant majoritairement des macrophages de type M2. L’infiltrat comprend aussi des lymphocytes T exprimant les points de contrôle immunitaire LAG-3 et, dans une moindre mesure, TIGIT [4]. Ces observations pourraient expliquer la résistance aux immunothérapies ciblant CTLA-4 et PD-1 des MU métastatiques.
Les équipes de Bertolotto et de Bakhoum se sont attachées à déterminer l’hétérogénéité des cellules tumorales et à caractériser les états transcriptionnels, montrant respectivement le rôle clé du facteur de transcription HES6 et du complexe d’ubiquitination PRC1 dans la progression métastatique du MU [4-7]. HES6 est un membre peu étudié de la voie NOTCH dont un rôle possible est la régulation de la motilité des cellules dans différents cancers [5].
Ces trois études reposent sur la technique de séquençage des ARN de cellules uniques ou scRNA-Seq (single cell RNA-seq), qui permet de connaître le transcriptome de chaque cellule composant un tissu.
Après biopsie (Figure 1A) et dissociation des tissus (Figure 1B), chaque cellule est isolée par microfluidique dans une gouttelette d’huile contenant des réactifs et une bille appelée GEM (gel bead in emulsion) comprenant un code barre, un identifiant moléculaire unique et une séquence polyT pour s’hybrider à la séquence polyA des ARNm (Figure 1C). Après lyse cellulaire et synthèse des ADN complémentaires (Figure 1D), les banques sont préparées (Figure 1E) et séquencées (Figure 1F). Les fragments de séquençage obtenus sont alors alignés par rapport à un génome de référence pour déterminer le profil d’expression génique de chaque cellule (Figure 1G, H) [8]. Cette approche apporte aussi des informations sur le profil d’expression génique des cellules pouvant être minoritaires dans un échantillon tissulaire et qui pourraient échapper à des analyses globales, ouvrant ainsi la voie à l’identification de nouvelles cibles thérapeutiques d’intérêt pour élaborer de nouveaux traitements [9, 10].
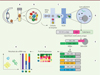 |
Figure 1. Technique de séquençage des ARN de cellules uniques. A. Dissection du tissu. B. Dissociation du tissu pour préparer une suspension de cellules. C. Isolement des cellules dans des gouttelettes d’huile. D. Lyse cellulaire. E. Hybridation des ARNm aux billes GEM, RT-PCR, et préparation des banques par ajout des adaptateurs. F. Séquençage à haut débit. G. Obtention du profil d’expression des gènes des cellules uniques. H.. Identification des clusters et déduction des types cellulaires. ARNm: ARN messager ; GEM : gel bead in emulsion ; RT-PCR : reverse transcriptase polymerase chain reaction. |
Ces analyses ont permis à l’équipe de Bertolotto de mettre en évidence une nouvelle signature moléculaire (appelée signature PC1) associée à un mauvais pronostic des MU primaires [6]. Soulignons que plusieurs signatures moléculaires ont déjà été identifiées à partir d’une analyse transcriptomique globale de MU primaires [11]. L’avantage de la signature PC1, qui comprend HES6, par rapport aux autres, est qu’elle prend en compte l’hétérogénéité intratumorale. De plus, elle permet de détecter des cellules individuelles exprimant des gènes de mauvais pronostic dans des tumeurs diagnostiquées de bon pronostic par les classifications usuelles (AJCC et cytogénétique). Ainsi, ces cellules, qui pourraient représenter un réservoir de cellules invasives conduisant au développement des métastases et aux récidives, sont difficilement détectables par une analyse globale du fait de leur faible nombre, mais peuvent être décelées grâce à l’analyse en cellules uniques et à la signature PC1.
Mise en évidence d’une hétérogénéité intratumorale génomique du MU
Les variations du nombre de copies peuvent également être déduites des données de séquençage en cellules uniques. Ces données ont révélé des déséquilibres chromosomiques caractéristiques du MU, comme la perte du chromosome 3 et un gain du chromosome 8q. Ce type d’analyse a aussi révélé que certains MU présentent plusieurs clones tumoraux caractérisés par des nombres de copies de gènes différents dans plusieurs régions chromosomiques. Il faut noter que les auteurs ont observé que l’hétérogénéité transcriptomique et génomique ne se chevauchent pas dans certaines tumeurs, suggérant que l’hétérogénéité transcriptomique n’est pas uniquement due à l’hétérogénéité génomique. L’origine de l’hétérogénéité intratumorale transcriptomique dans les MU reste en partie à déterminer. Remarquablement, des altérations cryptiques mineures non détectables par des analyses globales, de type hybridation génomique comparative, ont pu être mises en évidence à partir des données de séquençage. Par exemple, dans une des tumeurs primaires, 2 à 5 % des cellules présentaient une monosomie du chromosome 3. Cette information pourrait être importante pour le suivi du patient, sachant que la monosomie est associée à un risque métastatique élevé.
Impact de ces travaux sur les stratégies thérapeutiques
Ces différentes études [4, 7] ont permis d’établir un lien entre certaines sous-populations cellulaires et des mécanismes comme la dissémination des cellules cancéreuses (métastases) et l’induction par la tumeur d’une inflammation et de réponses immunitaires [4-7]. De plus, un état transcriptionnel particulier, associé au processus invasif et à la signature de mauvais pronostic PC1 et contrôlé en partie par le facteur de transcription HES6 et par ses gènes cibles éventuels (réseau génique appelé régulon), a été identifié. La description de ce profil particulier ouvre la voie au développement de nouvelles stratégies thérapeutiques [6, 12] (→).
(→) Voir la Nouvelle de E. Papasotiriou et al., page 733 de ce numéro
En conclusion, l’analyse des MU à l’échelle de la cellule unique a révélé une hétérogénéité intratumorale qui est considérée comme un obstacle majeur à l’établissement d’un pronostic fiable et à des traitements efficaces et durables. La caractérisation des différentes sous-populations cellulaires qui constituent les MU est un prérequis nécessaire pour surmonter cet obstacle. Des informations importantes permettant de mieux comprendre deux aspects critiques de la biologie du MU, la dissémination métastatique et l’échappement immunitaire, ont déjà été obtenues. De plus, ces études révèlent ainsi de nouvelles opportunités thérapeutiques, comme par exemple des immunothérapies fondées sur l’utilisation de molécules ciblant LAG-3 ou TIGIT ou des thérapies ciblées impliquant la signalisation d’HES6 [13] (→).
(→) Voir la Nouvelle de L. Nucera et al., page 740 de ce numéro
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Abréviations
AJCC : American Joint Committee on Cancer
CTLA-4 : Cytotoxic T lymphocyte antigen 4
HES6 : Hairy/Enhancer of Split (Hes) 6
LAG-3 : Lymphocyte Activation Gene-3
MITF : Facteur de transcription lié à la microphtalmie
PD-1 : Programmed cell death protein 1
PRC1 : Polycomb Repressive Complex 1
RELB : Facteur de transcription RELB
ROC : Receiver Operating Characteristic
SOX : Facteur de transcription SOX (SRY-related HMG box)
TIGIT : T-cell immunoreceptor with Ig and ITIM domains
Références
- Kujala E, Mäkitie T, Kivelä T. Very long-term prognosis of patients with malignant uveal melanoma. Invest Ophthalmol Vis Sci 2003 ; 44 : 4651–4659. [CrossRef] [PubMed] [Google Scholar]
- Eide N, Hoifødt HK, Nesland JM et al. Disseminated tumour cells in bone marrow of patients with uveal melanoma. Acta Ophthalmol 2013 ; 91 : 343–348. [CrossRef] [PubMed] [Google Scholar]
- Eide N, Faye RS, Høifødt HK et al. The results of stricter inclusion criteria in an immunomagnetic detection study of micrometastatic cells in bone marrow of uveal melanoma patients - Relevance for dormancy. Pathol Oncol Res 2019 ; 25 : 255–262. [CrossRef] [PubMed] [Google Scholar]
- Durante MA, Rodriguez DA, Kurtenbach S, et al. Single-cell analysis reveals new evolutionary complexity in uveal melanoma. Nat Commun 2020; 11 : 496. [CrossRef] [PubMed] [Google Scholar]
- Krossa I, Strub T, Martel A, Nahon-Esteve S, Lassalle S, Hofman P, Baillif S, Ballotti R, Bertolotto C. Recent advances in understanding the role of HES6 in cancers. Theranostics. 2022; 12 : 4374–85. [CrossRef] [PubMed] [Google Scholar]
- Pandiani C, Strub T, Nottet N, et al. Single-cell RNA sequencing reveals intratumoral heterogeneity in primary uveal melanomas and identifies HES6 as a driver of the metastatic disease. Cell Death Differ 2021; 28 : 1990–2000. [CrossRef] [PubMed] [Google Scholar]
- Bakhoum MF, Francis JH, Agustinus A, et al. Loss of polycomb repressive complex 1 activity and chromosomal instability drive uveal melanoma progression. Nat Commun 2021; 12 : 5402. [CrossRef] [PubMed] [Google Scholar]
- Schutz S. Séquençage des ARNm sur cellules uniques. bioinformatique génétique médecine. [Google Scholar]
- Foulon S. Développement du séquençage ARN ciblé sur cellules uniques en microfluidique de gouttes et applications. Thèse de doctorat, Université Paris sciences et lettres. 2019. [Google Scholar]
- Shintaku H, Nishikii H, Marshall LA et al. On-chip separation and analysis of RNA and DNA from single cells. Anal Chem 2014 ; 86 : 1953–1957. [CrossRef] [PubMed] [Google Scholar]
- Lamas NJ, Martel A, Nahon-Estève S, et al. Prognostic biomarkers in uveal melanoma: The status quo, recent advances and future directions. Cancers 2022; 14 : 96. [Google Scholar]
- Papasotiriou E, Chessel E, Costa C, et al. Quels traitements pour le mélanome uvéal ? Med Sci (Paris) 2022; 38 : 733–6. [Google Scholar]
- Nucera L, Perrée E, Pujas P, et al. Rôle du facteur de transcription HES6 dans le mélanome uvéal. Med Sci (Paris) 2022; 38 : 740–2. [Google Scholar]
© 2022 médecine/sciences – Inserm
Liste des figures
 |
Figure 1. Technique de séquençage des ARN de cellules uniques. A. Dissection du tissu. B. Dissociation du tissu pour préparer une suspension de cellules. C. Isolement des cellules dans des gouttelettes d’huile. D. Lyse cellulaire. E. Hybridation des ARNm aux billes GEM, RT-PCR, et préparation des banques par ajout des adaptateurs. F. Séquençage à haut débit. G. Obtention du profil d’expression des gènes des cellules uniques. H.. Identification des clusters et déduction des types cellulaires. ARNm: ARN messager ; GEM : gel bead in emulsion ; RT-PCR : reverse transcriptase polymerase chain reaction. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




