| Issue |
Med Sci (Paris)
Volume 38, Number 8-9, Août-Septembre 2022
|
|
|---|---|---|
| Page(s) | 723 - 725 | |
| Section | Forum | |
| DOI | https://doi.org/10.1051/medsci/2022098 | |
| Published online | 12 September 2022 | |
Mutations somatiques et longévité
Chroniques génomiques
Somatic mutations and longevity
Biologiste, généticien et immunologiste, Président d’Aprogène (Association pour la promotion de la génomique), 13007 Marseille, France
Abstract
New approaches allow precise measurement of somatic mutations in tissues. Applied to a diverse set of mammals, these methods show that somatic mutation load increases with age (as expected) but reaches similar levels near the end of life for animals with extremely different longevity. This is an important result that has many repercussions on concepts of aging and of evolution of longevity.
© 2022 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.

Au cours de deux chroniques récentes [1, 2] (→), nous avons exploré la relation entre la longévité d’une espèce et l’efficacité des mécanismes de réparation de l’ADN dont elle dispose. La comparaison des génomes de différentes espèces de Sébastes, poissons dont la longévité varie de 20 à 200 ans selon l’espèce, mettait en évidence une forte pression de sélection positive sur les gènes de réparation de l’ADN chez les poissons de plus grande longévité [1] ; la mesure directe de l’efficacité de ces mécanismes chez des mammifères de durées de vie très différentes suggérait une relation directe entre cette efficacité et la longévité maximale observée [2]. Ces données sont instructives et vont dans le sens d’une corrélation entre la réparation de l’ADN et la longévité, ce qui est cohérent avec l’idée que l’accumulation de mutations somatiques est un des principaux mécanismes du vieillissement [3, 4]. Pour mieux étayer cette hypothèse, il restait néanmoins nécessaire d’obtenir une évaluation directe du nombre de mutations somatiques accumulées, en fonction de l’âge chez différentes espèces. Or, malgré les grands progrès des techniques du séquençage de l’ADN, la détection de mutations apparaissant dans les cellules somatiques d’un individu reste difficile à réaliser : comme elles surviennent au hasard, chaque mutation n’est présente que dans une seule cellule (plus, éventuellement, ses quelques descendantes) et est donc indétectable lors du séquençage d’un fragment de tissu, en raison de sa très forte dilution dans l’ADN de l’échantillon. On peut certes résoudre ce problème en procédant au séquençage de cellules individuelles, mais cette approche est très délicate du point de vue technique, d’autant plus qu’il faut une séquence de très bonne qualité pour repérer, à coup sûr, des mutations somatiques. Cette approche a été mise en œuvre pour la deuxième étude rappelée ci-dessus [2], mais à petite échelle en raison de sa difficulté et de son coût – ce qui explique sans doute la grande dispersion des résultats obtenus. L’article qui fait l’objet de cette chronique [5] contourne le problème de manière astucieuse et est ainsi en mesure de fournir des résultats très cohérents – et fort instructifs.
(→) Voir les Chroniques génomiques de B. Jordan, m/s n° 2, février 2022, page 218 et m/s n° 3, mars 2022, page 317
Les cryptes du côlon (intestinal crypts)
Les cryptes du côlon sont de petites structures (Figure 1) tapissant l’épithélium du côlon. Elles peuvent être isolées par microdissection au laser ; l’intérêt de cibler ces cryptes est que l’ensemble de chacune descend d’une seule cellule souche intestinale, et constitue un clone récemment dérivé de cette cellule originale1. Le séquençage de l’ADN d’une crypte équivaut donc à celui d’une seule cellule – mais il est techniquement bien plus aisé. Pour le repérage des mutations somatiques, cette séquence est comparée à celle d’un échantillon de peau du même individu, dans laquelle les mutations somatiques sont invisibles car présentes chacune à une très faible fréquence. On a ainsi une possibilité « non acrobatique » de mesure du nombre de mutations somatiques accumulées dans un tissu de l’individu étudié. Les auteurs ont ainsi disséqué et séquencé, à partir de prélèvements obtenus lors d’autopsie, 208 cryptes intestinales provenant de 56 individus appartenant à 16 espèces allant de la souris à l’homme en passant par le chien, le lion, la girafe et le cheval, dont la longévité2 s’étale de 2 à 80 ans.
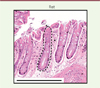 |
Figure 1. Image histologique de côlon de rat indiquant une crypte individuelle (entourée de pointillés). La barre noire en bas de l’image correspond à 250 μm. Extrait partiel de la figure 1 de [5] (© licence Creative commons). |
Des résultats très nets
Selon les individus et les échantillons, le nombre de mutations somatiques détectées varie de quelques centaines à quelques milliers. Et, comme le montre la Figure 2, ce nombre augmente de manière linéaire avec l’âge de l’individu. Les résultats sont très cohérents : pour l’homme, 28 cryptes provenant de 8 individus ont été séquencées ; pour la souris, 43 échantillons provenant de 12 animaux. Et l’on voit que pour un âge donné, les points sont très proches dans chaque espèce. Si maintenant on compare l’homme et la souris, l’allure des diagrammes est similaire, mais les échelles sont très différentes : chez l’homme, on atteint un chiffre de 3 000 mutations par génome, vers l’âge de 70 ans3 (→), alors que, pour la souris, une valeur de 1 500 est relevée dès l’âge de 2 ans – soit au voisinage de la limite de longévité. En d’autres termes, les deux espèces accumulent des mutations somatiques à un rythme inversement corrélé à leur longévité, ce qui entraîne une charge de mutations somatiques équivalente vers la fin de leur existence.
 |
Figure 2. Nombre de mutations somatiques accumulées par génome, en fonction de l’âge (en années), pour l’homme (à gauche) et la souris (à droite). Les données sont très cohérentes et montrent une charge de mutations comparable vers la fin de la vie. Extrait partiel de la figure 1 de [5] (© licence Creative commons). |
(→) Voir la Chronique génomique de B. Jordan, m/s n° 8-9, août-septembre 2021, page 802
Les autres animaux étudiés présentent eux aussi cette relation inverse, comme résumé dans la Figure 3 qui montre le nombre de mutations somatiques par année et par génome en fonction de la longévité de l’espèce considérée. Par exemple, le taux de mutations apparaissant par an est d’environ 50 chez l’homme contre près de 1 000 chez la souris. La courbe obtenue est très régulière, et notre espèce se situe à l’extrémité de la distribution, indiquant un faible taux de mutations somatiques sans doute lié à des mécanismes de réparation particulièrement efficaces et probablement responsables de notre longévité.
 |
Figure 3. Taux de mutations somatiques par année chez toutes les espèces étudiées, en fonction de leur longévité. Extrait partiel de la figure 3 de [5] (© licence Creative commons). |
In fine, les résultats rapportés dans cet article vont dans le même sens que ceux qui suggèrent une plus grande efficacité des mécanismes de réparation de l’ADN chez les espèces à grande longévité, qu’il s’agisse de poissons [1] ou de mammifères [2]. De plus, ils apportent des informations précises sur la vitesse d’accumulation des mutations somatiques au cours de l’existence, et démontrent une relation inverse entre cette vitesse et la longévité. Ils confirment ainsi l’hypothèse d’un rôle important de ces mutations dans le vieillissement [4]. Reste bien sûr à comprendre comment la sélection naturelle a pu agir sur les mécanismes de réparation de l’ADN pour aboutir à ce résultat…
Des informations précises
À côté de ce résultat important, cette étude apporte un ensemble de données factuelles intéressantes. La majeure partie des mutations somatiques repérées (80 % environ) sont des substitutions, le reste des mutations étant constitué de petites insertions ou délétions, et les réarrangements de plus grande ampleur étant très rares. Cela est valable pour les différentes espèces étudiées ; de même, les « signatures de mutation » [7]4, repérables dans la gamme de mutations observées chez l’homme, se retrouvent peu ou prou chez les autres espèces, ce qui suggère que les mécanismes de mutagenèse sont similaires. En possession de ces données précises, on peut aussi essayer de revisiter une question déjà ancienne, le « paradoxe de Peto », formulé dès 1977 [8]. En voici l’essentiel : compte tenu de ce que l’on sait de la carcinogenèse, on s’attendrait a priori à ce que le risque de cancer chez un organisme soit proportionnel au nombre de cellules susceptibles de subir une transformation cancéreuse, et donc augmente fortement avec la masse de l’animal considéré. Ce n’est visiblement pas le cas : le cancer n’est pas plus fréquent chez l’éléphant que chez la souris, au contraire. Une étude récente portant sur plus de cent mille individus de 191 espèces a montré de manière convaincante que le risque de cancer est indépendant tant de la masse corporelle que de la longévité de l’espèce [9]. Dans le travail rapporté ici, le taux de mutations somatiques s’avère largement indépendant de la masse de l’animal : la girafe et le rat-taupe nu (naked mole rat), par exemple – de longévités équivalentes -, accumulent chacun une centaine de mutations somatiques par année (Figure 3), alors que le rapport de leurs masses corporelles est de plus de 20 000. Pour rendre compte du paradoxe de Peto, il faudrait que le taux de mutations chez la girafe soit très inférieur à celui mesuré chez le rat-taupe nu, contrairement à ce qui est observé. On doit donc envisager d’autres mécanismes pour expliquer la relative protection des espèces de grande taille : chez l’éléphant par exemple, l’existence de vingt exemplaires du gène suppresseur de tumeurs TP53 (tumour protein 53) est très certainement l’un des éléments qui expliquent la faible incidence de cancer, deux à trois fois inférieure à celle observée chez l’homme qui ne dispose que d’un seul exemplaire de ce gène [10, 11] (→).
(→) Voir le Repères de J.C. Weill, m/s n° 11, novembre 2017, page 997
L’éléphant ne figure malheureusement pas dans l’étude rapportée ici [5], mais on peut parier, en examinant la Figure 3, que son taux de mutations somatiques devrait être de l’ordre de 70 par année pour une longévité de 50 ans.
Des réponses… et des questions
Le très joli travail de Cagan et al. [5] apporte ainsi des informations précises sur un paramètre important et jusqu’ici difficilement accessible. La relation inverse observée entre taux de mutations somatiques et longévité renforce l’hypothèse d’une relation causale entre l’accumulation de mutations et le vieillissement, et il est frappant de constater que des animaux de longévités très différentes ont, vers la fin de leur vie, accumulé un nombre comparable de mutations somatiques. Cela pose au moins deux questions. Tout d’abord, quels sont les processus responsables de ces différences ? La similitude des profils de mutation observés suggère que les mécanismes de mutations somatiques (et de réparation de l’ADN) sont similaires chez ces différentes espèces. Mais alors, pourquoi agissent-ils de manière aussi différente ? Et il faudra ensuite comprendre comment l’évolution a pu agir sur ces paramètres pour aboutir à la situation actuelle, marquée par une longévité accrue, sélectionnée chez certaines espèces et pas chez d’autres, une question posée depuis longtemps [3] mais que les résultats récents permettent d’aborder sous un nouvel angle.
Liens d’intérêt
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Notons que ce chiffre est très supérieur à celui des mutations germinales, évalué à moins de 100 par génération chez l’homme [6].
Références
- Jordan B. La longévité des sébastes. Med Sci (Paris) 2022; 38 : 218–21. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Jordan B. Réparation de l’ADN et longévité : les rongeurs et nous. Med Sci (Paris) 2022; 38 : 317–9. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Kirkwood TB Holliday R. The evolution of ageing and longevity. Proc R Soc Lond B Biol Sci 1979 ; 205 : 531–546. [CrossRef] [PubMed] [Google Scholar]
- Morley AA. The somatic mutation theory of ageing. Mutat Res 1995 ; 338 : 19–23. [CrossRef] [PubMed] [Google Scholar]
- Cagan A, Baez-Ortega A, Brzozowska N, et al. Somatic mutation rates scale with lifespan across mammals. Nature 2022; 604 : 517–24. [CrossRef] [PubMed] [Google Scholar]
- Jordan B. Les gènes des enfants de Tchernobyl. Med Sci (Paris) 2021; 37 : 802–5. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Alexandrov LB, Kim J, Haradhvala NJ, et al. The repertoire of mutational signatures in human cancer. Nature 2020; 578 : 94–101. [CrossRef] [PubMed] [Google Scholar]
- Peto R. In Origins of Human Cancer. Hiatt H et al., eds. Cold Spring Harbor Laboratory 1977; 45 : 1403–28. [Google Scholar]
- Vincze, O. Colchero F, Lemaître JF, et al. Cancer risk across mammals. Nature 2022; 601 : 263–7. [CrossRef] [PubMed] [Google Scholar]
- Abegglen LM Caulin AF Chan A, et al. Potential Mechanisms for Cancer Resistance in Elephants and Comparative Cellular Response to DNA Damage in Humans. JAMA 2015 ; 314 : 1850–1860. [CrossRef] [PubMed] [Google Scholar]
- Weill JC. Pourquoi n’avoir que deux copies du gène P53 ?. Med Sci (Paris) 2017 ; 33 : 997–999. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1. Image histologique de côlon de rat indiquant une crypte individuelle (entourée de pointillés). La barre noire en bas de l’image correspond à 250 μm. Extrait partiel de la figure 1 de [5] (© licence Creative commons). |
| Dans le texte | |
 |
Figure 2. Nombre de mutations somatiques accumulées par génome, en fonction de l’âge (en années), pour l’homme (à gauche) et la souris (à droite). Les données sont très cohérentes et montrent une charge de mutations comparable vers la fin de la vie. Extrait partiel de la figure 1 de [5] (© licence Creative commons). |
| Dans le texte | |
 |
Figure 3. Taux de mutations somatiques par année chez toutes les espèces étudiées, en fonction de leur longévité. Extrait partiel de la figure 3 de [5] (© licence Creative commons). |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




