| Issue |
Med Sci (Paris)
Volume 36, Number 4, Avril 2020
|
|
|---|---|---|
| Page(s) | 421 - 423 | |
| Section | Forum | |
| DOI | https://doi.org/10.1051/medsci/2020055 | |
| Published online | 01 May 2020 | |
Néandertal et Afrique, le retour
Chroniques génomiques
Neandertal DNA in Africans
UMR 7268 ADÉS, Aix-Marseille, Université /EFS/CNRS ; CoReBio PACA, case 901, Parc scientifique de Luminy, 13288 Marseille Cedex 09, France
Abstract
Sophisticated analyses of current human populations compared to a high-coverage Neandertal genome sequence indicate that, contrary to the previous consensus, African genomes carry a small but significant amount of Neandertal-specific DNA. This indicates back-migration into Africa of modern humans (carrying some Neandertal sequences) and underlines the complexity of ancient human migrations.
© 2020 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.

De l’ADN de Néandertal ?
Il y a dix ans, la première séquence d’un ADN d’homme de Néandertal [1] suggérait que des échanges génétiques avaient eu lieu entre des Néandertaliens et des hommes modernes (Homo sapiens, autrefois appelé homme de Cro-Magnon, et quasiment identique aux populations humaines actuelles). C’était une surprise : la question était débattue depuis plusieurs décennies, mais le consensus allait plutôt vers une absence d’interactions de notre espèce avec cette branche des hominidés, disparue depuis plus de trente mille ans [10] (→).
(→) Voir le Forum de P. Charlier et al., m/s n° 8-9, août-septembre 2018, page 745
Ces premiers résultats furent bientôt confirmés par des séquences de meilleure qualité, notamment celle d’une Néandertalienne provenant des monts de l’Altaï en Sibérie [2]. Au fil des années, les techniques d’analyse de l’ADN ancien (extrait d’ossements vieux de milliers ou dizaines de milliers d’années) allaient être encore perfectionnées et permettre de nombreuses études qui ont révolutionné notre connaissance de l’histoire ancienne de l’humanité, montrant l’importance des déplacements de populations et indiquant notamment que les populations actuelles en un lieu donné ne sont généralement pas les descendantes de celles qui occupaient le même espace il y a dix ou vingt mille ans [3, 4]. Au passage, on découvrait l’existence d’une autre lignée d’humains archaïques, les Denisoviens (du nom de la grotte en Sibérie où furent trouvés leurs ossements), et l’on était amené à soupçonner l’existence d’autre lignées archaïques encore à découvrir [4]. Mais la vision des échanges entre humains1 et Néandertaliens restait pour l’essentiel la même : ces évènements avaient eu lieu après la sortie d’Afrique du groupe d’humains dont descend l’ensemble des populations non africaines actuelles [11] (→), il y a 70 à 80 000 ans, et bien sûr avant la disparition des derniers Néandertaliens, il y a 30 000 ans environ. La trace de ces échanges, des séquences d’ADN de l’homme de Néandertal représentant une cinquantaine de mégabases (Mb, millions de bases d’ADN), soit deux pour cent du génome, n’était donc présente que chez les populations non africaines (Européens, Asiatiques, Amérindiens). On trouvait d’ailleurs un peu plus d’ADN de Néandertal chez les Asiatiques que chez les Européens, ce qui semblait difficile à expliquer.
(→) Voir la Synthèse de J.P. Henry, m/s n° 1, janvier 2019, page 39
Une nouvelle surprise
Un ensemble d’analyses toutes récentes, présentées dans un article de la revue Cell en février 2020 [5], vient modifier ces conclusions et montre la présence d’ADN néandertalien dans les génomes de populations africaines. La méthode d’analyse mise au point par ces auteurs exploite 2 504 séquences d’ADN complètes d’individus modernes répartis en 26 groupes géographiques (dont 504 Africains), obtenues dans le cadre du 1 000 Genomes Project [6], et les compare à la meilleure séquence d’ADN néandertalien existante, déjà signalée plus haut [2]. Contrairement aux méthodes employées jusqu’ici, celle-ci ne fait pas intervenir un panel de séquences africaines : elle est appelée IBDmix (identity by descent) et utilise des techniques statistiques sophistiquées pour éliminer les segments identiques (entre humains et Néandertaliens) mais provenant d’ancêtres communs à ces deux lignées2 et ne conserver que celles qui résultent d’une hybridation survenue postérieurement. Du coup, les séquences de type Néandertal chez les Africains, que les techniques précédentes tendaient à éliminer du fait de l’emploi d’un panel de séquences africaines, apparaissent clairement. Ces chercheurs ont ainsi identifié une proportion notable d’ADN néandertalien chez les Africains : de 16 à 18 Mb par individu, soit environ 0,5 % du génome. C’est nettement moins que la cinquantaine de mégabases trouvée chez les Européens ou les Asiatiques, mais ce n’est pas négligeable… Notons au passage que l’excès d’ADN de Néandertal précédemment observé chez les Asiatiques (environ 20 %) disparaît presque avec cette nouvelle méthode d’analyse puisqu’elle évalue les contributions néandertaliennes à 51 et 55 Mb pour Européens et Asiatiques, respectivement. La Figure 1 montre les résultats pour différentes populations ; on notera que la proportion d’ADN de l’homme de Néandertal est peu variable entre les quatre populations non-africaines étudiées (EAS, SAS, EUR, AMR, voir légende de la Figure). Elle varie également peu entre les cinq populations africaines étudiées (qui vont du Kenya au Nigéria, voir la légende).
 |
Figure 1. ADN de l’homme de Néandertal dans différentes populations (échelle verticale, en mégabases par individu). La tache indiquée pour chaque population (appelée violin plot) représente la distribution observée et ses valeurs extrêmes. À droite, la population africaine est éclatée en cinq groupes. EAS, East Asian ; SAS, South Asian ; EUR, European ; AMR, AdMixed Americans (Mexique, Porto Rico, Colombie, Pérou) ; ACB/ASW, African Caribbean & Afro-American ; AFR, African. Pour les groupes Africains : LWK, Luhya, Kenya ; GWD, Gambien, Gambie de l’Ouest ; MSL, Mende, Sierra Leone ; YRI, Yoruba, Nigeria ; ESN, Esan, Nigeria. Voir : https:// www.internationalgenome.org/faq/which-populations-are-part-your-study/ (extrait partiel et modifié de la figure 2 de [5]). |
Comme le montre l’extension des violin plots 3 de la Figure 1 , la quantité d’ADN néandertalien portée par chaque individu varie, entre 40 et 60 Mb par exemple chez les Européens. L’extension vers le haut du diagramme pour les populations afro-américaines (ACB/ASW) correspond au fait que, parmi ces populations, quelques individus ont une ascendance majoritairement européenne même s’ils sont socialement « Noirs » [7]. Les cinq populations africaines étudiées, quant à elles, montrent à peu près la même quantité d’ADN néandertalien, entre 15 et 20 MB par individu soit environ 0,5 % du génome contre 2 % pour les différentes populations non-africaines. Si l’on évalue maintenant la nature des séquences présentes chez chaque individu, on constate qu’elles varient de l’un à l’autre mais présentent un certain recouvrement. On peut alors comparer l’ensemble de l’ADN néandertalien représenté chez les non-Africains et le comparer à celui trouvé chez les Africains. La Figure 2 montre le résultat de cette comparaison et indique que la grande majorité des séquences de Néandertal « africaines » sont aussi trouvées chez les non-Africains.
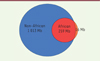 |
Figure 2. Ce diagramme indique l’ensemble des séquences néandertaliennes non redondantes trouvées chez les non-Africains (1 013 Mb, en bleu) et chez les Africains (259+16 Mb, orange). La quasi-totalité des séquences de Neandertal africaines sont aussi présentes chez les non-Africains (extrait partiel et modifié de la figure 2 de [5]). |
Un retour très probable
La présence de ces séquences de type Néandertal dans l’ADN de l’homme moderne africain peut a priori être la conséquence de différents évènements : une hybridation directe entre Néandertaliens et Africains suite à la présence en Afrique d’hommes de Néandertal (ce qui serait en contradiction avec toutes les données archéologiques et paléontologiques existantes), un retour en Afrique d’Européens après qu’ils ont acquis des segments d’ADN néandertaliens dans leur génome (donc il y a quelques dizaines de milliers d’années au plus), ou encore une hybridation très ancienne entre l’homme de Néandertal et l’homme moderne, lors d’une première sortie d’Afrique bien antérieure à celle qui a eu lieu il y a 70 à 80 000 ans. Dans ce dernier cas, des segments d’ADN « moderne » auraient pu être intégrés dans le génome de l’homme de Néandertal et apparaître ultérieurement dans les comparaisons entre ADN de l’homme de Néandertal et celui des différents groupes humains actuels. Différentes simulations effectuées par les auteurs montrent que la seconde hypothèse est de loin la plus probable : ce serait donc le retour en Afrique d’hommes modernes issus des populations qui en étaient sorties précédemment, et qui s’étaient croisés avec les hommes de Néandertal, qui aurait introduit dans le génome des Africains ces 0,5 % de séquences néandertaliennes. Une fois de plus, on voit que l’histoire de l’humanité, comme la révèlent les études sur l’ADN ancien, est bien plus complexe et plus mouvante qu’on ne l’avait supposé. Cela continue d’ailleurs, puisque l’on semble trouver maintenant dans l’ADN humain la trace de mélanges avec des hominidés très anciens remontant à plus d’un million d’années… [8]
Des implications fonctionnelles
Si l’on répertorie les régions d’ADN néandertalien aujourd’hui présentes dans le génome des Africains, on trouve que certaines d’entre elles (une douzaine) sont présentes de manière très fréquente. Leur examen indique qu’elles contiennent des gènes impliqués dans les fonctions immunitaires ainsi que dans la sensibilité (ou la résistance) aux radiations ultraviolettes [12] (→).
(→) Voir la Synthèse de M. Deschamps et L. Quintana-Murci, m/s n° 12, décembre 2016, page 1079
Inversement, on trouve des « déserts archaïques », des régions du génome qui ne contiennent jamais de segments d’ADN néandertalien – l’un de ces déserts contient le gène FOXP2 (Forkhead-box P2) [9], impliqué dans le langage. Le fait que ce gène soit situé dans un « désert » implique que tous les Africains portent la version « Homo sapiens » de ce gène, et non la version « Néandertal ». On voit donc que ces analyses peuvent révéler les traces des pressions sélectives auxquelles ont été soumises ces populations, et enrichir ainsi considérablement les données de l’anthropologie.
Notre image de l’histoire humaine continue donc à se compliquer au fil des résultats qu’apportent les études sur l’ADN ancien, rendus possibles par l’incroyable progrès des techniques de séquençage et la puissance accrue des méthodes d’analyse statistique. Il apparaît aujourd’hui que notre espèce, bien plus mobile qu’on ne le supposait, s’est mélangée au fil de nombreuses migrations avec différentes souches humaines archaïques aujourd’hui disparues [10], et que sa diversité actuelle a des racines très anciennes dont nous commençons tout juste à démêler les multiples embranchements.
Liens d’intérêt
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- Green RE, Krause J, Briggs AW, et al. A draft sequence of the Neandertal genome. Science 2010 ; 328 : 710–722. [Google Scholar]
- Prüfer K, Racimo F, Patterson N, et al. The complete genome sequence of a Neanderthal from the Altai mountains. Nature 2014 ; 505 : 43–49. [Google Scholar]
- Slatkin M, Racimo F. Ancient DNA and human history. Proc Natl Acad Sci USA 2016 ; 113 : 6380–6387. [CrossRef] [Google Scholar]
- Reich D.. Who we are and how we got here: ancient DNA and the new science of the human past ; Oxford : Oxford University Press, 2018. [Google Scholar]
- Chen L, Wolf AB, Fu W, et al. Identifying and interpreting apparent Neanderthal ancestry in African individuals. Cell 2020; pii: S0092–8674(20)30059–3. [Google Scholar]
- Siva N.. 1 000 genomes project. Nat Biotechnol 2008 ; 26 : 256. [CrossRef] [PubMed] [Google Scholar]
- Jordan B.. L’humanité au pluriel, la génétique et la question des races humaines. Paris : Seuil, 2008. [Google Scholar]
- Gibbons A. Strange bedfellows for human ancestors. Science 2020; 367 : 838–39. [Google Scholar]
- Enard W, Przeworski M, Fisher SE, et al. Molecular evolution of FOXP2, a gene involved in speech and language. Nature 2002 ; 418 : 869–872. [Google Scholar]
- Charlier P, Coppens Y, Héry-Arnaud G, Hassin J. Une anthropologie biologique de la disparition de l’homme de Néandertal : données récentes. Med Sci (Paris) 2018 ; 34 : 745–748. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Henry JP. Génétique et origine d’Homo sapiens. Med Sci (Paris) 2019 ; 35 : 39–45. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Deschamps M, Quintana-Murci L. Immunité innée et maladies chez l’homme : de l’introgression archaïque à la sélection naturelle. Med Sci (Paris) 2016 ; 32 : 1079–1086. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1. ADN de l’homme de Néandertal dans différentes populations (échelle verticale, en mégabases par individu). La tache indiquée pour chaque population (appelée violin plot) représente la distribution observée et ses valeurs extrêmes. À droite, la population africaine est éclatée en cinq groupes. EAS, East Asian ; SAS, South Asian ; EUR, European ; AMR, AdMixed Americans (Mexique, Porto Rico, Colombie, Pérou) ; ACB/ASW, African Caribbean & Afro-American ; AFR, African. Pour les groupes Africains : LWK, Luhya, Kenya ; GWD, Gambien, Gambie de l’Ouest ; MSL, Mende, Sierra Leone ; YRI, Yoruba, Nigeria ; ESN, Esan, Nigeria. Voir : https:// www.internationalgenome.org/faq/which-populations-are-part-your-study/ (extrait partiel et modifié de la figure 2 de [5]). |
| Dans le texte | |
 |
Figure 2. Ce diagramme indique l’ensemble des séquences néandertaliennes non redondantes trouvées chez les non-Africains (1 013 Mb, en bleu) et chez les Africains (259+16 Mb, orange). La quasi-totalité des séquences de Neandertal africaines sont aussi présentes chez les non-Africains (extrait partiel et modifié de la figure 2 de [5]). |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




