Figure 1.
Télécharger l'image originale
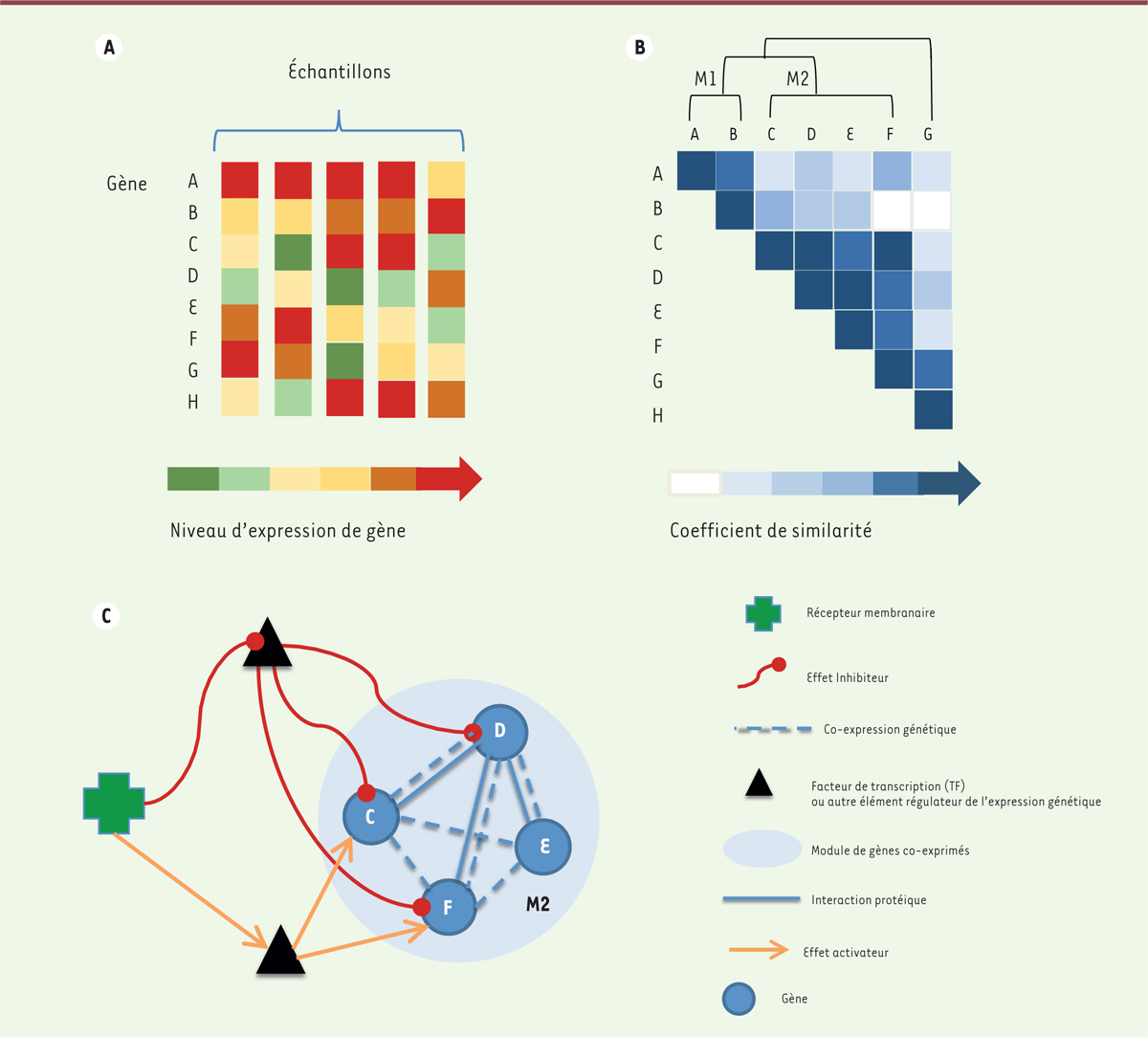
Du profil d’expression au réseau de régulation génique. A. Les profils d’expression pour chaque gène considéré (ici, on considère 7 gènes notés A, B, C, D, E, F et G) sont construits à partir de données transcriptomiques issues de différents échantillons, en concaténant les niveaux d’expression du gène considéré pour chaque condition testée. B..On peut inférer les modules de coexpression de gènes en calculant la distance entre les profils d’expression de chaque paire de gènes d’intérêt et en appliquant une méthode de classification non supervisée afin de les grouper en modules de gènes ayant des niveaux d’expression similaires sous les mêmes conditions (ici, deux modules sont inférés, M1 comportant les gènes A et B, et M2 contenant les gènes C, D, E et F). C. Réseau de régulation génique ou GRN (gene regulatory network) considérant les éléments régulateurs de l’expression des gènes du module M2, par exemple, les facteurs de transcription (TF), et un récepteur membranaire. Les interactions de régulation (activatrices ou inhibitrices de l’expression) sont soit inférées par corrélation entre les niveaux d’expression, soit prouvées expérimentalement (et une relation causale est alors établie).
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




