| Issue |
Med Sci (Paris)
Volume 32, Number 2, Février 2016
|
|
|---|---|---|
| Page(s) | 141 - 143 | |
| Section | Nouvelles | |
| DOI | https://doi.org/10.1051/medsci/20163202002 | |
| Published online | 02 March 2016 | |
Les paralogues de SUMO et la réponse interféron
SUMO paralogs and interferon response
Inserm UMR-S 1124, université Paris Descartes, 45, rue des Saints Pères, 75006 Paris, France
Les interférons
Les interférons (IFN) constituent une famille de cytokines impliquée dans diverses activités biologiques. Ils ont initialement été identifiés et nommés pour leur activité antivirale, mais ils exercent également des activités immuno-régulatrices, anti-prolifératives et apoptotiques. Les IFN activent la voie de signalisation JAK/STAT (Janus kinase/signal transducer and activator of transcription) et induisent la transcription de plus de 300 gènes, les ISG (IFN-stimulated genes), dont les produits sont les médiateurs des activités biologiques des IFN [1].
Les IFN sont classés en trois types sur la base de leur antigénicité et de la spécificité de chaque type à un récepteur membranaire. L’IFN de type I regroupe sept interférons parmi lesquels deux sont majoritaires : les IFNα et β [1]. La fixation des IFN de type I à leur récepteur permet l’activation de deux kinases, JAK1 (Janus kinase 1) et Tyk2 (tyrosine kinase 2), ainsi que la phosphorylation des facteurs de transcription STAT1 et STAT2 (signal transducer and activator of transcription). Les hétérodimères phosphorylés STAT1-STAT2 s’associent à IRF9 (IFN regulatory factor 9) pour former le complexe ISGF3 (IFN stimulated gene factor 3) qui migre vers le noyau et se fixe sur des séquences promotrices ISRE (IFN stimulated response element) (Figure 1A). Le facteur de transcription STAT1 est essentiel pour la formation du complexe ISGF3, comme le démontre l’observation que les souris invalidées pour le gène STAT1 ne répondent pas aux IFN de type I [2]. Il y a cependant de plus en plus de preuves que STAT2 peut former avec IRF9 un complexe alternatif « ISGF3-like », en l’absence de STAT1, capable de générer une réponse aux IFN de type I et III [3]. Les cellules n’exprimant pas STAT1 (STAT1-/-) ont un niveau d’expression de STAT2 très faible mais la réintroduction de STAT2 dans ces cellules restaure la réponse aux IFN de type I [4].
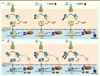 |
Figure 1. Modèle pour l’inhibition du signal de transduction de l’IFN par SUMO1 et SUMO3. A. Fixation de l’IFNα à son récepteur induit la phosphorylation de STAT1 et STAT2 qui forment des hétérodimères STAT1-STAT2. Ces hétérodimères s’associent au facteur de transcription IRF9 pour former le complexe ISGF3 qui migre vers le noyau et se fixe au motif ISRE. Il active alors la transcription des ISG tels que PML, TAP1 et PKR. L’expression de SUMO1 ou SUMO3 inhibe la phosphorylation de STAT1 en réponse à l’IFNα mais n’altère pas la liaison de ISGF3 à l’ISRE. Le complexe alternatif « ISGF3-like » est en fait formé par l’homodimère STAT2-STAT2 et IRF9. L’IFNα induit la conjugaison de PML à SUMO3 ce qui entraîne sa dégradation via le protéasome. Les réponses biologiques médiées par PML dans les cellules traitées à l’IFNα sont altérées. B. Fixation de l’IFNγ à son récepteur induit la phosphorylation de STAT1 qui forme des homodimères. Ces homodimères migrent vers le noyau, se fixent au motif GAS et activent la transcription des ISG tels que PML, PKR, IRF1, TAP1 et IP10. PML régule positivement le signal de transduction et la transcription en réponse à l’IFNγ en induisant une plus forte activation de STAT1. L’expression de SUMO1 ou SUMO3 inhibe la phosphorylation de STAT1 et la transcription en réponse à l’IFNγ. L’IFNγ induit la conjugaison de PML à SUMO3 ce qui entraîne sa dégradation via le protéasome et une plus forte inhibition de la transcription en réponse à l’IFNγ. GAS : gamma activation site ; IFN: interféron ; IP10 : IFN gamma-induced protein 10 ; IRF1/9 : IFN regulatory factor 1/9 ; ISG : IFN-stimulated gene ; ISGF3 : IFN stimulated gene factor 3 ; ISRE : IFN stimulated response element ; JAK1/2 : Janus kinase 1/2 ; PKR : double-stranded RNA-dependent protein kinase ; PML : promyelocytic leukaemia ; RNF4 : RING finger protein 4 ; STAT : signal transducer and activator of transcription ; SUMO : small ubiquitin modifier ; TAP1 : transporter associated with antigen processing 1. |
L’IFN de type II ne comprend qu’un seul membre, l’IFNγ. Après fixation de l’IFNγ à son récepteur, STAT1 est phosphorylé [1] par JAK1 et JAK2 et forme des homodimères qui transloquent dans le noyau où ils induisent la transcription de gènes ayant une séquence GAS (gamma activation site) dans leur promoteur (Figure 1B).
L’IFN de type III, également appelé IFNλ, a été découvert plus récemment et comporte quatre membres, IFNλ1 à IFNλ4. Les IFNλ se fixent à des récepteurs spécifiques exprimés à la surface des cellules épithéliales mais induisent la même signalisation que les IFN de type I.
Les protéines SUMO
Les protéines SUMO (small ubiquitin modifier) appartiennent à la famille des UBL (ubiquitin like proteins) [5]. Chez l’homme, il existe 4 paralogues1 de SUMO : SUMO1, SUMO2 et SUMO3 qui sont ubiquitaires, contrairement à SUMO4 qui est spécifique des cellules rénales, de la rate et des ganglions lymphatiques. SUMO2 et SUMO3 possèdent des séquences identiques à 95 %, mais qui ne sont identiques qu’à 50 % avec la séquence de SUMO1 [5]. Par conséquent, les protéines SUMO2 et SUMO3 sont reconnues par le même anticorps SUMO2/3.
SUMO2 et SUMO3, mais pas SUMO1, possèdent le motif consensus ΨKXD/E2 leur permettant de former des chaînes polySUMO2/3. Néanmoins, SUMO1 peut terminer une chaîne poly SUMO2/3.
La SUMOylation est une modification réversible qui implique la liaison covalente isopeptidique entre un résidu glycine dans la région C-terminale de SUMO et une lysine de la protéine substrat. La SUMOylation est impliquée dans de nombreux processus cellulaires tels que la régulation transcriptionnelle, la dégradation des protéines et la défense antivirale. Depuis la découverte de SUMO en 1996, le nombre de ses substrats identifiés dépasse les 500 [6].
SUMO inhibe la phosphorylation de STAT1 en réponse à l’IFNα et l’IFNγ
Un travail antérieur avait montré que STAT1 rendue non SUMOylable est hyper-phosphorylée en réponse à l’IFNγ [7]. Nous avons montré [8] que dans les cellules qui expriment en stable les différents paralogues de SUMO : (1) STAT1 interagit avec SUMO et est SUMOylée, ce qui réduit sa phosphorylation et sa translocation vers le noyau en réponse aux IFN de type I et type II ; (2) l’IFN régule négativement son propre signal en augmentant la SUMOylation de STAT1 ; et (3) bien que SUMO diminue la phosphorylation de STAT1 en réponse à l’IFNα, il n’affecte ni la phosphorylation de STAT2 ni sa translocation vers le noyau (Figure 1A).
Comme attendu, la réduction de l’activation de STAT1 par SUMO en réponse à l’IFNγ entraîne une diminution de la liaison de l’homodimère phosphorylé STAT1-STAT1 à la séquence GAS (Figure 1B). Par contre, la diminution de la phosphorylation de STAT1 en réponse à l’IFNα n’altère pas la liaison du complexe ISGF3 à la séquence ISRE, le complexe alternatif « ISGF3- like » ne nécessitant pas STAT1 phosphorylé puisqu’il est formé par l’homodimère phosphorylé STAT2-STAT2 et IRF9 (Figure 1A) [8].
SUMO inhibe la transcription en réponse à l’IFNγ sans altérer celle de l’IFNα
L’effet de SUMO sur la transcription a été testé en utilisant les séquences promotrices spécifiques de l’IFNα et l’IFNγ, respectivement ISRE et GAS, fusionnées à la luciférase. L’expression de SUMO1 ou de SUMO3 n’altère pas la transcription en réponse à l’IFNα mais inhibe la transcription en réponse à l’IFNγ (Figure 1). Fait intéressant sur lequel on reviendra plus loin, l’inhibition de la transcription en réponse à IFNγ est de 30 % dans les cellules qui surexpriment SUMO1 et de 80 % dans les cellules qui surexpriment SUMO3.
L’augmentation de la conjugaison de PML à SUMO en réponse à l’IFN entraîne sa dégradation via le protéasome
PML (promyelocytic leukaemia) est l’une des protéines produite par les gènes dont la transcription est induite par les IFN, les ISG (IFN-stimulated genes). PML permet la formation de structures appelées corps nucléaires PML (PML NBs, PML nuclear bodies) (Figure 1) [9–12] (→).
(→) Voir la Nouvelle de T. Regad et M.K. Chelbi-Alix, m/s n° 1, janvier 2002, page 25, et la Synthèse de M.A. Maroui et al., m/s n° 8-9, août-septembre 2014, page 765
Il est connu que l’arsenic augmente la SUMOylation de PML et que SUMO2/3 peut agir comme un signal de recrutement de RNF4 (RING finger protein 4), une SUMO-E3-ligase qui se lie aux polychaînes SUMO de PML (Figure 1). Ceci entraîne l’ubiquitination de PML et sa dégradation dans les cellules traitées à l’arsenic [13, 14]. Dans les cellules qui expriment SUMO3, le traitement à l’IFNα ou l’IFNγ induit la conjugaison de PML à SUMO3 à des temps précoces. L’expression de l’ARNm de PML a donc lieu sans augmentation de la quantité de la protéine PML, celle-ci étant dégradée via le protéasome. Cette dégradation de PML n’est pas observée quand ces cellules sont traitées avec un inhibiteur du protéasome ou déplétées en RNF4.
L’inhibition massive de la réponse transcriptionnelle de l’IFNγ par SUMO3 est due à la dégradation de PML
L’expression de chaque isoforme nucléaire de PML régule positivement la phosphorylation de STAT1 et la transcription en réponse à l’IFNγ (Figure 1B) [15]. La déplétion en RNF4 stabilise PML et augmente donc la phosphorylation de STAT1 et la transcription en réponse à l’IFNγ [8].
Comme mentionné précédemment, la transcription en réponse à IFNγ est inhibée de 30 % par SUMO1 et de 80 % par SUMO3 [8]. Cette plus forte inhibition est en fait due, dans les cellules exprimant SUMO3, à la dégradation de PML SUMOylée par le protéasome. En effet, lorsque ces cellules sont déplétées en RNF4, on observe une restauration de la concentration en protéine PML, ce qui ramène l’inhibition de la transcription de 80 % à 30 %.
Conclusions
L’inhibition de la voie de signalisation de l’IFNγ par SUMO est associée à une réduction de l’apoptose, de l’inhibition de la croissance cellulaire et du chimiotactisme. De façon réciproque, l’inhibition de la SUMOylation par diminution de l’expression d’Ubc9 (ubiquitin conjugating enzyme 9), unique enzyme de conjugaison, entraîne en réponse à l’IFNγ une augmentation de la phosphorylation de STAT1 et des réponses biologiques. L’expression de SUMO rend donc les cellules résistantes à l’IFNγ. De plus, SUMO3, mais pas SUMO1, bloque la capacité de l’IFNα et de l’IFNγ à augmenter l’expression de la protéine PML et réduit la capacité de l’IFN à conférer la résistance au virus de l’encéphalomyocardite (EMCV) [8]. Ceci est en accord avec des résultats précédents montrant que PML joue un rôle clé pour inhiber l’EMCV [16].
Pris dans leur ensemble, nos résultats mettent en évidence un rôle nouveau de SUMO dans la modulation de la réponse aux interférons de type I et II.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Remerciements
Ce travail a été financé par l’Agence Nationale de la Recherche (ANR 11BSV3002803)
Références
- Schneider WM, Chevillotte MD, Rice CM. Interferon-stimulated genes: a complex web of host defenses. Annu Rev Immunol 2014 ; 32 : 513–545. [CrossRef] [PubMed] [Google Scholar]
- Meraz MA, White JM, Sheehan KC, et al. Targeted disruption of the Stat1 gene in mice reveals unexpected physiologic specificity in the JAK-STAT signaling pathway. Cell 1996 ; 84 : 431–442. [CrossRef] [PubMed] [Google Scholar]
- Fink K, Grandvaux N. STAT2 and IRF9: Beyond ISGF3. JAKSTAT 2013 ; 2 : e27521. [PubMed] [Google Scholar]
- Blaszczyk K, Olejnik A, Nowicka H, et al. STAT2/IRF9 directs a prolonged ISGF3-like transcriptional response and antiviral activity in the absence of STAT1. Biochem J 2015 ; 466 : 511–524. [CrossRef] [PubMed] [Google Scholar]
- Johnson ES. Protein modification by SUMO. Annu Rev Biochem 2004 ; 73 : 355–382. [CrossRef] [PubMed] [Google Scholar]
- Lamoliatte F, Caron D, Durette C, et al. Large-scale analysis of lysine SUMOylation by SUMO remnant immunoaffinity profiling. Nat Commun 2014 ; 5 : 5409. [CrossRef] [PubMed] [Google Scholar]
- Begitt A, Droescher M, Knobeloch KP, Vinkemeier U. SUMO conjugation of STAT1 protects cells from hyperresponsiveness to IFNgamma. Blood 2011 ; 118 : 1002–1007. [CrossRef] [PubMed] [Google Scholar]
- Maarifi G, Maroui MA, Dutrieux J, et al. Small ubiquitin-like modifier alters IFN response. J Immunol 2015 ; 195 : 2312–2324. [CrossRef] [PubMed] [Google Scholar]
- Chelbi-Alix MK, Quignon F, Pelicano L, et al. Resistance to virus infection conferred by the interferon-induced promyelocytic leukemia protein. J Virol 1998 ; 72 : 1043–1051. [PubMed] [Google Scholar]
- Geoffroy MC, Chelbi-Alix MK. Role of promyelocytic leukemia protein in host antiviral defense. J Interferon Cytokine Res 2011 ; 31 : 145–158. [Google Scholar]
- Regad T, Chelbi-Alix MK. PML, un nouvel intermédiaire de l’effet antiviral de l’interféron. Med Sci (Paris) 2002 ; 18 : 25–27. [CrossRef] [EDP Sciences] [Google Scholar]
- Maroui MA, El Asmi F, Dutrieux J, et al. Implication des corps nucléaires PML dans l’immunité intrinsèque et innée. Med Sci (Paris) 2014 ; 30 : 765–771. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Tatham MH, Geoffroy MC, Shen L, et al. RNF4 is a poly-SUMO-specific E3 ubiquitin ligase required for arsenic-induced PML degradation. Nat Cell Biol 2008 ; 10 : 538–546. [CrossRef] [PubMed] [Google Scholar]
- Maroui MA, Kheddache-Atmane S, El Asmi F, et al. Requirement of PML SUMO interacting motif for RNF4- or arsenic trioxide-induced degradation of nuclear PML isoforms. PLoS One 2012 ; 7 : e44949. [CrossRef] [PubMed] [Google Scholar]
- El Bougrini J, Dianoux L, Chelbi-Alix MK. PML positively regulates interferon gamma signaling. Biochimie 2011 ; 93 : 389–398. [CrossRef] [PubMed] [Google Scholar]
- Maroui MA, Pampin M, Chelbi-Alix MK. Promyelocytic leukemia isoform IV confers resistance to encephalomyocarditis virus via the sequestration of 3D polymerase in nuclear bodies. J Virol 2011 ; 85 : 13164–13173. [CrossRef] [PubMed] [Google Scholar]
© 2016 médecine/sciences – Inserm
Liste des figures
 |
Figure 1. Modèle pour l’inhibition du signal de transduction de l’IFN par SUMO1 et SUMO3. A. Fixation de l’IFNα à son récepteur induit la phosphorylation de STAT1 et STAT2 qui forment des hétérodimères STAT1-STAT2. Ces hétérodimères s’associent au facteur de transcription IRF9 pour former le complexe ISGF3 qui migre vers le noyau et se fixe au motif ISRE. Il active alors la transcription des ISG tels que PML, TAP1 et PKR. L’expression de SUMO1 ou SUMO3 inhibe la phosphorylation de STAT1 en réponse à l’IFNα mais n’altère pas la liaison de ISGF3 à l’ISRE. Le complexe alternatif « ISGF3-like » est en fait formé par l’homodimère STAT2-STAT2 et IRF9. L’IFNα induit la conjugaison de PML à SUMO3 ce qui entraîne sa dégradation via le protéasome. Les réponses biologiques médiées par PML dans les cellules traitées à l’IFNα sont altérées. B. Fixation de l’IFNγ à son récepteur induit la phosphorylation de STAT1 qui forme des homodimères. Ces homodimères migrent vers le noyau, se fixent au motif GAS et activent la transcription des ISG tels que PML, PKR, IRF1, TAP1 et IP10. PML régule positivement le signal de transduction et la transcription en réponse à l’IFNγ en induisant une plus forte activation de STAT1. L’expression de SUMO1 ou SUMO3 inhibe la phosphorylation de STAT1 et la transcription en réponse à l’IFNγ. L’IFNγ induit la conjugaison de PML à SUMO3 ce qui entraîne sa dégradation via le protéasome et une plus forte inhibition de la transcription en réponse à l’IFNγ. GAS : gamma activation site ; IFN: interféron ; IP10 : IFN gamma-induced protein 10 ; IRF1/9 : IFN regulatory factor 1/9 ; ISG : IFN-stimulated gene ; ISGF3 : IFN stimulated gene factor 3 ; ISRE : IFN stimulated response element ; JAK1/2 : Janus kinase 1/2 ; PKR : double-stranded RNA-dependent protein kinase ; PML : promyelocytic leukaemia ; RNF4 : RING finger protein 4 ; STAT : signal transducer and activator of transcription ; SUMO : small ubiquitin modifier ; TAP1 : transporter associated with antigen processing 1. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




