| Issue |
Med Sci (Paris)
Volume 31, Number 3, Mars 2015
|
|
|---|---|---|
| Page(s) | 261 - 267 | |
| Section | M/S Revues | |
| DOI | https://doi.org/10.1051/medsci/20153103011 | |
| Published online | 08 April 2015 | |
Le cœur des ARN non codants
Un long chemin à découvrir
In the heart of noncoding RNA: a long way to go
Unité de cardiologie expérimentale, département de médecine, centre hospitalier universitaire vaudois, 1011
Lausanne, Suisse
La récente identification des longs ARN non codants et le début de leur caractérisation fonctionnelle dans une variété de tissus constituent des avancées scientifiques majeures. Ces avancées ont permis de comprendre les mécanismes moléculaires contrôlant l’expression des gènes en réponse à des changements environnementaux et dans l’adaptation physiopathologique des organes. En particulier, les longs ARN non codants apparaissent comme d’importants régulateurs épigénétiques régulant le remodelage de la chromatine. Dans le cœur, ces transcrits contrôlent la spécification et la différenciation cellulaires au cours du développement. Par ailleurs, ils participent, chez l’adulte, à l’adaptation au stress hémodynamique faisant suite à un dommage tissulaire. La haute spécificité cellulaire d’expression des longs ARN non codants fait de ces molécules des cibles thérapeutiques et des biomarqueurs extrêmement attractifs.
Abstract
The identification and characterization of long noncoding RNA in a variety of tissues represent major achievements that contribute to our understanding of the molecular mechanisms controlling gene expression. In particular, long noncoding RNA play crucial roles in the epigenetic regulation of the adaptive response to environmental cues via their capacity to target chromatin modifiers to specific locus. In addition, these transcripts have been implicated in controlling splicing, translation and degradation of messenger RNA. Long noncoding RNA have also been shown to act as decoy molecules for microRNA. In the heart, a few long noncoding RNA have been demonstrated to regulate cardiac commitment and differentiation during development. Furthermore, recent findings suggest their involvement as regulators of the pathophysiological response to injury in the adult heart. Their high cellular specificity makes them attractive target molecules for innovative therapies and ideal biomarkers.
© 2015 médecine/sciences – Inserm
Organisation du génome et régulation en réseau
En 2003, le génome humain était finalement séquencé. Cet indéniable succès scientifique, vu à l’époque comme l’étape indispensable à la compréhension des processus biologiques, a rapidement fait place à d’autres questions. En effet, connaître la séquence de tous les gènes ne donne aucune indication sur les mécanismes d’activation et sur l’orchestration nécessaire qui contrôlent l’expression temporelle et spatiale de ces gènes. Ensuite, il est rapidement apparu que les gènes codant pour les protéines ne représentaient qu’une petite fraction du génome, soit environ 2 %. La fonction de la majorité des trois milliards de bases composant le génome nous est en fait encore largement inconnue. À ce propos, des espèces très éloignées sont caractérisées par des différences phénotypiques marquées, alors même que le nombre de gènes codants y est très semblable. Cette simple observation suggère que la complexité biologique tire son origine dans une autre portion du génome. Dans cet univers de séquences non codantes, on trouve notamment les séquences régulatrices, qui permettent l’expression coordonnée des gènes, de manière spécifique dans un organe ou dans une cellule particulière, et qui forment le substrat de la complexité. La création d’un catalogue des régulateurs de l’expression des gènes est nécessaire, non seulement pour mieux comprendre les phénomènes biologiques, mais également l’origine des états pathologiques.
Dans ce contexte, le projet ENCODE (Encyclopedia of DNA elements) s’attache depuis une dizaine d’années à annoter systématiquement le génome (voir le site http://www.nature.com/encode/) ; l’objectif principal est d’identifier les éléments fonctionnels. Un élément fonctionnel peut être défini comme une séquence déterminée sur le génome et portant une information spécifique. Par exemple, l’information peut être celle qui permet la production d’un ARN, codant ou non codant. Mais l’élément fonctionnel peut également être défini par la présence d’une « signature protéique » sur l’ADN, telle que la liaison d’un facteur de transcription ou d’autres régulateurs de l’expression. Il peut s’agir aussi d’une modification biochimique de l’ADN ou des histones. Par exemple, la méthylation des cytosines dans les promoteurs des gènes est généralement associée à une répression de l’expression. D’autre part, la méthylation de la lysine 4 de l’histone 3 (H3K4Me3) marque un promoteur, alors que l’acétylation de l’histone 3 sur la lysine 27 (H3K27Ac) marque une séquence amplificatrice (enhancer) activée. L’ensemble de ces modifications complexes sont la base de la régulation épigénétique de l’expression des gènes. Comme on le voit, on s’affranchit d’un modèle linéaire qui stipule que l’une des quelques 20 000 séquences composant la partie codante du génome contient l’information permettant la synthèse d’un ARN messager qui, lui-même, permettra la synthèse d’une protéine responsable de l’expression d’un caractère. Au contraire, l’un des messages du projet ENCODE est que plus de 80 % du génome contiennent des éléments fonctionnels pouvant être associés à des fonctions biochimiques dans au moins un type de cellules composant notre organisme. L’organisation du génome, telle que l’envisage ENCODE, permet donc une régulation en réseau où de multiples régions sont appelées à moduler l’expression finale d’un ou plusieurs gènes, de façon à assurer une réponse coordonnée.
Rôles des longs ARN non codants
L’avènement de nouvelles technologies n’est évidemment pas étranger à notre capacité à comprendre les régulations complexes qui régissent le génome. En particulier, les techniques de séquençage à haut débit se sont rapidement imposées comme des outils indispensables. Combiné ou non à des méthodes permettant la purification préalable d’éléments fonctionnels, par exemple l’immunoprécipitation spécifique de fragment du génome, le séquençage à haut débit a rendu possible l’étude systématique de l’activité du génome lors de processus biologiques. Notamment, l’analyse du transcriptome est évidemment utile pour étudier de manière exhaustive les modulations de l’expression des gènes en réponse à des stimulus environnementaux ou des modifications génétiques. Cependant, la contribution du séquençage de l’ARN s’est avérée spécialement déterminante pour identifier de nouvelles unités transcriptionnelles. En effet, associée à des analyses bio-informatiques permettant la cartographie et la reconstruction du transcriptome, cette technologie permet d’identifier de nouveaux ARN. On a donc rapidement réalisé qu’un grand nombre de transcrits étaient produits à partir de régions du génome qui ne codaient pas pour des protéines. On connaissait déjà des ARN non codants. Par exemple, les ARN ribosomaux et les ARN de transfert assurent des fonctions dans la traduction des ARN messagers ; les petits ARN nucléaires sont impliqués dans la régulation de l’épissage [39], et les microARN inactivent de façon spécifique des ARN messagers. En revanche, il est devenu évident aujourd’hui que la majorité des ARN non codants est constituée d’autres molécules, que l’on appelle longs ARN non codants [1, 40]. En effet, les régions du génome que l’on pensait être de grands déserts tranquilles s’affirment comme des régulateurs essentiels de l’expression des gènes, à travers leur capacité à produire de longs ARN non codants. Pour une introduction au domaine, on peut suggérer au lecteur les revues [2–5].
Caractéristiques des longs ARN non codants
Les longs ARN non codants sont définis comme des ARN de taille supérieure à environ 200 nucléotides et qui ne possèdent pas, ou très peu, de capacité à coder pour des protéines. Le potentiel codant peut être calculé sur la base de plusieurs paramètres incluant, notamment, la détermination des cadres de lecture de l’ARN, l’identification des codons d’initiation et de terminaison, et la reconnaissance des jonctions d’épissage. On définit également les longs ARN non codants en fonction de la localisation des séquences associées sur le génome. Par rapport au gène codant le plus proche, une séquence générant un long ARN non codant peut être intragénique (chevauchant partiellement les séquences d’un gène codant) ou intergénique (entre deux gènes codants). L’ARN non codant peut être alors transcrit dans le même sens ou en sens contraire de l’ARN messager du gène de référence. Il est important de noter que les longs ARN non codants sont généralement moins conservés que les gènes codants, lorsque l’on compare différentes espèces de vertébrés. Néanmoins, lorsque l’on examine deux espèces proches comme la souris et l’homme, on identifie habituellement des séquences orthologues. Ceci précisé, tant que les principes de la signalisation utilisée par les longs ARN non codants pour communiquer avec leurs partenaires moléculaires ne seront pas connus, il nous sera difficile d’apprécier le degré de conservation dans leurs parties fonctionnelles élémentaires. À ce propos, certains longs ARN non codants partagent avec les ARN messagers une certaine communauté de structure. En effet, une fraction du long transcriptome non codant est composée d’ARN présentant une structure multi-exonique et apparaît donc soumise à des processus d’épissage au même titre que les messagers. Les ARN non codants multi-exoniques sont, par ailleurs, polyadénylés et présentent une coiffe à leur extrémité 5’. D’autre part, il existe des longs ARN non codants dépourvus d’introns qui apparaissent transcrits de façon bidirectionnelle depuis un site d’initiation généralement positionné dans des séquences amplificatrices. Certaines séquences amplificatrices (marquées par H3K27Ac) produisent aussi de longs ARN non codants multi-exoniques, au même titre que des ARN non codants issus de séquences promotrices (marquées par H3K4Me3). À ce stade de notre connaissance, il est difficile de savoir si les longs ARN non codants dérivés de séquences amplificatrices possèdent des fonctions distinctes selon leur structure bidirectionnelle ou multi-exonique. Finalement, les promoteurs de gènes codants peuvent également produire de longs ARN non codants en sens opposé de la transcription du gène codant associé [6, 7]. Au-delà d’une simple signature transcriptionnelle, la fonction de ces longs transcrits n’est pas connue.
Fonctions des longs ARN non codants
Les longs ARN non codants apparaissent investis de diverses fonctions dont le rôle dans les processus physiopathologiques assurant l’intégrité cellulaire et l’homéostasie des organes reste à définir. Cependant, l’étude de systèmes modèles a permis d’élucider certains mécanismes moléculaires associés à cette nouvelle classe de molécules (Figure 1). En particulier, les longs ARN non codants jouent un rôle important comme régulateurs de la transcription. En effet, de nombreux longs ARN non codants sont impliqués dans le remodelage de la chromatine. Plus précisément, ils permettent de cibler des complexes modificateurs de la chromatine vers des locus spécifiques [3]. Par exemple, les longs ARN non codants HOTAIR (HOX transcript antisense RNA) et ANRIL (antisens non-coding RNA in the INK4 locus) [41] s’associent avec le complexe répresseur 2 du polycomb (PRC2) et fonctionnent comme initiateurs de l’inhibition de la transcription de gènes en induisant la production de marques répressives (H3K27Me3) sur la chromatine cible [8, 9]. D’autre part, les longs ARN non codants peuvent réguler la transcription via une interaction directe avec des composants essentiels de la machinerie transcriptionnelle, notamment en déplaçant des éléments fonctionnels, comme la polymérase II ou des facteurs de transcription. Ils peuvent également occuper des sites régulateurs spécifiques sur l’ADN, empêchant par là même la liaison d’activateurs de la transcription. Par exemple, GAS5 (growth arrest-specific 5) imite par sa structure le site de liaison du récepteur aux glucocorticoïdes et réprime les évènements transcriptionnels associés à ces hormones [10]. Par ailleurs, des longs ARN non codants contribuent à la diversité en s’appariant aux ARN messagers, en bloquant les sites d’épissage alternatif et en favorisant l’apparition préférentielle de certains variants [11]. Une autre fonction des longs ARN non codants semble être liée à leur capacité à être reconnus par des microARN. En effet, les longs ARN non codants agissent parfois comme des leurres, permettant aux ARN messagers ciblés par les microARN d’échapper à la dégradation. Le trio ARN messager, microARN et long ARN non codant forme alors un réseau appelé compétitif (competitive endogenous network), où le degré d’expression de l’un des éléments du réseau influence les niveaux des deux autres composants [12].
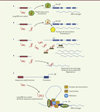 |
Figure 1. Rôles des longs ARN non codants. Les longs ARN non codants agissent à travers divers mécanismes pour réguler l’expression des gènes codants. A. Ils dirigent des modificateurs de la chromatine vers des gènes cibles spécifiques. B. Ils déplacent des régulateurs de l’expression (activateurs ou répresseurs) des séquences régulatrices de l’expression. C. Ils entrent en compétition avec des microARN et protègent des ARN messagers de la dégradation par ces microARN. D. Ils modulent des évènements d’épissage et interfèrent avec la traduction. E. Ils promeuvent l’association des séquences amplificatrices et promotrices pour initier la transcription. La Figure décrit une activation en cis, mais l’association peut aussi s’effectuer avec une région du génome non adjacente pour activer un gène en trans. |
Les longs ARN non codants dérivés de séquences amplificatrices
Une importante sous-classe de longs ARN non codants comprend les longs ARN non codants dérivés de séquences amplificatrices (enhancer-associated long noncoding RNA) [13, 14]. Les séquences amplificatrices sont des composants essentiels du génome qui permettent l’expression spécifique des gènes dans les cellules et les tissus. Ces séquences contrôlent également la temporalité de l’expression au moment de la différenciation cellulaire, et donc pendant le développement embryonnaire. On estime que le génome humain contient environ un million de séquences amplificatrices [15]. Ce nombre, comparé aux quelques 20 000 gènes codants, suggère que ces gènes sont chacun sous le contrôle de nombreuses séquences amplificatrices, et démontre l’énorme complexité de la régulation de l’expression. Les séquences amplificatrices régulent l’expression des gènes codants immédiatement adjacents à travers une régulation en cis, mais ces mêmes séquences peuvent aussi réguler l’expression de gènes éloignés en trans. En effet, les séquences amplificatrices activées s’associent physiquement à plusieurs régions du génome et coordonnent ainsi l’expression de gènes situés dans des locus différents. La première démonstration d’une transcription généralisée à partir de site de séquences amplificatrices activées a été obtenue par un séquençage à haut débit d’ARN isolé de neurones [16]. Cette observation a été ensuite confirmée dans de nombreux types cellulaires et dans plusieurs espèces. La transcription de séquences amplificatrices apparaît dès lors comme une signature caractéristique d’un état d’activation. Brièvement, l’expérience montre que les transcrits sont produits à partir de régions du génome caractérisées par la présence de marques épigénétiques H3K4Me1 au moment où ces séquences acquièrent une marque d’activation, telle que H3K27Ac [17, 18].
Le mécanisme d’action des longs ARN non codants dérivés de séquences amplificatrices est encore le sujet d’intenses recherches. Cependant, il apparaît que ces molécules d’ARN jouent un rôle crucial dans la formation des boucles de chromatine qui permettent la mise en contact des séquences amplificatrices avec les sites d’initiation de la transcription de leurs gènes cibles [19–21]. En d’autres termes, les longs ARN non codants initialisent, et probablement stabilisent, l’association des séquences amplificatrices et des promoteurs cibles, favorisant par là même la transcription (Figure 1E). L’importance des longs ARN non codants dérivés de séquences amplificatrices dans ce processus est formellement démontrée par des expériences d’inactivation, dans lesquelles ces ARN sont dégradés par l’utilisation d’oligonucléotides spécifiques, résultant par voie de conséquence en une extinction de l’expression des gènes cibles [22].
De façon extrêmement intéressante, la vaste majorité des polymorphismes mononucléotidiques identifiés dans des études d’associations pangénomiques et associés à des traits phénotypiques liés à des pathologies chez l’homme, se situent dans des séquences amplificatrices [23–25]. Il est dès lors probable qu’une grande partie de ces mutations ponctuelles modifient l’expression ou la fonction de transcrits non codants issus des séquences amplificatrices. De plus, les polymorphismes associés à des pathologies d’un organe particulier affectent principalement des séquences amplificatrices actives spécifiquement dans l’organe considéré [25]. Par exemple, les polymorphismes associés à des changements phénotypiques de paramètres électrocardiographiques se situent préférentiellement dans des séquences amplificatrices actives dans le cœur. Cette dernière observation souligne à quel point la caractérisation fonctionnelle des longs ARN non codants est cruciale pour notre compréhension des pathologies humaines et pour notre capacité à produire des traitements personnalisés.
Les longs ARN non codants et le cœur
La démonstration de l’importance des longs ARN non codants dans le cœur n’en est qu’à ses débuts. Une vingtaine d’études sont publiées à ce jour, mais on peut prédire une intensification de la recherche dans ce domaine au cours des prochaines années. On peut globalement séparer les études s’intéressant au rôle des longs ARN non codants dans la différenciation cardiogénique de celles qui recherchent l’implication des longs ARN non codants dans l’homéostasie cardiaque. Dans ce dernier cas, les chercheurs s’intéressent, notamment, à découvrir de nouveaux marqueurs de l’état physiopathologique du cœur.
Braveheart, Fendrr et Kcnq1 au cours du développement cardiaque
La première étude publiée dans le domaine s’est intéressée à découvrir des longs ARN non codants en utilisant principalement une stratégie par comparaison d’expression. Dès lors, en comparant l’expression de longs ARN non codants dans des cellules souches embryonnaires et dans différents tissus, en particulier le cœur, les auteurs ont identifié Braveheart, un long ARN non codant contrôlant la spécification des cellules non différenciées en mésoderme cardiaque [26]. Des cellules souches embryonnaires, dans lesquelles Braveheart est inactivé, ne sont plus capables de se différencier en cardiomyocytes. Il semble que Braveheart se situe, hiérarchiquement, en amont d’un réseau de gènes cardiogéniques comprenant, en particulier, d’importants facteurs de transcription tels MesP1 (mesoderm posterior 1 homolog), Hand1 (heart- and neural crest derivatives-expressed protein 1), Hand2, Nkx2.5 (NK2 homeobox 5) and Tbx20 (T-box 20). Il faut remarquer que ces études ont été menées chez la souris et qu’aucun orthologue de Braveheart n’a été découvert chez l’homme. Ce fait ne remet pas en cause l’importance de Braveheart, mais soulève la question de la conservation de certains longs ARN non codants. Par ailleurs, le mécanisme moléculaire associé à l’activité de Braveheart n’est pas complètement clair. Braveheart s’apparie avec PRC2 (complexe répresseur polycomb) et donc contrôle la répression de gènes cibles. Dans des cellules souches embryonnaires dépourvues de Braveheart, le promoteur de MesP1 est enrichi en marques répressives H3K27Me3. Il semblerait donc que le potentiel cardiogénique de Braveheart s’appuie sur sa capacité à déplacer PRC2 des promoteurs de gènes cibles cardiogéniques, en particulier MesP1, pour promouvoir leur expression et donc la différenciation cellulaire.
De façon similaire, Fendrr (FOXF1 adjacent non-coding developmental regulatory RNA) est un long ARN non codant exprimé de façon transitoire dans le mésoderme latéral pendant le développement [27]. Fendrr est exprimé à partir d’un locus contenant Foxf1 (forkhead box F1), un gène essentiel à la détermination du mésoderme latéral. Ce transcrit se lie également à PRC2 et permet de cibler la formation de marques répressives vers des gènes spécifiques, en particulier Foxf1 et Pitx2 (paired-like homeodomain transcription factor 2). Cependant, Fendrr interagit aussi avec un composant de TrxG (Trithorax group), un complexe modificateur épigénétique et activateur de gènes. Fendrr joue donc un rôle dans le maintien de l’équilibre entre gènes activés et réprimés au cours du développement. L’abolition de l’expression de Fendrr induit des malformations fœtales, notamment des malformations du cœur, qui démontrent l’implication de Fendrr dans le contrôle de la morphogenèse cardiaque. Finalement, Kcnq1ot1 (K voltage-gated channel, member 1, overlapping transcript 1) est un long ARN non codant contrôlant l’expression de Kcnq1 pendant le développement du cœur [28].
Longs ARN non codants et hypertrophie cardiaque dans le cœur adulte
Dans le cœur adulte, Chrf (cardiac hypertrophy-related factor) est un long ARN non codant produit en réponse à un stimulus hypertrophique, en l’occurrence l’administration d’angiotensine II [29]. Ce transcrit est reconnu par un microARN, miR-489, qui lui-même cible un important régulateur de l’hypertrophie, Myd88 (myeloid differentiation primary response gene 88). L’inactivation de Chrf réduit l’expression de Myd88, alors que sa surexpression l’augmente. Ces observations suggèrent que Chrf s’oppose aux effets négatifs de miR-489 sur Myd88, et que ces trois transcrits forment un réseau endogène compétitif régulant le niveau de la réponse adaptative. L’expression cardiaque de Chrf en réponse au stress favorise donc l’émergence d’une hypertrophie compensatoire en permettant l’activation de voies d’activation sous le contrôle de Myd88.
Un autre cas intéressant de long ARN non codant impliqué dans la régulation de l’hypertrophie cardiaque est celui de Mhrt (MyHeart) [30]. Ce transcrit est produit à partir du locus contenant Myh6 (myosin, heavy chain 6, protein) et Myh7, codant respectivement pour les isoformes α et β de la chaîne lourde de la myosine. Mhrt est transcrit en sens inverse de Myh7, et représente un exemple d’ARN non codant anti-sens, chevauchant des séquences codantes. Pendant le développement de l’hypertrophie cardiaque, on observe un changement d’expression des isoformes de la myosine. Le cœur hypertrophié exprime principalement Myh7 au détriment de Myh6. Les niveaux d’expression cardiaque de Mhrt sont diminués en réponse à une surcharge de pression, suggérant que la diminution de ce transcrit non codant permet la réexpression de Myh7. Effectivement, si l’on force le cœur à exprimer Mhrt, le changement d’isoformes de la myosine est aboli et le cœur résiste mieux au stress imposé par la surcharge de pression. De façon intéressante, Myh6 et Mhrt sont sous contrôle d’un même complexe répressif de la chromatine contenant BRG1 (Brahma-related gene 1). Dès lors, BRG1 se lie au promoteur commun de Myh6 et Mhrt pour induire leur répression. Cependant, un mécanisme de feedback s’opère également sur BRG1 via Mhrt. En effet, Mhrt séquestre BRG1 et l’empêche d’exercer ses fonctions répressives sur la chromatine dans le cœur normal. Ces mécanismes fondamentaux de la physiologie cardiaque démontrent l’importance des longs ARN non codants dans l’homéostasie du cœur.
Longs ARN non codants : biomarqueurs de l’état pathologique cardiaque ?
Les études systématiques de l’expression de longs ARN non codants dans le cœur sont encore peu nombreuses [31–35]. Une des raisons principales est que l’annotation des gènes non codants est encore très succincte. Dès lors, le nombre de longs ARN non codants identifiés à ce jour est relativement petit et ces derniers ne couvrent pas (loin de là) l’ensemble du génome non codant. Par ailleurs, les longs ARN non codants dont l’expression a été évaluée dans le cœur ont d’abord été découverts dans des tissus autres que le cœur [32, 35–37]. Étant donné la haute spécificité tissulaire de ces molécules, il n’est dès lors pas étonnant que les longs ARN non codants connus à ce jour ne soient pas particulièrement exprimés dans les cellules cardiaques. Néanmoins, certaines études ont évalué la possibilité que des longs ARN non codants puissent représenter des marqueurs de pathologies cardiaques détectables dans la circulation [32, 37]. L’étude la plus probante a utilisé une approche par puce contenant quelques milliers de longs ARN non codants annotés pour identifier des transcrits différemment exprimés dans le plasma de patients en phase de transition vers une insuffisance cardiaque [32]. Les auteurs démontrent qu’un long ARN non codant appelé LIPCAR (long intergenic noncoding RNA predicting cardiac remodeling), est un indicateur d’insuffisance cardiaque chronique. L’élévation des taux plasmatiques de LIPCAR peut servir de facteur pronostique permettant de prédire la survie des patients en insuffisance cardiaque. La fonction précise de LIPCAR n’est pas connue. Cependant, il est remarquable qu’une grande proportion des longs ARN non codants détectables dans le plasma avaient, dans cette étude, pour origine l’ADN mitochondrial. Il est dès lors possible que certains de ces longs ARN non codants soient impliqués dans la régulation de l’activité mitochondriale. La détection de l’expression dans les tissus cardiaques chez l’homme n’est évidemment pas chose facile. Néanmoins, une étude a comparé des patients présentant une pathologie cardiaque ischémique à d’autres présentant une pathologie cardiaque non ischémique [31]. Dans une deuxième étape, les auteurs ont répété leur analyse après la pose d’un système d’assistance ventriculaire. L’identification des transcrits non codants par séquençage d’ARN et reconstruction du transcriptome, ainsi que l’analyse de l’expression différentielle, ont permis de définir une signature d’expression de longs ARN non codants distinguant cœurs ischémiques et non ischémiques. De plus, en réponse à la pose d’un dispositif d’assistance et pendant le remodelage tissulaire inverse, une dynamique d’expression marquée des ARN non codants composant cette signature est observée, suggérant que ces transcrits constituent d’excellents marqueurs de pathologies cardiaques.
Longs ARN non codants et infarctus du myocarde
Dans ce contexte, nous avons entrepris d’identifier l’ensemble du transcriptome non codant exprimé dans le cœur à la suite d’un infarctus du myocarde [33]. Cette étude, récemment publiée, décrit la découverte de 1 500 nouveaux gènes non codants dont l’expression de plus de 20 % d’entre eux est modulée après l’infarctus. Les longs ARN non codants associés à ces gènes ont été caractérisés de façon à évaluer leur fonction. La vaste majorité de ces longs ARN non codants sont polyadénylés, multiexoniques et dérivent de séquences amplificatrices. Par ailleurs, la plupart des transcrits ont une haute spécificité cardiaque. Ces deux observations indiquent que les longs ARN non codants, en particulier ceux qui sont modulés en réponse au stress, sont impliqués dans la maintenance de l’intégrité tissulaire [38]. Par ailleurs, il est intéressant de constater que l’expression de ces nouveaux longs ARN non codants est corrélée très étroitement à certains paramètres physiologiques définissant la fonction et les dimensions du cœur. En d’autres termes, les résultats démontrent que les longs ARN non codants non seulement sont spécifiques du cœur, mais aussi qu’ils sont restreints à certaines régions de cet organe, voire même à des sous-populations de cellules, par exemple les fibroblastes cardiaques. Ils peuvent dès lors servir de marqueurs spécifiques de la réactivité vis-à-vis d’un stress et de l’adaptation physiologique qui en résulte. À titre d’exemple, l’expression de certains longs ARN non codants est sensible à un remodelage du ventricule ou du septum, mais n’est pas modifiée lors d’un changement de fonction. Ce type de transcrits pourrait donc s’avérer très utile pour identifier des pathologies, telles que des insuffisances cardiaques avec fonction ventriculaire préservée. On trouve également une corrélation importante entre l’expression des longs ARN non codants dérivés de séquences amplificatrices et celle des gènes codants immédiatement adjacents. L’extinction ciblée de l’expression des longs ARN non codants dérivés de séquences amplificatrices affecte spécifiquement l’expression des gènes adjacents. Ces molécules représentent dès lors des cibles thérapeutiques extrêmement attractives pour moduler l’expression de gènes codants, et donc pour agir sur des voies d’activation particulières. À ce propos, nous avons démontré dans une étude parallèle l’importance des longs ARN non codants dérivés de séquences amplificatrices au cours du développement cardiaque et dans les états pathologiques du cœur [34]. Finalement, il est remarquable que près de 70 % des longs ARN non codants identifiés chez la souris possèdent un orthologue chez l’homme.
Conclusion
L’étude des longs ARN non codants n’en est qu’à ses débuts. Au cours des prochaines années, il est probable que la découverte de nouveaux transcrits va s’accélérer. Les longs ARN non codants représentent des marqueurs très spécifiques des états normaux et pathologiques dans chaque organe. Par ailleurs, ces transcrits peuvent être détectés dans les fluides corporels, ce qui facilite leur utilisation comme marqueurs diagnostiques et pronostiques. Ces molécules ont donc le potentiel de s’imposer comme des cibles incontournables des prochains traitements. En particulier, les thérapies ciblant l’activité transcriptionnelle des séquences amplificatrices de façon à rétablir l’intégrité cellulaire apparaît comme une des approches les plus innovantes des années à venir. La médecine des pathologies cardiaques, un organe dont le pouvoir régénérateur est limité, tirera sans doute un grand bénéfice de ces futurs développements.
Liens d’intérêt
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- Mattick JS. Challenging the dogma: the hidden layer of non-protein-coding RNAs in complex organisms. Bioessays 2003 ; 25 : 930–939. [CrossRef] [PubMed] [Google Scholar]
- Wapinski O, Chang HY. Long noncoding RNAs and human disease. Trends Cell Biol 2011 ; 21 : 354–361. [CrossRef] [PubMed] [Google Scholar]
- Lee JT. Epigenetic regulation by long noncoding RNAs. Science 2012 ; 338 : 1435–1439. [CrossRef] [PubMed] [Google Scholar]
- Hu W, Alvarez-Dominguez JR, Lodish HF. Regulation of mammalian cell differentiation by long non-coding RNAs. EMBO Rep 2012 ; 13 : 971–983. [CrossRef] [PubMed] [Google Scholar]
- Orom UA, Shiekhattar R. Long noncoding RNAs usher in a new era in the biology of enhancers. Cell 2013 ; 154 : 1190–1193. [CrossRef] [PubMed] [Google Scholar]
- Preker P, Nielsen J, Kammler S, et al. RNA exosome depletion reveals transcription upstream of active human promoters. Science 2008 ; 322 : 1851–1854. [CrossRef] [PubMed] [Google Scholar]
- Sigova AA, Mullen AC, Molinie B, et al. Divergent transcription of long noncoding RNA/mRNA gene pairs in embryonic stem cells. Proc Natl Acad Sci USA 2013 ; 110 : 2876–2881. [CrossRef] [Google Scholar]
- Rinn JL, Kertesz M, Wang JK, et al. Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs. Cell 2007 ; 129 : 1311–1323. [CrossRef] [PubMed] [Google Scholar]
- Kotake Y, Nakagawa T, Kitagawa K, et al. Long non-coding RNA ANRIL is required for the PRC2 recruitment to and silencing of p15(INK4B) tumor suppressor gene. Oncogene 2011 ; 30 : 1956–1962. [CrossRef] [PubMed] [Google Scholar]
- Kino T, Hurt DE, Ichijo T, et al. Noncoding RNA gas5 is a growth arrest- and starvation-associated repressor of the glucocorticoid receptor. Sci Signal 2010; 3 : ra8. [PubMed] [Google Scholar]
- Zong X, Tripathi V, Prasanth KV. RNA splicing control: yet another gene regulatory role for long nuclear noncoding RNAs. RNA Biol 2011 ; 8 : 968–977. [CrossRef] [PubMed] [Google Scholar]
- Salmena L, Poliseno L, Tay Y, et al. A ceRNA hypothesis: the Rosetta Stone of a hidden RNA language?. Cell 2011 ; 146 : 353–358. [CrossRef] [PubMed] [Google Scholar]
- Orom UA, Derrien T, Beringer M, et al. Long noncoding RNAs with enhancer-like function in human cells. Cell 2010 ; 143 : 46–58. [CrossRef] [PubMed] [Google Scholar]
- Lam MT, Li W, Rosenfeld MG, Glass CK. Enhancer RNAs and regulated transcriptional programs. Trends Biochem Sci 2014 ; 39 : 170–182. [CrossRef] [PubMed] [Google Scholar]
- Xie W, Ren B. Developmental biology. Enhancing pluripotency and lineage specification. Science 2013 ; 341 : 245–247. [Google Scholar]
- Kim TK, Hemberg M, Gray JM, et al. Widespread transcription at neuronal activity-regulated enhancers. Nature 2010 ; 465 : 182–187. [CrossRef] [PubMed] [Google Scholar]
- Heintzman ND, Hon GC, Hawkins RD, et al. Histone modifications at human enhancers reflect global cell-type-specific gene expression. Nature 2009 ; 459 : 108–112. [CrossRef] [PubMed] [Google Scholar]
- Kaikkonen MU, Spann NJ, Heinz S, et al. Remodeling of the enhancer landscape during macrophage activation is coupled to enhancer transcription. Mol Cell 2013 ; 51 : 310–325. [CrossRef] [PubMed] [Google Scholar]
- Lin YC, Benner C, Mansson R, et al. Global changes in the nuclear positioning of genes and intra- and interdomain genomic interactions that orchestrate B cell fate. Nat Immunol 2012 ; 13 : 1196–1204. [CrossRef] [PubMed] [Google Scholar]
- Sanyal A, Lajoie BR, Jain G, Dekker J. The long-range interaction landscape of gene promoters. Nature 2012 ; 489 : 109–113. [CrossRef] [PubMed] [Google Scholar]
- Zhang Y, Wong CH, Birnbaum RY, et al. Chromatin connectivity maps reveal dynamic promoter-enhancer long-range associations. Nature 2013 ; 504 : 306–310. [CrossRef] [PubMed] [Google Scholar]
- Li W, Notani D, Ma Q, et al. Functional roles of enhancer RNAs for oestrogen-dependent transcriptional activation. Nature 2013 ; 498 : 516–520. [CrossRef] [PubMed] [Google Scholar]
- Maurano MT, Humbert R, Rynes E, et al. Systematic localization of common disease-associated variation in regulatory DNA. Science 2012 ; 337 : 1190–1195. [CrossRef] [PubMed] [Google Scholar]
- Vernot B, Stergachis AB, Maurano MT, et al. Personal and population genomics of human regulatory variation. Genome Res 2012 ; 22 : 1689–1697. [CrossRef] [PubMed] [Google Scholar]
- Hnisz D, Abraham BJ, Lee TI, et al. Super-enhancers in the control of cell identity and disease. Cell 2013 ; 155 : 934–947. [CrossRef] [PubMed] [Google Scholar]
- Klattenhoff CA, Scheuermann JC, Surface LE, et al. Braveheart, a long noncoding RNA required for cardiovascular lineage commitment. Cell 2013 ; 152 : 570–583. [CrossRef] [PubMed] [Google Scholar]
- Grote P, Wittler L, Hendrix D, et al. The tissue-specific lncRNA Fendrr is an essential regulator of heart and body wall development in the mouse. Dev Cell 2013 ; 24 : 206–214. [CrossRef] [PubMed] [Google Scholar]
- Korostowski L, Sedlak N, Engel N. The Kcnq1ot1 long non-coding RNA affects chromatin conformation and expression of Kcnq1, but does not regulate its imprinting in the developing heart. PLoS Genet 2012 ; 8 : e1002956. [CrossRef] [PubMed] [Google Scholar]
- Wang K, Liu F, Zhou LY, et al. The long noncoding RNA CHRF regulates cardiac hypertrophy by targeting miR-489. Circ Res 2014 ; 114 : 1377–1388. [CrossRef] [PubMed] [Google Scholar]
- Han P, Li W, Lin CH, et al. A long noncoding RNA protects the heart from pathological hypertrophy. Nature 2014 ; doi : 10.1038/nature13596 [Google Scholar]
- Yang KC, Yamada KA, Patel AY, et al. Deep RNA sequencing reveals dynamic regulation of myocardial noncoding RNAs in failing human heart and remodeling with mechanical circulatory support. Circulation 2014 ; 129 : 1009–1021. [CrossRef] [PubMed] [Google Scholar]
- Kumarswamy R, Bauters C, Volkmann I, et al. Circulating long noncoding RNA, LIPCAR, predicts survival in patients with heart failure. Circ Res 2014 ; 114 : 1569–1575. [CrossRef] [PubMed] [Google Scholar]
- Ounzain S, Micheletti R, Beckmann T, et al. Genome-wide profiling of the cardiac transcriptome after myocardial infarction identifies novel heart-specific long non-coding RNAs. Eur Heart J 2014 ; doi : 10.1093/eurheartj/ehu180 [Google Scholar]
- Ounzain S, Pezzuto I, Micheletti R, et al. Functional importance of cardiac enhancer-associated noncoding RNAs in heart development and disease. J Mol Cell Cardiol 2014; 76C : 55–70. [CrossRef] [Google Scholar]
- Matkovich SJ, Edwards JR, Grossenheider TC, et al. Epigenetic coordination of embryonic heart transcription by dynamically regulated long noncoding RNAs. Proc Natl Acad Sci USA 2014 ; 111 : 12264–12269. [CrossRef] [Google Scholar]
- Zangrando J, Zhang L, Vausort M, et al. Identification of candidate long non-coding RNAs in response to myocardial infarction. BMC Genomics 2014 ; 15 : 460. [CrossRef] [PubMed] [Google Scholar]
- Vausort M, Wagner DR, Devaux Y. Long non-coding RNAs in patients with acute myocardial infarction. Circ Res 2014 ; 115 : 668–677. [CrossRef] [PubMed] [Google Scholar]
- Wamstad JA, Wang X, Demuren OO, Boyer LA. Distal enhancers: new insights into heart development and disease. Trends Cell Biol 2014 ; 24 : 294–302. [CrossRef] [PubMed] [Google Scholar]
- Abel Y, Clerget G, Bourguignon-Igel V, et al. Les petits ARN nucléolaires nous surprennent encore !. Med Sci (Paris) 2014 ; 30 : 297–302. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Mathieu EL, Belhocine M, Dao LTM, et al. Rôle des longs ARN non codants dans le développement normal et pathologique. Med Sci (Paris) 2014 ; 30 : 790–796. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Pasmant E, Laurendeau I, Sabbagh B, et al. ANRIL ou l’étrange histoire d’un grand ARN non codant. Med Sci (Paris) 2010 ; 26 : 564–566. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1. Rôles des longs ARN non codants. Les longs ARN non codants agissent à travers divers mécanismes pour réguler l’expression des gènes codants. A. Ils dirigent des modificateurs de la chromatine vers des gènes cibles spécifiques. B. Ils déplacent des régulateurs de l’expression (activateurs ou répresseurs) des séquences régulatrices de l’expression. C. Ils entrent en compétition avec des microARN et protègent des ARN messagers de la dégradation par ces microARN. D. Ils modulent des évènements d’épissage et interfèrent avec la traduction. E. Ils promeuvent l’association des séquences amplificatrices et promotrices pour initier la transcription. La Figure décrit une activation en cis, mais l’association peut aussi s’effectuer avec une région du génome non adjacente pour activer un gène en trans. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




