| Issue |
Med Sci (Paris)
Volume 30, Number 5, Mai 2014
|
|
|---|---|---|
| Page(s) | 503 - 505 | |
| Section | Nouvelles | |
| DOI | https://doi.org/10.1051/medsci/20143005009 | |
| Published online | 13 June 2014 | |
Suivi à la trace du parcours intracellulaire de l’adénovirus au cours des étapes précoces du cycle infectieux
Tracking the adenovirus intracellular route during the early steps of the infectious cycle
Centre international de recherche en infectiologie (CIRI) Inserm U1111, CNRS UMR5308, équipe nucléovir, 46, allée d’Italie, 69007
Lyon, France
La majorité des virus à ADN, qu’ils soient enveloppés ou pas, se répliquent dans le noyau. Ceci implique qu’après pénétration dans la cellule, la capside du virus doit être à même de se diriger rapidement vers le noyau pour y déposer le génome viral. Ce cheminement de la capside à travers le cytoplasme constitue une étape critique du cycle infectieux de ces virus, car c’est au cours de ce voyage que la particule subit des étapes de maturation essentielles à son désassemblage ainsi qu’à la libération de l’ADN viral (ADNv) (ou uncoating en anglais) sans pour autant l’exposer trop précocement aux systèmes cellulaires innés de détection de l’ADNv.
L’adénovirus, un modèle pour l’étude des phases précoces du cycle infectieux des virus à ADN non enveloppés, à réplication nucléaire
Parmi les virus à ADN, l’adénovirus (AdV), comme le virus de l’herpès simplex de type 1 (HSV-1), délivre son génome ADN dans le noyau de la cellule hôte. Urs Greber, de l’université de Zurich, fait partie de ces chercheurs qui, avec son équipe, s’emploient depuis de nombreuses années et avec une précision d’orfèvre à décortiquer toutes les étapes précoces du cycle infectieux de l’AdV, depuis sa fixation à son récepteur jusqu’à l’entrée de l’ADNv dans le noyau. Les travaux antérieurs d’Urs Greber ont largement contribué à l’élucidation de plusieurs étapes de ce cycle en montrant qu’après attachement à son récepteur, l’AdV est internalisé par endocytose dans des vésicules recouvertes de clathrine, puis s’échappe rapidement dans le cytosol à partir des endosomes précoces [1] pour être transporté vers le noyau le long des câbles de microtubules par l’intermédiaire de la dynéine et de la dynactine [2, 3]. Au cours de ce transport, la capside de l’AdV subit la perte de quelques protéines structurales tout en maintenant l’ADN viral séquestré à l’intérieur de la particule. La libération de l’ADNv ne surviendrait qu’au moment où la capside se fixe aux complexes protéiques des pores nucléaires (NPC), grâce à l’action de la kinésine-1 qui en induit le désassemblage ainsi qu’un remodelage des NPC et une perméabilisation partielle de la membrane nucléaire [4]. Ce mécanisme permettrait d’éviter la reconnaissance de l’ADNv par les senseurs cytoplasmiques qui participent à la réponse antivirale innée.
L’apport de l’imagerie à l’étude du cycle infectieux viral
Afin d’approfondir la compréhension de ces étapes et, en particulier, d’identifier le moment exact à partir duquel l’ADNv se sépare de la capside, il a fallu développer des techniques qui permettent de suivre à la fois l’ADNv et les protéines structurales de l’AdV, et qui soient suffisamment sensibles pour être en mesure de quantifier la présence de ces deux composants dans différents compartiments cellulaires. La méthode couramment utilisée pour ce genre d’investigation repose sur la détection des protéines par immunofluorescence (IF) et de l’ADNv par hybridation in situ à l’aide de sondes marquées (FISH). Cependant, l’étape d’hybridation des acides nucléiques dénature les protéines et en empêche fréquemment la détection par des anticorps. D’autres techniques reposent sur l’insertion dans l’ADNv de plusieurs motifs pouvant être reconnus par des protéines chimériques fluorescentes, tels que ceux qui dérivent de l’opéron LacO sur lequel se fixe le répresseur protéique LacI [5]. Bien que cette technique permette de visualiser l’ADN dans des cellules vivantes, elle comporte aussi de nombreux désavantages. Notamment, plusieurs copies de séquences LacO doivent être introduites pour obtenir un signal différent du bruit de fond, et les cellules infectées doivent être au préalable génétiquement modifiées pour produire une protéine LacI fluorescente.
Pour surmonter ces obstacles, l’équipe d’Urs Greber a utilisé une technique qui consiste à incorporer dans l’ADNv des analogues de nucléosides couplés à un groupe éthynyl, pouvant être détectés grâce à une réaction chimique Click (une cycloaddition 1,3-dipolaire catalysée par le cuivre (I) entre un azoture et un alcyne) réalisée directement dans les cellules fixées [6]. Cette technologie, qui a été développée pour marquer l’ADN cellulaire, repose généralement sur l’incorporation dans l’ADN du 5-éthynyl-2´-déoxyuridine (EdU), produit commercialement disponible, qui est simplement ajouté au milieu de culture. En suivant le même principe, l’équipe d’Urs Greber a analysé en détail l’incorporation de différents analogues de nucléosides (EdU, EdA, et EdC) dans l’ADNv de l’AdV, l’HSV-1 et le virus de la vaccine, au cours de leur production dans des cellules infectées, ainsi que ses conséquences fonctionnelles sur l’efficacité de la réplication virale. Dans le cas de l’AdV, seuls deux analogues, l’EdC et l’EdA, permettent de marquer de façon efficace l’ADNv sans compromettre sa capacité à se répliquer [7]. Cette spécificité dépend du virus utilisé puisque d’autres virus, comme l’HSV-1 ou le virus de la vaccine, requièrent d’autres analogues nucléotidiques. Cette méthode, couplée à une visualisation par microscopie confocale, permet d’obtenir des images plus précises et résolutives que celles qui sont obtenues par FISH (fluorescent in situ hybridation) (Figure 1). De plus, cette technique permet aussi une codétection des protéines par immunofluorescence. Ainsi, le marquage de l’ADNv de l’AdV par de l’EdC, suivi de la détection d’une des protéines virales essentielles à sa réplication, la DNA-binding protein (DBP), permet de visualiser ces deux composants viraux dans le noyau cellulaire avec une précision surprenante, et révèle une organisation insoupçonnée des centres de réplication virale, avec la DBP formant un anneau entouré par deux cercles concentriques d’ADNv (Figure 2). Cette image suggère une organisation complexe de ces corps nucléaires d’origine virale.
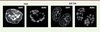 |
Figure 1. Comparaison des méthodes de FISH et Click pour la détection de l’ADN de l’AdV de type 5. L’ADN de l’AdV de type 5 a été recherché lors de sa réplication dans le noyau de cellules humaines, 16 heures post-infection. Les panneaux montrent les images obtenues après coloration de l’ADN cellulaire par le 4’,6-diamidino-2-phenylindole (DAPI, en bleu) et détection de l’ADNv soit par hybridation en utilisant des sondes marquées spécifiques, soit après incorporation d’EdC et réaction Click (Source : I-Hsuan Wang, Maarit Suomalainen and Urs F. Greber, Institute of Molecular Life Sciences, Université de Zurich, Suisse). |
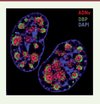 |
Figure 2. Marquage par IF et réaction Click des centres de réplication nucléaires de l’AdV de type 5. La réaction a été faite 16 heures après l’infection de cellules humaines. Le marquage de l’ADNv a été réalisé après incorporation d’EdC et visualisé avec de l’Alexa-Fluor® 594 azide (en rouge). La protéine virale DBP a été détectée par IF (en vert). L’ADN cellulaire est marqué au DAPI (en bleu). (Source : I-Hsuan Wang, Maarit Suomalainen and Urs F. Greber, Institute of Molecular Life Sciences, Université de Zurich, Suisse). |
Suivi des particules d’adénovirus lors de leur cheminement vers le noyau
Dans la suite de l’étude, l’équipe a cherché à détecter et suivre dans des cellules infectées des particules d’AdV produites en présence d’EdA + EdC puis purifiées. Les résultats, surprenants, montrent qu’effectivement la détection des génomes peut se faire si les capsides ont été partiellement désassemblées (pour permettre la réaction Click), événement qui se produit naturellement dans la cellule infectée lorsque les capsides sortent des endosomes précoces. La détection de l’ADNv, couplée au marquage d’une protéine de la capside par immunofluorescence, permet de préciser les événements survenant lors des étapes précoces de l’infection par l’AdV. Notamment, en couplant cette technique de marquage de l’ADNv avec une méthode précédemment mise au point pour quantifier les particules d’AdV échappées des endosomes [8], l’équipe a mis en évidence un phénomène précédemment insoupçonné : la présence de l’ADNv nu, non protégé par la capside, dans le cytoplasme. Ce phénomène n’est pas observable après traitement des cellules par la leptomycine B, un inhibiteur des mécanismes d’export via les pores nucléaires qui empêche le désassemblage complet de la capside de l’AdV. Cela suggère que la séparation de l’ADN viral de la capside, précédemment décrite comme survenant lors de l’interaction de la capside avec le NPC et aboutissant à l’internalisation de l’ADNv dans le noyau, pourrait aussi induire le relargage d’ADN dans le cytosol. La présence d’ADNv dans le cytosol pourrait être à l’origine de sa reconnaissance par les senseurs de l’ADN de l’immunité innée, phénomène a priori nuisible à la réplication du virus. Pour expliquer cette observation inattendue, les auteurs suggèrent l’existence d’une réponse antivirale au niveau des pores nucléaires, qui préviendrait l’entrée de l’ADNv dans le noyau. Enfin, cette même étude montre que l’ADNv peut être suivi jusque dans le noyau. Une quantification des molécules d’ADNv détectées dans le noyau de cellules infectées avec différentes doses d’AdV permet de conclure que l’entrée et/ou la déposition des génomes viraux dans le noyau est un phénomène rapidement saturable, et suggère qu’au-delà d’une certaine dose, les génomes viraux seraient dégradés.
Conclusion
Dans l’ensemble, la méthode de marquage de l’ADNv décrite dans cette étude ouvre des perspectives passionnantes pour valider et approfondir les résultats déjà obtenus par des techniques plus classiques de biochimie ou de biologie cellulaire. Dans le cas de l’AdV, les résultats présentés permettent aussi d’approfondir la compréhension des mécanismes d’entrée du virus et de défense antivirale. Grâce à cette méthode, il est maintenant possible de suivre de façon extrêmement précise le devenir intracellulaire de plusieurs composants viraux au cours du cycle infectieux. Malgré le niveau d’expertise élevé requis pour la mise en œuvre de ces techniques, on peut d’emblée prévoir qu’une évolution très rapide des méthodes de marquage et de détection des génomes viraux, notamment par de la microscopie à haute résolution, nous permettra de révéler de nouveaux aspects insoupçonnés du cycle infectieux de nombreux autres virus.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- Gastaldelli M, Imelli N, Boucke K, et al. Infectious adenovirus type 2 transport through early but not late endosomes. Traffic 2008 ; 9 : 2265–2278. [CrossRef] [PubMed] [Google Scholar]
- Engelke MF, Burckhardt CJ, Morf MK, Greber UF. The dynactin complex enhances the speed of microtubule-dependent motions of adenovirus both towards and away from the nucleus. Viruses 2011 ; 3 : 233–253. [CrossRef] [PubMed] [Google Scholar]
- Bremner KH, Scherer J, Yi J, et al. Adenovirus transport via direct interaction of cytoplasmic dynein with the viral capsid hexon subunit. Cell Host Microbe 2009 ; 6 : 523–535. [CrossRef] [PubMed] [Google Scholar]
- Strunze S, Engelke MF, Wang IH, et al. Kinesin-1-mediated capsid disassembly and disruption of the nuclear pore complex promote virus infection. Cell Host Microbe 2011 ; 10 : 210–223. [CrossRef] [PubMed] [Google Scholar]
- Tsukamoto T, Hashiguchi N, Janicki SM, et al. Visualization of gene activity in living cells. Nat Cell Biol 2000 ; 2 : 871–878. [CrossRef] [PubMed] [Google Scholar]
- Salic A, Mitchison TJ. A chemical method for fast and sensitive detection of DNA synthesis in vivo. Proc Natl Acad Sci USA 2008 ; 105 : 2415–2420. [CrossRef] [Google Scholar]
- Wang IH, Suomalainen M, Andriasyan V, et al. Tracking viral genomes in host cells at single-molecule resolution. Cell Host Microbe 2013 ; 14 : 468–480. [CrossRef] [PubMed] [Google Scholar]
- Suomalainen M, Luisoni S, Boucke K, et al. A direct and versatile assay measuring membrane penetration of adenovirus in single cells. J Virol 2013 ; 87 : 12367–12379. [CrossRef] [PubMed] [Google Scholar]
© 2014 médecine/sciences – Inserm
Liste des figures
 |
Figure 1. Comparaison des méthodes de FISH et Click pour la détection de l’ADN de l’AdV de type 5. L’ADN de l’AdV de type 5 a été recherché lors de sa réplication dans le noyau de cellules humaines, 16 heures post-infection. Les panneaux montrent les images obtenues après coloration de l’ADN cellulaire par le 4’,6-diamidino-2-phenylindole (DAPI, en bleu) et détection de l’ADNv soit par hybridation en utilisant des sondes marquées spécifiques, soit après incorporation d’EdC et réaction Click (Source : I-Hsuan Wang, Maarit Suomalainen and Urs F. Greber, Institute of Molecular Life Sciences, Université de Zurich, Suisse). |
| Dans le texte | |
 |
Figure 2. Marquage par IF et réaction Click des centres de réplication nucléaires de l’AdV de type 5. La réaction a été faite 16 heures après l’infection de cellules humaines. Le marquage de l’ADNv a été réalisé après incorporation d’EdC et visualisé avec de l’Alexa-Fluor® 594 azide (en rouge). La protéine virale DBP a été détectée par IF (en vert). L’ADN cellulaire est marqué au DAPI (en bleu). (Source : I-Hsuan Wang, Maarit Suomalainen and Urs F. Greber, Institute of Molecular Life Sciences, Université de Zurich, Suisse). |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




