| Issue |
Med Sci (Paris)
Volume 30, Number 3, Mars 2014
|
|
|---|---|---|
| Page(s) | 329 - 331 | |
| Section | Dernière heure | |
| DOI | https://doi.org/10.1051/medsci/20143003022 | |
| Published online | 31 March 2014 | |
Pithovirus sibericum : réveil d’un virus géant de plus de 30 000 ans
Pithovirus sibericum: awakening of a giant virus of more than 30 000 years
Information génomique et structurale IGS (UMR7256), CNRS, Aix-Marseille Université, Institut de microbiologie de la Méditerranée-IMM-FR 3479, parc scientifique de Luminy, 163, avenue de Luminy, case 934, 13288 Marseille Cedex 09, France
Des virus aussi complexes que des bactéries
La découverte de Mimivirus il y plus de 10 ans a bouleversé notre conception des virus [1]. En effet, les virus étaient considérés comme des pathogènes de très petites dimensions, ayant peu de gènes, pourtant capables de détourner la machinerie de la cellule qu’ils infectent à leur profit, déclenchant ainsi des dérèglements cellulaires pouvant aller jusqu’à la destruction totale des cellules. Les virus sont différents des cellules et il n’existe pas d’intermédiaire entre le monde viral et le monde cellulaire [2]. Concrètement, ils correspondent à la fraction non retenue par les filtres de stérilisation de porosité de l’ordre de 0,2 µm. Mimivirus, premier virus visible au microscope optique, rivalise avec le monde bactérien par la taille de sa particule, de l’ordre d’un demi-micromètre, et surtout par la taille et la complexité de son génome de plus d’un million de paires de bases, codant pour un millier de protéines dont certaines que l’on croyait réservées au monde cellulaire [3]. Cependant, Mimivirus, par son contenu en gènes, reste un virus classique présentant une affinité phylogénétique avec les virus appartenant à la grande famille des grands virus à ADN nucléocytoplasmiques. Cette famille de grands virus comprend les Pox-, les Irido-, les Asfarvirus, pathogènes de vertébrés et d’invertébrés isolés dans le contexte de différentes maladies, et les phycoDNAvirus, infectant des algues. Ils présentent tous des niveaux d’autonomie différents par rapport à la cellule qu’ils infectent. Certains, comme les phycoDNAvirus, nécessitent absolument la présence du noyau cellulaire et de la machinerie de leur hôte pour transcrire leurs gènes. Les Iridovirus utilisent le noyau cellulaire mais possèdent également une machinerie leur permettant d’exprimer certains de leurs gènes dans le cytoplasme, dans ce que l’on appelle des usines à virions. Enfin, les Asfarvirus, les Poxvirus et les Mimivirus apportent leur machinerie transcriptionnelle en même temps que leur génome au moment de l’infection, ce qui leur permet de se répliquer dans des usines à virions cytoplasmiques. Avec Mimivirus, seulement capable d’infecter des amibes du genre Acanthamoeba, pour la première fois l’intérêt scientifique des virus n’est plus cantonné aux maladies qu’ils peuvent causer. D’autres virus aussi grands et complexes que Mimivirus ont par la suite été recherchés et isolés dans l’environnement [4–8], montrant ainsi qu’ils étaient loin d’être rares et avaient probablement été occultés en raison de leur trop grande taille et des techniques utilisées pour isoler les virus des échantillons environnementaux. On compte aujourd’hui une dizaine de ces virus géants à ADN, maintenant référencés comme les Mimiviridae.
Les Pandoravirus : des géants parmi les géants
Alors que l’on commençait à penser que les virus géants appartiendraient tous à cette même grande famille de virus icosaédriques, est survenue la découverte des Pandoravirus, isolés d’abord au Chili dans le Pacifique, puis en Australie dans un étang d’eau douce [9]. Les tailles de ces virus et leurs génomes rivalisaient cette fois avec ceux du monde des eucaryotes. Les Pandoravirus ont des génomes de 2 Mb et plus, et codent pour 1 500 à 2 550 protéines, dans une particule en forme d’amphore de 1 µm de long pour 0,5 µm de diamètre. Fait encore plus marquant, seules 6 % de leurs protéines ont des homologues clairs dans les bases de données, incluant les données de métagénomique. Ces géants parmi les géants sont pourtant dépourvus de la machinerie de transcription nécessaire à l’expression de leurs gènes, et doivent utiliser celle du noyau de la cellule qu’ils infectent. Chose rare chez les virus, 10 % de leurs gènes comportent des introns qui requièrent la machinerie d’épissage nucléaire pour la maturation des transcrits. Ainsi, malgré une complexité génomique deux fois supérieure à celle des Mimiviridae, les Pandoravirus requièrent une participation de la cellule hôte plus importante pour leur réplication. Enfin, ils ne présentent aucune affinité phylogénétique avec les grands virus à ADN nucléocytoplasmiques. Ces virus occultés pendant des années ne semblent pourtant pas rares comme l’atteste leur présence dans des lieux aussi éloigné que l’Amérique et l’Australie. Leur présence en Europe est également suggérée par le Pr. Rolf Michel dans des publications qui montrent des parasites d’amibes étonnamment semblables aux Pandoravirus [10, 11].
À ce stade, on pouvait donc penser qu’il existait deux types de virus, les virus icosahédriques probablement dérivés d’un ancêtre commun, et les Pandoravirus dont l’histoire évolutive est différente. Ces résultats suggéraient également que l’émergence de la vie cellulaire ait pu s’accompagner d’une diversité beaucoup plus foisonnante de formes pré-cellulaires que celles envisagées classiquement.
Pithovirus sibericum, « retour vers le futur »
C’est dans ce contexte que surgit la découverte de Pithovirus sibericum dont la particule présente une morphologie ovoïde évoquant celles des Pandoravirus [12]. Nous nous attendions donc à ce qu’il s’agisse d’un autre Pandoravirus jusqu’à ce que l’analyse de son génome à ADN révèle qu’il ne présentait aucun lien phylogénétique avec les Pandoravirus. Son génome, comme celui des Mimiviridae, est riche en adénine + thymine (64 %), alors que celui des Pandoravirus est riche en guanine + cytosine (> 61 %). Le génome de Pithovirus, bien que de 600 kb, est étonnamment plus petit que celui des Pandoravirus. Il se place seulement en huitième position parmi les plus grands virus à ADN, et pourtant sa particule virale est la plus grosse qui soit référencée, d’une longueur de 1,5 µm pour un diamètre de 0,5 µm. Celle-ci apparaît d’ailleurs très peu dense à l’exception, comme dans le cas des Pandoravirus, d’une sphère de 50 nm de diamètre dont on ne connaît pas encore la composition. Son génome ne code que pour 467 protéines dont les deux tiers correspondent à des séquences ne présentant aucune homologie avec celles présentes dans les bases de données. Dans le dernier tiers, on trouve autant de protéines homologues de protéines d’eucaryotes, que de protéines de bactéries ou de virus. L’analyse phylogénétique des quelque cinquante protéines homologues aux protéines virales montre que celles-ci présentent une affinité claire pour les Iridovirus, les Marseillevirus et les Mimivirus, tous icosaédriques. On a donc affaire à un virus ressemblant plus à la famille des grands virus à ADN qu’aux Pandoravirus, avec pourtant une forme de particule en amphore que l’on croyait réservée aux Pandoravirus. De plus, alors que les Pandoravirus requièrent un passage par le noyau cellulaire, Pithovirus reste cytoplasmique et utilise sa propre machinerie de transcription.
La comparaison détaillée des structures et du contenu en protéines de Pithovirus révèle des différences marquées avec les Pandoravirus (Figure 1). Tout d’abord, le protéome des particules purifiées ne révèle aucune protéine en commun avec les Pandoravirus. Ensuite, alors que les particules des Pandoravirus sont entourées d’un tégument de 60 nm composé de trois couches d’épaisseurs équivalentes, dont une couche intermédiaire lamellaire, l’enveloppe des Pithovirus, également de 60 nm, est très dense aux électrons et semble composée d’une seule couche faite de stries parallèles. Le bouchon présent à l’apex des particules des Pandoravirus est diffus et difficile à visualiser, tandis que celui de Pithovirus, mieux défini, est saillant avec une structure hexagonale spectaculaire en nid d’abeille. Une membrane est présente sous l’enveloppe des deux types de particules, mais cette membrane dans les particules de Pithovirus se termine sous forme d’une bobine qui semble connectée au bouchon. Les particules de Pithovirus, comme celles de tous les virus infectant les amibes du genre Acanthamoeba, sont phagocytées, et l’infection débute par l’ouverture de la particule et la fusion de la membrane interne avec celle de la vacuole permettant ainsi le transfert de tout ce qui se trouve dans les particules dans le cytoplasme de la cellule infectée.
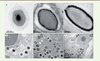 |
Figure 1. Exemples de virus géants observés au microsocope électronique à transmission. A. Megavirus chilensis ; B. Pandoravirus salinus ; C. Pithovirus sibericum ; D. Usine à virions (VF) d’où bourgeonnent les capsides de Mégavirus avant d’être chargées en ADN puis entourées de leur couche de fibrilles. E. Particules de Pandoravirus en cours d’assemblage. Des particu les matures sont également visibles. F. Particules de Pithovirus en cours de formation à la périphérie de l’usine à virions. Le noyau de la cellule (N) reste intact même pour les temps les plus tardifs. |
Pithovirus sibericum, un virus ressuscité à partir d’un échantillon environnemental Sibérien datant de plus de 30 000 ans, est ainsi un nouveau type de virus partageant la morphologie des Pandoravirus tout en présentant une certaine affinité avec la famille plus classique des grands virus à ADN nucléocytoplasmique. Cette découverte suggère que le véhicule en forme d’amphore partagé par les Pandoravirus et Pithovirus pourrait être en fait utilisé pour véhiculer des génomes de tailles et de compositions (ADN simple brin, ADN double brin, ARN) aussi variées que ceux du véhicule icosaédrique.
Le même groupe Allemand du Pr R. Michel a également publié il y a une dizaine d’années la description d’un parasite d’amibe de morphologie identique à celle de Pithovirus [13]. Ce virus pourrait donc encore exister de nos jours mais ne pas avoir été reconnu comme un virus en raison de sa morphologie atypique.
Enfin, cette étude soulève un problème sociétal. À l’ère du réchauffement climatique, si Pithovirus a pu être réactivé par simple décongélation d’un fragment de permafrost vieux de 30 000 ans, c’est également vrai des autres virus actuellement présents dans les couches de permafrost en cours de dégel ou d’exploitations industrielles actives. Pithovirus n’est pas un pathogène pour l’homme, mais qu’en est-il de tous les autres virus éventuellement présents dans les couches plus profondes de permafrost qui seront mises au jour par l’exploitation industrielle de l’arctique ?
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- La Scola B, Audic S, Robert C, et al. A giant virus in amoebae. Science 2003 ; 299 : 2033. [CrossRef] [PubMed] [Google Scholar]
- Lwoff A. The concept of virus. J Gen Microbiol 1957 ; 17 : 239–253. [CrossRef] [PubMed] [Google Scholar]
- Raoult D, Audic S, Robert C, et al. The 1.2-megabase genome sequence of Mimivirus. Science 2004 ; 306 : 1344–1350. [CrossRef] [PubMed] [Google Scholar]
- Arslan D, Legendre M, Seltzer V, et al. Distant Mimivirus relative with a larger genome highlights the fundamental features of Megaviridae. Proc Natl Acad Sci USA 2011 ; 108 : 17486–17491. [CrossRef] [Google Scholar]
- La Scola B, Campocasso A, N’Dong R, et al. Tentative characterization of new environmental giant viruses by MALDI-TOF mass spectrometry. Intervirology 2010 ; 53 : 344–353. [CrossRef] [PubMed] [Google Scholar]
- Colson P, Yutin N, Shabalina SA, et al. Viruses with more than 1, 000 genes: Mamavirus, a new Acanthamoeba polyphaga mimivirus strain, and reannotation of Mimivirus genes. Genome Biol Evol 2011 ; 3 : 737–742. [CrossRef] [PubMed] [Google Scholar]
- Yoosuf N, Yutin N, Colson P, et al. Related giant viruses in distant locations and different habitats: Acanthamoeba polyphaga Moumouvirus represents a third lineage of the Mimiviridae that is close to the megavirus lineage. Genome Biol Evol 2012 ; 4 : 1324–1330. [CrossRef] [PubMed] [Google Scholar]
- Boughalmi M, Saadi H, Pagnier I, et al. High-throughput isolation of giant viruses of the Mimiviridae and Marseilleviridae families in the Tunisian environment. Environ Microbiol 2013 ; 15 : 2000–2007. [CrossRef] [PubMed] [Google Scholar]
- Philippe N, Legendre M, Doutre G, et al. Pandoraviruses: amoeba viruses with genomes up to 2.5 Mb reaching that of parasitic eukaryotes. Science 2013 ; 341 : 281–286. [CrossRef] [PubMed] [Google Scholar]
- Scheid P, Zöller L, Pressmar S, et al. An extraordinary endocytobiont in Acanthamoeba sp. isolated from a patient with keratitis. Parasitol Res 2008 ; 102 : 945–950. [CrossRef] [PubMed] [Google Scholar]
- Scheid P, Hauröder B, Michel R. Investigations of an extraordinary endocytobiont in Acanthamoeba sp.: development and replication. Parasitol Res 2010 ; 106 : 1371–1377. [CrossRef] [PubMed] [Google Scholar]
- Legendre M, Bartoli J, Shmakova L, et al. Thirty-thousand-year-old distant relative of giant icosahedral DNA viruses with a pandoravirus morphology. Proc Natl Acad Sci USA 2014 (souspresse). [Google Scholar]
- Michel R, Schmid EN, Hoffmann R, Müller KD. Endoparasite KC5/2 encloses large areas of sol-like cytoplasm within Acanthamoebae. Normal behavior or aberration? Parasitol Res 2003 ; 91 : 265–266. [CrossRef] [PubMed] [Google Scholar]
© 2014 médecine/sciences – Inserm
Liste des figures
 |
Figure 1. Exemples de virus géants observés au microsocope électronique à transmission. A. Megavirus chilensis ; B. Pandoravirus salinus ; C. Pithovirus sibericum ; D. Usine à virions (VF) d’où bourgeonnent les capsides de Mégavirus avant d’être chargées en ADN puis entourées de leur couche de fibrilles. E. Particules de Pandoravirus en cours d’assemblage. Des particu les matures sont également visibles. F. Particules de Pithovirus en cours de formation à la périphérie de l’usine à virions. Le noyau de la cellule (N) reste intact même pour les temps les plus tardifs. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




