| Issue |
Med Sci (Paris)
Volume 30, Number 1, Janvier 2014
|
|
|---|---|---|
| Page(s) | 21 - 24 | |
| Section | Nouvelles | |
| DOI | https://doi.org/10.1051/medsci/20143001006 | |
| Published online | 24 January 2014 | |
Le ribosome
Un nouvel acteur de la tumorigenèse ?
The ribosome: a new player in tumorigenesis?
1
Centre de recherche en cancérologie de Lyon, UMR Inserm 1052 CNRS 5286, Centre Léon Bérard, 28, rue Laennec, F-69373, Lyon, France
2
Université de Lyon, F-69003, Lyon, France
3
Université Lyon 1, ISPB, Lyon, F-69622, France, F-69000 Lyon, France
Identifiés dans les années 1950, les ribosomes sont des complexes ribo-nucléoprotéiques qui traduisent les ARN messagers (ARNm) en protéines. Depuis, d’importants efforts ont été consacrés à déterminer la structure tridimensionnelle des ribosomes. Cette structure a d’abord été obtenue pour les ribosomes procaryotes et a été récompensée par le prix Nobel de chimie en 2009 (V. Ramakrishnan, T.A. Steitz et A.E. Yonath, voir [1]). Nous disposons aujourd’hui des structures atomiques des ribosomes de nombreux organismes procaryotes et eucaryotes, y compris humain, et d’une première vision de la dynamique du mécanisme moléculaire de lecture du code génétique et de synthèse des protéines.
Ces dernières années ont également vu l’émergence de caractéristiques inattendues des ribosomes, qui ouvrent de nouvelles perspectives dans la compréhension de leur rôle biologique. En effet, les ribosomes montrent une certaine hétérogénéité de composition selon les cellules, et participent directement à la régulation de la synthèse protéique. Le ribosome serait une « plate-forme de régulation » intervenant dans l’efficacité, la spécificité et la fidélité de traduction, ainsi que dans les modifications réversibles et irréversibles des protéines. La diversité de composition et la dynamique de structure du ribosome seraient à l’origine de ses multiples fonctions, et, suite à des altérations, favoriserait le développement de pathologies.
Ribosome et régulation de la traduction
Le ribosome humain est composé de 80 protéines et de quatre ARN ribosomiques (ARNr) organisés en deux sous-unités (40S et 60S) [2]. Le cœur du ribosome, qui comporte les sites catalytiques (centre de décodage et centre de transfert de la liaison peptidique), présente une structure quasi-identique de la bactérie à l’homme. L’activité catalytique du ribosome est portée par les ARNr, qui, en association avec les protéines ribosomiques, vérifient l’appariement codon : anti-codon et assurent la formation de la liaison peptidique. Les modifications chimiques des ARNr influencent leurs structures, les interactions ARN : ARN et ARN : protéines, et contrôlent ainsi leurs fonctions. À ce jour, 95 pseudo-uridylations et 105 méthylations de ribose ont été identifiées sur les ARNr qui sont respectivement catalysées par la dyskérine et la fibrillarine (FBL).
La régulation de la traduction s’effectue essentiellement lors de la phase d’initiation qui fait intervenir de nombreux facteurs (eIF) qui jouent le rôle d’activateurs et d’adaptateurs entre les éléments de la machinerie de traduction et l’ARNm (Figure 1) [3]. La sélectivité des mécanismes de régulation implique des éléments dits cis-régulateurs, localisés dans les régions non traduites des ARNm, tels que des séquences permettant la fixation de protéines ou de microARN. Par ailleurs, il existe deux modes d’initiation de la traduction : dans le mode dépendant de la coiffe, la sous-unité 40S reconnaît la coiffe à l’extrémité 5’ de l’ARNm, puis parcourt la région 5’ non traduite et débute la traduction à partir d’un codon AUG par recrutement de la sous-unité 60S ; dans le mode indépendant de la coiffe, le ribosome reconnaît l’ARNm par une séquence IRES (internal ribosomal entry sites) et débute la traduction au niveau d’un codon AUG situé après cette séquence. Ce second type d’initiation de la traduction est activé dans des conditions physiopathologiques particulières et concerne un nombre restreint d’ARNm.
 |
Figure 1. Régulation de l’initiation de la traduction. L’ARNm est représenté en noir sur lequel sont montrés les éléments cis-régulateurs : coiffe, structures secondaires (tige-boucle), IRES (internal ribosome entry sites), uORF (upstream open reading frame), séquences de reconnaissance par des miARN et des protéines. Les éléments trans-régulateurs sont schématisés en bleu : eIF (eukaryotic initiation factors), ITAF (IRES trans-acting factors), RNA-BP (RNA-binding proteins), miARN et PABP (polyA binding proteins). Les ribosomes sont présentés en gris avec les sous-unités 40S et 60S. |
Altérations quantitatives et qualitatives de la biogenèse des ribosomes au cours de la tumorigenèse
Le lien entre cancer et altération quantitative des ribosomes est clairement établi depuis plusieurs années. Dans les cellules cancéreuses, la production des ribosomes est anormalement augmentée en réponse à une hyperactivation de l’ARN polymérase I (ARN pol I) résultant de l’activation d’oncogènes (c-myc, ∆N-nétrine-1) ou de l’inactivation de suppresseurs de tumeur (pRb, p53) [4, 5]. Cette altération quantitative des ribosomes favorise la prolifération des cellules cancéreuses, qui nécessite une production rapide et abondante de protéines. Cependant, récemment, a émergé la notion qu’une altération de composition des ribosomes - ou altération qualitative - pouvait être également impliquée dans les cancers. Une susceptibilité accrue au développement de cancers a été observée chez des patients atteints de maladies génétiques appelées ribosomopathies (anémie de Diamond Blackfan, syndrome-5q, syndrome de Shwachman Diamond, dyskératose congénitale) [3, 6–9]. Des variations génétiques entraînent la production de ribosomes dont la composition en protéines ou en pseudo-uridylation des ARNr est modifiée, conduisant à une altération de leur fonction.
Dans ce contexte, notre équipe a été la première à montrer que, dans les cancers, les ribosomes présentent des différences de méthylation des ARNr. Dans un modèle cellulaire de cancer du sein rendu agressif, nous avons observé que le patron de méthylation des ARNr était modifié [10]. Cette observation a été étendue à un modèle mammaire de progression tumorale, dans lequel nous avons observé une augmentation de la méthylation des ARNr en corrélation avec le phénotype tumoral [11]. Notre équipe a décrypté le mécanisme liant le phénotype tumoral et la méthylation des ARNr, et a montré qu’en réponse à l’inactivation de p53, l’expression de la méthyltransférase des ARNr (FBL) et la méthylation des ARNr sont augmentées (Figure 2) [11]. Il s’est avéré que p53 sauvage se lie au gène FBL et réprime sa transcription ; cette répression étant ainsi levée dans les cellules cancéreuses déficientes pour p53.
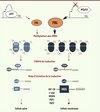 |
Figure 2. Altération qualitative des ribosomes : impact sur la tumorigenèse. L’inactivation de p53 (MTp53) conduit à une surexpression de la fibrillarine (FBL). La quantité anormale de FBL induit une production de ribosomes dont les patrons de méthylation sont altérés. Ces ribosomes modifiés participent à la diminution de la fidélité de traduction et à l’augmentation de la traduction d’ARNm contenant des IRES et codant pour des protéines favorisant la tumorigenèse. |
Nous avons ensuite exploré les conséquences fonctionnelles de ces altérations sur la traduction et le développement tumoral. L’altération de la méthylation des ARNr affecte l’activité intrinsèque du ribosome (diminution de la fidélité de traduction et de la reconnaissance des codons stop) et modifie la sélectivité des ARNm traduits, notamment par une augmentation de l’utilisation des IRES des ARNm de l’IGF-1R (récepteur de l’insulin growth factor-1), c-myc, VEGFA (vascular endothelial growth factor-A), FGF1 (fibroblast growth factor1) et FGF2 [10, 11]. L’importance dans le phénotype tumoral de cette régulation traductionnelle liée à l’altération de la méthylation des ARNr a été démontrée par deux observations. Premièrement, des cellules surexprimant FBL ont une capacité proliférative augmentée et acquièrent la capacité de proliférer sous la forme de colonies sans ancrage (anchorage independent) à un support solide. Deuxièmement, une forte expression de FBL est associée à un mauvais pronostic et à la récurrence de la maladie chez des patientes atteintes d’un cancer du sein. Ces travaux innovants montrent que l’inactivation de gènes clés de la tumorigenèse tels que p53 affecte directement la qualité des ribosomes. Ainsi, au cours de la progression tumorale, ces ribosomes de composition altérée participent directement à la dérégulation de la traduction, et favorisent la progression tumorale.
Les ribosomes : perspectives en cancérologie
L’augmentation de la production de ribosomes est une caractéristique bien connue des cellules cancéreuses, utilisée comme marqueur de l’état malin d’une cellule à travers la morphologie des nucléoles, lieu de leur production. La surexpression de facteurs de la biogenèse des ribosomes, tels que FBL, ne semble pas simplement corréler avec la surproduction de ribosomes, mais apparaît aussi comme un nouveau marqueur indépendant de mauvais pronostic. De plus, l’altération de la qualité des ribosomes a un impact direct sur l’activité intrinsèque de traduction de ces derniers. Bien que la répercussion de la diminution de la fidélité des ribosomes sur le protéome soit difficile à évaluer à ce jour, les conséquences phénotypiques liées au changement de mode d’initiation de la traduction sont clairement établies et reflètent une augmentation de l’expression de protéines oncogéniques clés. Nos travaux apportent ainsi des données moléculaires nouvelles qui soutiennent l’existence d’un changement du mode d’initiation de la traduction, de dépendant à indépendant de la coiffe, au cours de la tumorigenèse, favorisant la traduction d’ARNm codant pour des protéines oncogéniques qui contribuent directement au développement tumoral [4, 10, 11].
La dérégulation de la biogenèse des ribosomes pourrait ainsi être, en plus d’une conséquence, un moteur décisif conférant des avantages sélectifs à la cellule tumorale. Peut-on alors considérer les ribosomes comme une cible potentielle de thérapeutiques anti-cancer ? Une étude récente est en faveur de cette hypothèse, qui montre que l’inhibition de l’ARN Pol I par une petite molécule (CX5461) induit une cytotoxicité sélective des cellules cancéreuses en épargnant les cellules saines dans un modèle leucémique murin [12]. L’ensemble de ces travaux ouvrent des perspectives prometteuses avec l’identification d’une nouvelle catégorie de cibles thérapeutiques en cancérologie : les ribosomes.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt avec les données publiées dans cet article.
Références
- Romby P, Marzi S, Westhof E. La structure atomique du ribosome en pleine lumière. Med Sci (Paris) 2009 ; 25 : 977–981. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Melnikov S, Ben-Shem A, Garreau de Loubresse N, et al. One core, two shells : bacterial and eukaryotic ribosomes. Nat Struct Mol Biol 2012 ; 19 : 560–567. [CrossRef] [PubMed] [Google Scholar]
- Jackson RJ, Hellen CUT, Pestova TV. The mechanism of eukaryotic translation initiation and principles of its regulation. Nat Rev Mol Cell Biol 2010 ; 10 : 113–127. [CrossRef] [Google Scholar]
- Ruggero D., Translational control in cancer etiology. Cold Spring Harb Perspect Biol 2013 ; 5 : a012336. [CrossRef] [PubMed] [Google Scholar]
- Delloye-Bourgeois C, Goldschneider D, Paradisi A, et al. Nucleolar localization of a Netrin-1 isoform enhances tumor cell proliferation. Sci Signal 2012 ; 5 : ra57. [CrossRef] [PubMed] [Google Scholar]
- Aguissa-Touré A, Da Costa, Leblanc T, et al. Anémie de Diamond-Blackfan. Med Sci (Paris) 2009 ; 25 : 69–76. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Hoareau-Aveilla C, Henry Y, Leblanc T. La dyskératose congénitale. Med Sci (Paris) 2008 ; 24 : 390–398. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Xue S, Barna M. Specialized ribosomes : a new frontier in gene regulation and organismal biology. Nat Rev Mol Cell Biol 2012 ; 13 : 355–369. [CrossRef] [PubMed] [Google Scholar]
- Jack K, Bellodi C, Landry DM, et al. rRNA pseudouridylation defects affect ribosomal ligand binding and translational fidelity from yeast to human cells. Mol Cell 2011 ; 44 : 660–666. [CrossRef] [PubMed] [Google Scholar]
- Belin S, Beghin A, Solano-Gonzàlez E, et al. Dysregulation of ribosome biogenesis translational capacity is associated with tumor progression of human breast cancer cells. PLoS One 2009 ; 4 : e7147. [CrossRef] [PubMed] [Google Scholar]
- Marcel V, Ghayad SE, Belin S, et al. p53 acts as a safeguard of translational control by regulating fibrillarin and rRNA methylation in cancer. Cancer Cell 2013 ; 24 : 318–330. [CrossRef] [PubMed] [Google Scholar]
- Bywater MJ, Poortinga G, Sanij E, et al. Inhibition of RNA polymerase I as a therapeutic strategy to promote cancer-specific activation of p53. Cancer Cell 2012 ; 22 : 51–65. [CrossRef] [PubMed] [Google Scholar]
© 2014 médecine/sciences – Inserm
Liste des figures
 |
Figure 1. Régulation de l’initiation de la traduction. L’ARNm est représenté en noir sur lequel sont montrés les éléments cis-régulateurs : coiffe, structures secondaires (tige-boucle), IRES (internal ribosome entry sites), uORF (upstream open reading frame), séquences de reconnaissance par des miARN et des protéines. Les éléments trans-régulateurs sont schématisés en bleu : eIF (eukaryotic initiation factors), ITAF (IRES trans-acting factors), RNA-BP (RNA-binding proteins), miARN et PABP (polyA binding proteins). Les ribosomes sont présentés en gris avec les sous-unités 40S et 60S. |
| Dans le texte | |
 |
Figure 2. Altération qualitative des ribosomes : impact sur la tumorigenèse. L’inactivation de p53 (MTp53) conduit à une surexpression de la fibrillarine (FBL). La quantité anormale de FBL induit une production de ribosomes dont les patrons de méthylation sont altérés. Ces ribosomes modifiés participent à la diminution de la fidélité de traduction et à l’augmentation de la traduction d’ARNm contenant des IRES et codant pour des protéines favorisant la tumorigenèse. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




