| Issue |
Med Sci (Paris)
Volume 27, Number 5, Mai 2011
|
|
|---|---|---|
| Page(s) | 476 - 479 | |
| Section | Nouvelles | |
| DOI | https://doi.org/10.1051/medsci/2011275010 | |
| Published online | 25 May 2011 | |
Reprogrammation épigénétique
L’apport de l’inactivation du chromosome X
Epigenetic reprogramming
The importance of X inactivation
Medical Research Council (MRC), Centre Development in Stem Cell Biology, Institute for Stem Cell Research, School of Biological Sciences, University of Edinburgh, MRC EH9 3JQ, Royaume-Uni
Au cours de ces dernières années, de nombreuses études suscitées par la découverte déterminante de Shinya Yamanaka [1] ont révélé l’extraordinaire puissance d’un nombre limité de facteurs de transcription dans l’induction de la pluripotence : Oct4, Sox2, Klf4, c-Myc. En effet, si le développement embryonnaire peut être considéré comme une série d’étapes ordonnées au cours desquelles des cellules pluripotentes voient leur potentiel développemental réduit et restreint à un seul et unique type cellulaire (épidermique, musculaire, nerveux, hépatique, etc.), l’expression forcée de ces quatre facteurs de transcription dans des cellules somatiques (induced pluripotent stem cells, iPS) induit la reprogrammation de leur épigénome et rétablit tout le potentiel perdu au cours de la différenciation. Outre son intérêt pour induire la reprogrammation in vitro, cette nouvelle méthode d’analyse de l’épigénétique des mammifères permettra sans doute une meilleure compréhension des deux vagues de reprogrammation épigénétique qui caractérisent l’embryogenèse [2]. Ces deux stades cruciaux de l’embryogenèse consistent en : (1) la formation de la masse cellulaire interne (MCI) du blastocyste précoce, qui nécessite l’effacement des marques épigénétiques spécifiques des gamètes ; (2) la formation de la lignée germinale, qui requiert l’effacement des marques somatiques récemment établies pour que s’établisse l’épigénotype gamétique.
Activation des deux chromosomes X dans les cellules pluripotentes (ES et iPS)
Il va de soi que la reprogrammation doit mettre en jeu un grand nombre de mécanismes touchant tous les aspects de la vie de la cellule, du contrôle des flux d’énergie jusqu’à la restructuration du cytosquelette en passant par la mise en place du profil d’expression génique approprié. Afin de mieux appréhender ce processus, il est donc nécessaire d’analyser minutieusement les évènements moléculaires qui, sur les plans chromatinien et transcriptionnel, permettent la transition d’un profil épigénétique somatique à un profil épigénétique caractéristique de cellules pluripotentes. Cette tâche est titanesque si on veut l’appréhender dans sa totalité et nécessite des approches pluridisciplinaires à large spectre en génomique, biochimie, biologie moléculaire et cellulaire, bio-informatique, modélisation, etc. Une approche plus modeste, utilisant des systèmes judicieusement choisis, pourra donc être d’un intérêt considérable pour décrypter les principes et hypothèses de fonctionnement, généralisables ensuite à l’échelle du génome entier. L’inactivation du chromosome X (mécanisme de compensation de dose des gènes liés au chromosome X rencontré chez tous les mammifères [3]), et plus particulièrement le contrôle de ses régulateurs majeurs Xist et Tsix [4], constitue un modèle pertinent pour l’étude des mécanismes de reprogrammation de cellules somatiques murines.
L’acquisition de la pluripotence in vivo et in vitro est accompagnée systématiquement par la reprogrammation de l’inactivation du chromosome X de telle façon que non seulement l’X inactif (Xi) est réactivé, mais qu’en plus chacun des deux X acquiert la compétence requise pour pouvoir devenir à son tour l’X inactivé lors de la différenciation de ces cellules pluripotentes [5]. Chez l’embryon, ce processus a lieu dans la masse cellulaire interne (MCI) du blastocyste, où l’inactivation passe d’un mode soumis à l’empreinte parentale (inactivation exclusive de l’X paternel) à un mode aléatoire (n’importe lequel des deux X peut être inactivé au cours de la différenciation). Par conséquent, les cellules souches embryonnaires (ES), dérivées de la MCI, disposent de deux chromosomes X actifs, et il en va de même pour les cellules pluripotentes (iPS) issues du processus de reprogrammation in vitro. A contrario, l’inactivation aléatoire d’un des deux X est mise en place lors de la différenciation cellulaire.
Réprimer Xist pour réactiver le Xi lors de l’acquisition de la pluripotence
Présent sur le chromosome X, le gène Xist produit un long ARN non codant qui a les propriétés uniques de n’être exprimé qu’à partir du Xi et de former un domaine nucléaire dédié à la répression des gènes du Xi [6]. Dans les cellules pluripotentes, l’expression de Xist est réprimée, alors que leur différenciation induit l’expression monoallélique de Xist à partir d’un seul X aléatoirement choisi, qui devient alors le Xi (Figure 1). Différentes approches ont montré que, du moins dans les cellules ES, le promoteur de Xist est réfractaire au recrutement de la machinerie basale de transcription, et est enfoui dans une structure chromatinienne fermée. En revanche, dans les cellules différenciées où l’inactivation d’un des deux X a eu lieu, le promoteur de l’allèle de Xist présent sur le Xi recrute efficacement la machinerie de transcription et sa chromatine est enrichie en marques euchromatiques. Le promoteur de Xist situé sur le X actif se trouve quant à lui dans un état hétérochromatique, incapable de recruter la machinerie de transcription. Ces marques différentielles et leur acquisition au cours de la différenciation sont certainement des déterminants importants des mécanismes qui dictent quel chromosome X deviendra le Xi [4].
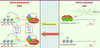 |
Figure 1 Couplage moléculaire entre la régulation de l’inactivation du chromosome X et la pluripotence. Dans les cellules pluripotentes, Nanog, Oct4 et Sox2 sont recrutés au niveau de l’intron 1 du gène Xist et maintiennent Xist dans un état silencieux. Rex1, Klf4 et c-Myc sont recrutés par DXPas34 pour activer la transcription de Tsix. Tsix induit quant à lui une configuration fermée sur le promoteur de Xist, en rendant tous les allèles présents épigénétiquement équivalents. Dans les cellules différenciées, une structure chromatinienne particulière différencie les allèles actifs et inactifs du gène Xist, qui est donc transcrit ou non, et Tsix n’est pas transcrit. |
Tsix est un deuxième gène non codant essentiel au bon déroulement de l’inactivation de l’X. Exprimé à partir du même locus que Xist, mais dans l’orientation inverse, Tsix est un des premiers gènes antisens naturels qui ont été caractérisés. Tsix est fortement exprimé dans les cellules ES alors qu’il s’éteint en cours de différenciation [4]. De façon frappante, sa transcription (ou l’ARN produit) est nécessaire à la mise en place de la structure chromatinienne du locus Xist dans les cellules ES. En particulier, l’invalidation génétique de Tsix provoque l’ouverture chromatinienne du promoteur de Xist. Ceci entraîne l’induction systématique de Xist lorsque la différenciation est déclenchée. Tsix agit donc, dans les cellules ES, comme un régulateur spécifique de l’état chromatinien de Xist [4].
Il est important de signaler que l’expression forcée de Xist dans des cellules ES induit l’inactivation [6]. Donc, en termes moléculaires, il est primordial de réprimer Xist pour réactiver le Xi lors de l’acquisition de la pluripotence. Précédemment, nous avons montré que les trois piliers de l’état pluripotent, Nanog, Oct4 et Sox2, couplent la réactivation du Xi à la pluripotence par la répression qu’ils exercent directement sur Xist [7]. Cependant, d’autres activités sont clairement nécessaires pour reprogrammer Xist au cours de l’acquisition de la pluripotence : étant donné l’héritabilité potentielle des marques chromatiniennes, la chromatine du locus Xist doit être « mise à zéro » pour éviter que les marques qui différenciaient l’allèle actif de l’allèle inactif ne perpétuent l’inactivation du Xi hérité. Cela revient à activer nécessairement Tsix.
Régulation de l’expression de Tsix : un rôle pour Klf4, c-Myc et Rex1
Après avoir montré que Nanog, Oct4 et Sox2 répriment directement Xist dans les cellules ES, il semblait logique d’étendre l’analyse des relations entre facteurs de pluripotence et gènes de contrôle de l’inactivation à l’étude de Klf4 et c-Myc. Dans cette nouvelle étude [8], l’emploi de l’immunoprécipitation de chromatine a été central et a révélé que Klf4 et c-Myc sont associés à la région 5’ de Tsix dans les cellules ES indifférenciées. De plus, le recrutement de ces deux facteurs a lieu à proximité de DXPas34, un minisatellite associé à Tsix dont l’activité enhancer avait été mise en évidence préalablement [9]. La délétion génétique de DXPas34 aboutit à la perte de fixation de Klf4 et c-Myc et s’accompagne d’un défaut majeur de l’activité de recrutement de l’ARN polymérase II par le promoteur de Tsix. Klf4 et c-Myc sembleraient donc être les facteurs-clés de l’activation spécifique de Tsix dans les cellules pluripotentes. Cependant, l’expression de Klf4 et c-Myc n’est pas exclusive aux cellules pluripotentes. En effet, l’analyse de cellules souches du trophectoderme, dans lesquelles l’expression de Tsix est fortement diminuée, a révélé la présence de Klf4 et c-Myc au niveau de la région 5’ de Tsix. Ceci indique que d’autres facteurs, strictement spécifiques aux cellules pluripotentes, doivent donc assurer les forts niveaux d’expression de Tsix dans les cellules pluripotentes.
Différentes études avaient montré que DXPas34 est constitué d’une répétition en tandem de sites de fixation pour Ctcf et Yy1, deux facteurs de transcription ubiquitaires [9]. Dans ce contexte, comme le facteur de pluripotence Rex1 est évolutivement lié à Yy1 et interagit avec l’ADN au niveau de séquence de même spécificité, il devenait un candidat de choix pour réguler Tsix via DXPas34. Notre découverte du recrutement de Rex1 par DXPas34 dans les cellules ES indifférenciées confirmait cette hypothèse [8]. En utilisant l’ARN interférence pour réduire son niveau d’expression, le mécanisme d’action de Rex1 a été décortiqué : Rex1 semble nécessaire à l’échappement de l’ARN polymérase II du promoteur pour transcrire activement Tsix. Rex1 ne régule donc pas l’initiation de la transcription de Tsix, mais son élongation, et confère à Tsix une activité maximale et optimale uniquement dans les cellules pluripotentes. En conclusion, DXPas34 apparaît comme un module de recrutement de Klf4, c-Myc et Rex1 pour d’une part recruter l’ARN polymérase II au promoteur de Tsix (activité qui est probablement dépendante de Klf4) et d’autre part faciliter l’élongation de la transcription via l’action de Rex1 (en synergie probable avec c-Myc, voir ci-dessous).
Rôles-clés de nouveaux facteurs dans la reprogrammation
Ces résultats [8] complètent donc notre travail précédent [7] et montrent que les deux régulateurs majeurs de l’inactivation de l’X, Xist et Tsix, sont directement contrôlés par les facteurs-clés de la pluripotence et de la reprogrammation (Figure 1). Outre leur intérêt pour connaître le domaine de l’inactivation de l’X [10], ces données placent le locus Xist/Tsix au premier plan scientifique en tant que paradigme de la reprogrammation épigénétique et conduisent à des hypothèses qui pourraient être extrapolées au phénomène global déclenché par l’expression forcée de Klf4, c-Myc, Oct4 et Sox2. Par exemple, bien qu’essentiels au démarrage du processus de reprogrammation, ces quatre facteurs ne sont clairement pas suffisants. La réexpression d’autres facteurs-clés associés au réseau de régulation génique spécifique des cellules pluripotentes est nécessaire pour que la transition soit complètement achevée. Ceci a été démontré pour Nanog [11], comme anticipé par l’analyse de la régulation de Xist [5], et pourrait se révéler vrai pour d’autres régulateurs tels que Rex1. En effet, l’analyse de la régulation de Tsix suggère que Rex1 pourrait jouer un rôle-clé en donnant une spécificité développementale à l’action de c-Myc, gène à expression ubiquitaire qui est nécessaire pour que l’élongation de la transcription s’effectue correctement [12]. Par ailleurs, comme l’illustre le rôle de Tsix sur la chromatine, des changements locaux et globaux de la chromatine doivent certainement se produire pendant la reprogrammation pour faciliter la sortie de l’état somatique et stabiliser l’état pluripotent. À cet égard, la découverte d’ARN non codants impliqués dans la reprogrammation épigénétique [13] pousse l’analogie un pas plus loin.
L’étude de l’inactivation du chromosome X a été pionnière pour comprendre de nombreux aspects de la régulation du génome, avec la mise en évidence des ARN non codants, de la transcription antisens et de l’organisation nucléaire. Aujourd’hui, il semble clair que ce domaine de recherche aura une place prépondérante dans la compréhension de la reprogrammation épigénétique. ‡
Conflit d’intérêts
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
Références
- Takahashi K, Yamanaka S. Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors. cell 2006 ; 126 : 663-676. [Google Scholar]
- Surani MA, Hayashi K, Hajkova P. Genetic and epigenetic regulators of pluripotency. Cell 2007 ; 128 : 747-762. [Google Scholar]
- Chow J, Heard E. X inactivation and the complexities of silencing a sex chromosome. Curr Opin Cell Biol 2009 ; 21 : 359-366. [CrossRef] [PubMed] [Google Scholar]
- Navarro P, Avner P. An embryonic story: analysis of the gene regulative network controlling Xist expression in mouse embryonic stem cells. Bioessays 2010 ; 32 : 581-588. [CrossRef] [PubMed] [Google Scholar]
- Navarro P, Avner P. When X-inactivation meets pluripotency: an intimate rendez-vous. FEBS Lett 2009 ; 583 : 1721-1727. [CrossRef] [PubMed] [Google Scholar]
- Wutz A, Jaenisch R. A shift from reversible to irreversible X inactivation is triggered during ES cell differentiation. Mol Cell 2000 ; 5 : 695-705. [CrossRef] [PubMed] [Google Scholar]
- Navarro P, Chambers I, Karwacki-Neisius V, et al. Molecular coupling of Xist regulation and pluripotency. Science 2008 ; 321 : 1693-1695. [CrossRef] [PubMed] [Google Scholar]
- Navarro P, Oldfield A, Legoupi J, et al. Molecular coupling of Tsix regulation and pluripotency. Nature 2010 ; 468 : 457-460. [CrossRef] [PubMed] [Google Scholar]
- Vigneau S, Clerc P. Sans Tsix, les mâles aussi inactivent leur chromosome X. Med Sci (Paris) 2006 ; 22 : 926-928. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Navarro P. Inactivation du chromosome X et pluripotence. Med Sci (Paris) 2009 ; 25 : 15-17. [CrossRef] [EDP Sciences] [PubMed] [Google Scholar]
- Silva J, Nichols J, Theunissen TW, et al. Nanog is the gateway to the pluripotent ground state. Cell 2009 ; 138 : 722-737. [CrossRef] [PubMed] [Google Scholar]
- Rahl PB, Lin CY, Seila AC, et al. c-Myc regulates transcriptional pause release. Cell 2010 ; 141 : 432-445. [CrossRef] [PubMed] [Google Scholar]
- Loewer S, Cabili MN, Guttman M, et al. Large intergenic non-coding RNA-RoR modulates reprogramming of human induced pluripotent stem cells. Nat Genet 2010 ; 42 : 1113-1117. [CrossRef] [PubMed] [Google Scholar]
© 2011 médecine/sciences – Inserm / SRMS
Liste des figures
 |
Figure 1 Couplage moléculaire entre la régulation de l’inactivation du chromosome X et la pluripotence. Dans les cellules pluripotentes, Nanog, Oct4 et Sox2 sont recrutés au niveau de l’intron 1 du gène Xist et maintiennent Xist dans un état silencieux. Rex1, Klf4 et c-Myc sont recrutés par DXPas34 pour activer la transcription de Tsix. Tsix induit quant à lui une configuration fermée sur le promoteur de Xist, en rendant tous les allèles présents épigénétiquement équivalents. Dans les cellules différenciées, une structure chromatinienne particulière différencie les allèles actifs et inactifs du gène Xist, qui est donc transcrit ou non, et Tsix n’est pas transcrit. |
| Dans le texte | |
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




