Figure 1.
Télécharger l'image originale
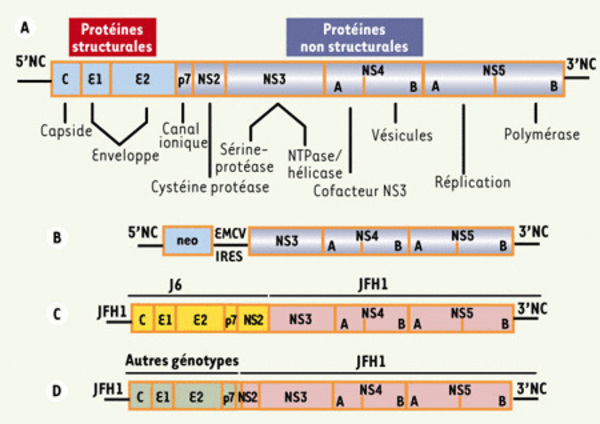
Organisation du génome du virus de l’hépatite C et structure des réplicons ou des génomes infectieux. A. Représentation schématique du génome viral. La fonction des protéines clivées est indiquée. C est la protéine de capside, E1 et E2 sont les glycoprotéines d’enveloppe, p7 forme un canal ionique impliqué dans la réplication du virus, NS2 est une cystéine protéase, NS3 forme un complexe stable avec NS4A créant une sérine protéase alors que son domaine carboxy-terminal contient une activité de phosphatase des nucléotides et une activité hélicase. NS4B peut induire la formation de vésicules intracellulaires servant de support à la réplication du virus. La phosphoprotéine NS5A est un co-facteur de réplication. NS5B est l’ARN polymérase dépendante de l’ARN. B. Structure des réplicons subgénomiques. La traduction du gène neo (conférant la résistance à G418) est sous la dépendance de l’IRES du HCV alors que les autres protéines sont sous la dépendance du site d’entrée dans le ribosome (internal ribosomal entry site, IRES) du virus de l’encéphalomyocardite (EMCV). C. Représentation schématique du virus chimère construit par Lindenbach et al. La région comprise entre le gène de capside et NS2 est dérivée de la souche J6 (génotype 2a), les autres régions du génome appartiennent à la souche JFH1 (génotype 2a). D. Représentation schématique des virus chimères construits par Pietschmann et al. Les fragments génomiques ont été fusionnés immédiatement après le premier domaine transmembranaire de NS2.
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




