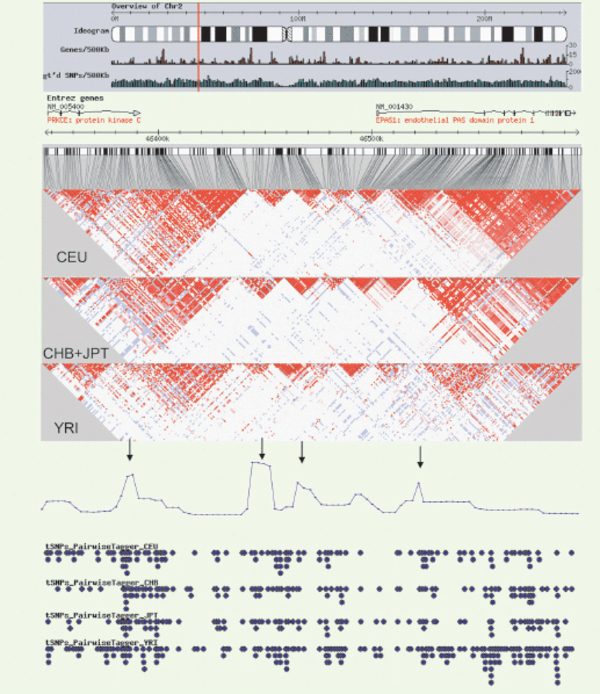
Figure 2.
Télécharger l'image originale
DL observé sur une région du chromosome 2 dans les populations HapMap. Les données du site HapMap (http://www.hapmap.org) ont été utilisées. Section 1 : Localisation des gènes et des SNP génotypés sur tout le chromosome 2. Section 2 : Localisation des gènes et des SNP génotypés entre les positions 46346800 et 46596799 de l’assemblage NCBI 34 du chromosome 2. Section 3 : Détail du DL entre les SNP de la région pour les populations HapMap (les populations JPT et CHB ont été combinées). Le programme Haploview (http://www.broad.mit.edu/mpg/haploview/) avec la méthode des intervalles de confiance de Gabriel [10] a été utilisé. Différentes teintes de rouge : couleur en fonction du D’ (rouge foncé : D’=1) et aucune preuve de recombinaison (selon les intervalles de confiance). En blanc : D’ <1,0 et preuve de recombinaison. En bleu : D’ = 1,0, mais la présence de recombinaison est non-concluante. Les blocs d’haplotypes sont indiqués en noirs et contiennent au moins 95 % de paires de SNP sans preuve de recombinaison. Section 4 : Taux de recombinaison moyen pour les quatre populations mesuré avec la méthode de McVean [16]. Les points chauds de recombinaison sont indiqués avec une flèche. Section 5 : SNP marqueurs sélectionnés selon l’algorithme Tagger pairwise (http://www.broad.mit.edu/mpg/tagger/) pour les quatre populations.
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




