Figure 1.
Télécharger l'image originale
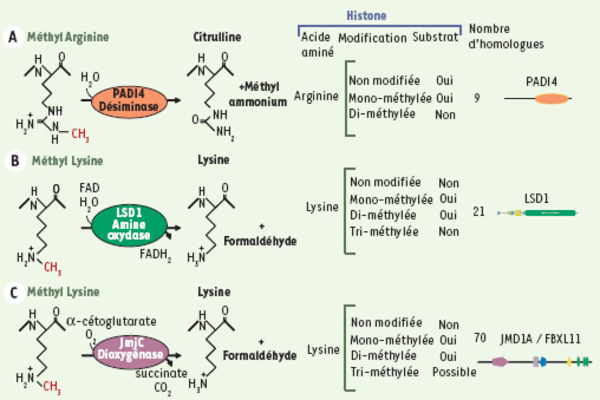
Mécanismes enzymatiques de la déméthylation et spécificité de substrat des histone déméthylases. A. La méthylation des arginines est supprimée par les arginine désiminases (PADI) pour engendrer une citrulline et du méthylammonium. Les enzymes PADI sont peu spécifiques et sont actives sur différentes arginines monométhylées des histones. Elles peuvent même convertir des arginines non-méthylées en citrullines. B. Deux mécanismes rendent compte de la déméthylation des méthyl-lysines. Les amine oxydases (de type LSD1) régénèrent une lysine et du formaldéhyde. La formation d’une imine intermédiaire par transfert de deux atomes d’hydrogène sur le cofacteur FAD nécessite un azote protoné, ce qui limite la réaction aux lysines mono- et diméthylées. C. Les di-oxygénases à domaine JmjC engendrent également une lysine et du formaldéhyde mais peuvent théoriquement déméthyler les tri-méthyl-lysines. À la différence des histone déméthylases spécifiques des arginines, les histones déméthylases spécifiques des lysines ont une très grande spécificité de substrat : LSD1 pour les lysines 4 ou 9 de l’histone H3, selon le cofacteur associé, et FBLX11 pour la lysine 36 de l’histone H3. Le nombre de protéines humaines qui possèdent des domaines désiminases, amine oxidases ou JmjC similaires à ceux de PADI4, LSD1 ou FBXL11 est indiqué (http://www.sanger.ac.uk/Software/Pfam/). Seule la déméthylation des monométhyl-lysine est montrée (adapté de [14]).
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




