| Numéro |
Med Sci (Paris)
Volume 40, Numéro 6-7, Juin-Juillet 2024
Nos jeunes pousses ont du talent !
|
|
|---|---|---|
| Page(s) | 584 - 586 | |
| Section | Partenariat médecine/sciences - Écoles doctorales - Masters | |
| DOI | https://doi.org/10.1051/medsci/2024077 | |
| Publié en ligne | 8 juillet 2024 | |
Enquête exclusive sur le psoriasis
Les kératinocytes et les fibroblastes passent aux aveux grâce à la transcriptomique
Exclusive investigation on psoriasis: Keratinocytes and fibroblasts confess through transcriptomics
1
Master 2 Immunologie Intégrative et Systémique, Parcours Immunologie, Master Biologie Moléculaire et cellulaire, Sorbonne Université, Paris, France
2
Master 2 Immunologie Translationnelle et Biothérapies, Parcours Immunologie, Master Biologie Moléculaire et cellulaire, Sorbonne Université, Paris, France
3
Inserm UMRS1138, Sorbonne Université, Paris, France
a imranebenmoussa2@gmail.com
b bienfait.abasi12@gmail.com
c Fatimzehra.afrhkhan@gmail.com
d mountadir.ines@gmail.com
e claire.deligne@sorbonne-universite.fr
Le psoriasis est une maladie cutanée qui est une fenêtre sur la complexité de la réponse immunitaire. Affectant environ 2 % de la population française, cette maladie inflammatoire chronique à l’étiologie mal comprise se manifeste par des lésions cutanées et une inflammation persistante impliquant de nombreux acteurs de l’immunité innée et acquise. Ce contexte pro-inflammatoire est à l’origine de la polarisation de lymphocytes T (LT) en lymphocytes T helper 1 (Th1), Th17 et Th22, au centre de la réponse immunitaire, et conduit, entre autres, à l’hyperprolifération des kératinocytes, une des caractéristiques majeures du psoriasis. Néanmoins, les mécanismes sous-jacents associés à cette maladie et l’intrication des acteurs cellulaires et moléculaires locaux demeurent encore mal connus. L’étude que nous présentons [1] apporte une nouvelle perspective sur les acteurs cellulaires du psoriasis grâce aux techniques de séquençage de l’ARN sur cellule unique (single cell RNA sequencing, scRNAseq) et de séquençage spatial. Cela leur a permis de réaliser une étude non biaisée et exhaustive afin de réviser notre compréhension de la physiopathologie du psoriasis et de révéler, en particulier, le rôle crucial et jusqu’alors inconnu des fibroblastes dans l’orientation et l’amplification de la prolifération des kératinocytes et le renforcement des réponses inflammatoires et immunitaires liées au psoriasis.
Décryptage des acteurs clés du psoriasis par scRNAseq
Par rapport au bulk RNA sequencing, qui renseigne sur l’expression des gènes d’une population cellulaire de façon indiscriminée [2], le scRNAseq permet d’analyser l’expression des gènes de cellules individuelles. Dans l’étude à laquelle nous nous sommes intéressés, les auteurs ont commencé par caractériser les composants cellulaires impliqués dans le psoriasis par scRNAseq à partir de biopsies de peaux lésionnelles, de peau en périphérie de la lésion, ou de peau saine (Figure 1). Après avoir traité les échantillons, une technique nommée UMAP (uniform manifold approximation and projection for dimension reduction) a été utilisée afin de visualiser les différentes sous-populations cellulaires présentes dans chaque échantillon. Cette méthode a permis aux auteurs d’identifier, de quantifier et de caractériser les types cellulaires présents, dont les kératinocytes, qui présentaient un profil transcriptomique unique dans un contexte psoriasique – ce qui était connu –, mais également des fibroblastes [3]. Les auteurs ont mis en évidence par clustering, un processus visant à regrouper des données fondées sur leurs similarités ou leurs caractéristiques communes, des sous-populations de fibroblastes transcriptionnellement distinctes dans la peau psoriasique. Parmi ceux exprimant le gène SFRP2 (secreted frizzled-related proteins 2) [4], certains présentaient des signatures pro-inflammatoires, caractérisées par l’expression de marqueurs inflammatoires, comme l’IFN-γ (interféron gamma), le TNF-α (tumor necrosis factor alpha) et l’IL-17A (interleukine 17A), et de protéases, comme la cathepsine S, connue pour son rôle dans l’activation de l’IL-36G [5], une cytokine pro-inflammatoire synthétisée par les kératinocytes. L’IL-36G activée contribuerait ainsi à l’amplification de la sécrétion de l’IL-17A et du TNF-α (Figure 2), et les fibroblastes contribueraient activement à la pathogenèse du psoriasis en association avec les kératinocytes. Les cellules myéloïdes ainsi qu’une sous population de LT CD8 sécréteurs d’IL-17A (Tc17) ont également été mis en évidence comme étant des acteurs sous-jacents à la pathogenèse du psoriasis.
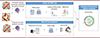 |
Figure 1. Détail des méthodes transcriptomiques utilisées dans l’étude. Ce schéma illustre les principales étapes techniques réalisées par les auteurs. Des biopsies cutanées de tissus psoriasiques, en périphérie de la lésion ou sains ont été collectées. Les tissus ont été analysés en scRNAseq grâce à la technologie 10X afin d’analyser l’expression différentielle des gènes cellule par cellule. En parallèle, la transcriptomique spatiale a permis d’identifier les transcrits localement dans le tissu. Enfin, l’analyse intégrée des ligands-récepteurs a offert une idée de l’interactome de ces populations localement (figure créée avec BioRender). |
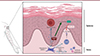 |
Figure 2. Représentation des interactions cellulaires et moléculaires dans le psoriasis. Les différentes interactions entre cellules, ainsi que les cytokines sécrétées dans la peau affectée par le psoriasis sont illustrées. L’étude met en évidence le rôle des fibroblastes SFRP2+ dans la sécrétion de chimiokines dans l’attraction de cellules myéloïdes de Tc17 mais aussi de kératinocytes. Elle révèle aussi la sécrétion de la cathepsine S par les fibroblastes SFRP2+ qui clive et active l’IL-36G sécrétée par les kératinocytes. L’IL-36G activée amplifie la sécrétion d’IL-17A et de TNF-α. Les Tc17, attirés par le CXCL12 sécrété par les fibroblastes SFRP2+, produisent les cytokines IL-17A et TNF-α (figure créée avec BioRender). |
Localisation tissulaire et interactions des cellules par séquençage spatial
Afin de comprendre le rôle des interactions cellulaires locales dans le psoriasis, le séquençage spatial est une technique particulièrement intéressante et indispensable. Cette technique vise en effet à quantifier le nombre de transcrits à des emplacements spatiaux distincts dans un tissu donné, en hybridant des sondes oligonucléotidiques associées aux coordonnées spatiales du tissu, ce qui permet de capturer et de séquencer les molécules d’ARN à partir de tissus congelés [6]. Les signatures associées aux sous-populations identifiées grâce au scRNAseq ont servi à annoter les résultats issus du séquençage spatial, permettant de cartographier l’emplacement des populations cellulaires. Comme attendu, les kératinocytes étaient répartis dans l’épiderme. Les cellules myéloïdes, les LT et les cellules endothéliales se trouvaient dans le derme superficiel, et les fibroblastes étaient localisés dans le derme.
Des changements de composition et de communication cellulaires ont cependant été observés entre les peaux saines et psoriasiques, notamment une modification des interactions ligands-récepteurs entre les kératinocytes, les fibroblastes, les LT et les cellules myéloïdes. Ces interactions ont été identifiées grâce à un algorithme, permettant l’interprétation des données du scRNAseq en détectant des paires de récepteur-ligand exprimées par les différentes populations cellulaires, ce qui indique les interactions cellulaires potentielles. Ce travail met en lumière un réseau inflammatoire complexe complétant les principales voies connues dans le psoriasis, en intégrant les sous-populations avec leur localisation tissulaire et leurs interactions. Cela a, par exemple, permis d’identifier les fibroblastes SFRP2+ comme aussi cruciaux que les kératinocytes dans le recrutement des cellules immunitaires via de nombreuses cytokines et chimiokines inflammatoires, notamment CCL13 (C-C motif chemokine ligand 13), CCL19, et CXCL12 (C-X-C motif chemokine ligand 12), dont les récepteurs sont exprimés par les cellules myéloïdes et les Tc17 présents dans le tissu. Les fibroblastes orienteraient ainsi la réponse immunitaire inflammatoire localement en modifiant les cellules environnantes, en recrutant activement les cellules immunitaires, et en interagissant étroitement avec les kératinocytes.
Conclusion et Perspectives
Plusieurs études ont déjà démontré le rôle de certaines populations cellulaires dans le psoriasis, comme les macrophages, les kératinocytes ou encore les LT, de manière indépendante les unes des autres [7]. Cependant, l’absence de techniques intégratives permettant d’englober l’identification et l’hétérogénéité intrinsèque de ces populations cellulaires, des molécules qu’elles sécrètent, mais surtout de leurs interactions, était une limite.
Cette étude dépasse la simple utilisation du scRNAseq ou du séquençage spatial. C’est en effet leur analyse combinée, avec celle de l’interaction ligand-récepteur, qui a permis de donner une vue complète des acteurs cellulaires et moléculaires locaux dans le psoriasis. Elle a également permis d’identifier comment des sous-populations de fibroblastes ont un impact local sur les cellules immunitaires et dialoguent avec les kératinocytes dans le tissu. Cependant, ces approches présentent des limites, comme la faible résolution des données du séquençage spatial, limitant l’exploration de voies de signalisation faiblement modulées, et le biais de capture des cellules à analyser aboutissant ici à l’absence de détection des neutrophiles, dont la présence a pourtant déjà été démontrée [8].
Les techniques classiques en immunologie, comme la cytométrie en flux, reposent sur le choix des marqueurs et des populations étudiées par l’investigateur, biaisant de facto les découvertes, contrairement à la transcriptomique. Dans le psoriasis, d’autres études ont utilisé le scRNAseq pour décrypter les altérations cellulaires et métaboliques dépendantes du stade de la maladie [9] et des populations de LT CD8 [10]. Ces approches combinées ont par ailleurs été appliquées dans d’autres maladies, notamment pour révéler la diversité et la localisation cellulaire dans certains cancers. Par exemple, une étude menée par Liu et al. en 2023 [11], a montré grâce à cette approche combinée que des cellules cancéreuses du sein disséminées présentaient une activation de voies métaboliques et qu’elles interagissaient avec d’autres cellules du système immunitaire [11]. Ces résultats soulignent le potentiel d’une approche intégrée et non biaisée pour caractériser des maladies dont les mécanismes et les acteurs cellulaires et moléculaires ne sont pas complètement connus. Les découvertes de nouveaux biomarqueurs dans cette étude ouvrent la voie à de nouvelles perspectives diagnostiques et potentiellement thérapeutiques dans le psoriasis.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- Ma F, Plazyo O, Billi AC, et al. Single cell and spatial sequencing define processes by which keratinocytes and fibroblasts amplify inflammatory responses in psoriasis. Nat Commun 2023; 14 : 3455. [CrossRef] [PubMed] [Google Scholar]
- Kim YS, Choi J, Lee SH. Single-cell and spatial sequencing application in pathology. J Pathol Transl Med 2023; 57 : 43–51. [CrossRef] [PubMed] [Google Scholar]
- Wei K, Nguyen HN, Brenner MB. Fibroblast pathology in inflammatory diseases. J Clin Invest 2021; 131 : e149538. [CrossRef] [PubMed] [Google Scholar]
- Tabib T, Morse C, Wang T, et al. SFRP2/DPP4 and FMO1/LSP1 Define Major Fibroblast Populations in Human Skin. J Invest Dermatol 2018 ; 138 : 802–810. [CrossRef] [PubMed] [Google Scholar]
- Ainscough JS, Macleod T, McGonagle D, et al. Cathepsin S is the major activator of the psoriasis-associated proinflammatory cytokine IL-36γ. Proc Natl Acad Sci U S A 2017 ; 114 : E2748–E2757. [CrossRef] [PubMed] [Google Scholar]
- Williams CG, Lee HJ, Asatsuma T, et al. An introduction to spatial transcriptomics for biomedical research. Genome Med 2022; 14 : 68. [CrossRef] [PubMed] [Google Scholar]
- Elder JT, Bruce AT, Gudjonsson JE, et al. Molecular dissection of psoriasis: integrating genetics and biology. J Invest Dermatol 2010 ; 130 : 1213–1226. [CrossRef] [PubMed] [Google Scholar]
- Mellor LF, Gago-Lopez N, Bakiri L, et al. Keratinocyte-derived S100A9 modulates neutrophil infiltration and affects psoriasis-like skin and joint disease. Ann Rheum Dis 2022; 81 : 1400–8. [CrossRef] [PubMed] [Google Scholar]
- Castillo RL, Sidhu I, Dolgalev I, et al. Spatial transcriptomics stratifies psoriatic disease severity by emergent cellular ecosystems. Sci Immunol 2023; 8 : eabq7991. [CrossRef] [PubMed] [Google Scholar]
- Liu J, Chang HW, Huang ZM, et al. Single-cell RNA sequencing of psoriatic skin identifies pathogenic Tc17 cell subsets and reveals distinctions between CD8+ T cells in autoimmunity and cancer. J Allergy Clin Immunol 2021; 147 : 2370–80. [CrossRef] [PubMed] [Google Scholar]
- Liu YM, Ge JY, Chen YF, et al. Combined Single-Cell and Spatial Transcriptomics Reveal the Metabolic Evolvement of Breast Cancer during Early Dissemination. Adv Sci (Weinh) 2023; 10 : e2205395. [CrossRef] [PubMed] [Google Scholar]
© 2024 médecine/sciences – Inserm
Liste des figures
 |
Figure 1. Détail des méthodes transcriptomiques utilisées dans l’étude. Ce schéma illustre les principales étapes techniques réalisées par les auteurs. Des biopsies cutanées de tissus psoriasiques, en périphérie de la lésion ou sains ont été collectées. Les tissus ont été analysés en scRNAseq grâce à la technologie 10X afin d’analyser l’expression différentielle des gènes cellule par cellule. En parallèle, la transcriptomique spatiale a permis d’identifier les transcrits localement dans le tissu. Enfin, l’analyse intégrée des ligands-récepteurs a offert une idée de l’interactome de ces populations localement (figure créée avec BioRender). |
| Dans le texte | |
 |
Figure 2. Représentation des interactions cellulaires et moléculaires dans le psoriasis. Les différentes interactions entre cellules, ainsi que les cytokines sécrétées dans la peau affectée par le psoriasis sont illustrées. L’étude met en évidence le rôle des fibroblastes SFRP2+ dans la sécrétion de chimiokines dans l’attraction de cellules myéloïdes de Tc17 mais aussi de kératinocytes. Elle révèle aussi la sécrétion de la cathepsine S par les fibroblastes SFRP2+ qui clive et active l’IL-36G sécrétée par les kératinocytes. L’IL-36G activée amplifie la sécrétion d’IL-17A et de TNF-α. Les Tc17, attirés par le CXCL12 sécrété par les fibroblastes SFRP2+, produisent les cytokines IL-17A et TNF-α (figure créée avec BioRender). |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




