Figure 1.
Télécharger l'image originale
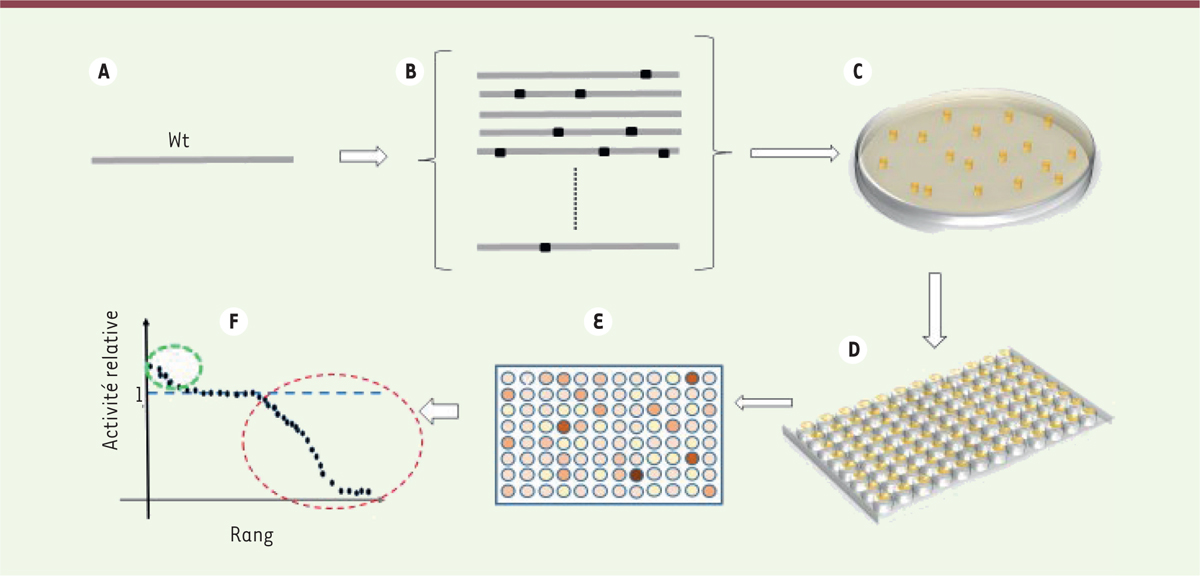
Les méthodes développées par Frances Arnold permettent de reconstituer en laboratoire les processus d’évolution et de les appliquer à l’amélioration des propriétés d’enzymes. Le schéma général est de partir d’une protéine naturelle que l’on souhaite faire évoluer. Le gène « sauvage » (A) de cette protéine va être aléatoirement muté, par exemple par PCR (polymerase chain reaction) mutagène (B), cloné dans un vecteur d’expression, puis intégré dans des microorganismes. La population résultante (C) comportera un grand nombre de variants de la protéine d’origine. Un sous-ensemble de ces variants pourra être échantillonné dans des cultures individuelles (D) puis l’activité de l’enzyme produit par chacun de ces clones sera évaluée, par exemple par mesure de la variation d’absorbance ou de fluorescence (E) produite par l’activité de l’enzyme. Si l’on classe les variants selon leur performance relative (l’enzyme de départ a une activité relative de 1), on observe typiquement qu’une fraction importante des variants (entourés en rouge) comporte des mutations dont l’effet est défavorable mais qu’également une fraction, souvent plus faible (entourés en vert), comporte des mutations qui améliorent l’activité mesurée (F). Si ces améliorations sont faibles, elles peuvent être cumulées entre elles, si bien qu’après plusieurs cycles d’évolution, les améliorations obtenues peuvent être importantes.
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




