Figure 1
Télécharger l'image originale
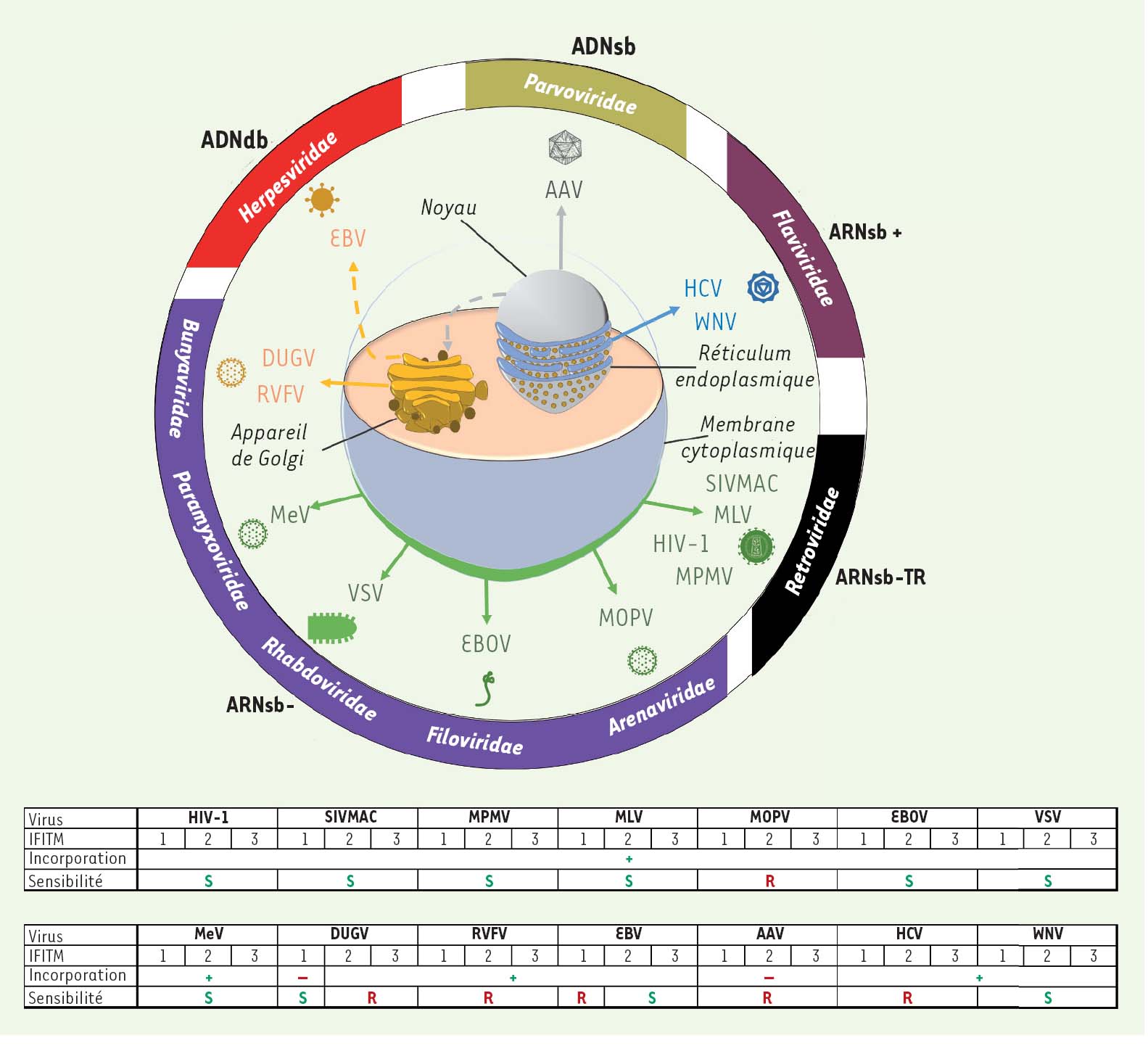
Mode d’assemblage des virions et influence des protéines IFITM sur le pouvoir infectieux des particules virales néoformées. Les différents virus sont représentés avec leur caractéristique génomique et leur groupe d’appartenance. ARNsb - : génome à ARN simple brin de polarité néga t i v e; ARNsb-TR : génome à ARN simple brin et nécessitant une transcriptase inverse; ARNsb + : génome à ARN simple brin de polarité positive; ADNsb : génome à ADN simple brin; ADNdb : génome à ADN double brin. Les virus étudiés sont indiqués. VIH-1 : virus de l’immunodéficience humaine de type 1; SIVMAC : virus de l’immunodéficience simienne du macaque; MPMV : virus simien Mason-Pfizer; MLV : virus de la leucémie murine; MOPV : virus Mopeia; EBOV : virus Ebola; VSV : virus de la stomatite vésiculaire; MeV : virus de la rougeole; DUGV : virus Dugbe; RVFV : virus de la fièvre de la vallée du Rift; EBV : virus Epstein-Barr; AAV : virus adéno-associé; HCV : virus de l’hépatite C; WNV : virus West Nile. Les tableaux récapitulent le rôle des IFITM pour chaque virus, à savoir leur incorporation dans les particules virales (indiquée par + ou -); La résistance ou la sensibilité des virus aux IFITM sont notées respectivement par R et S (adapté de [10]).
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




