Figure 1.
Télécharger l'image originale
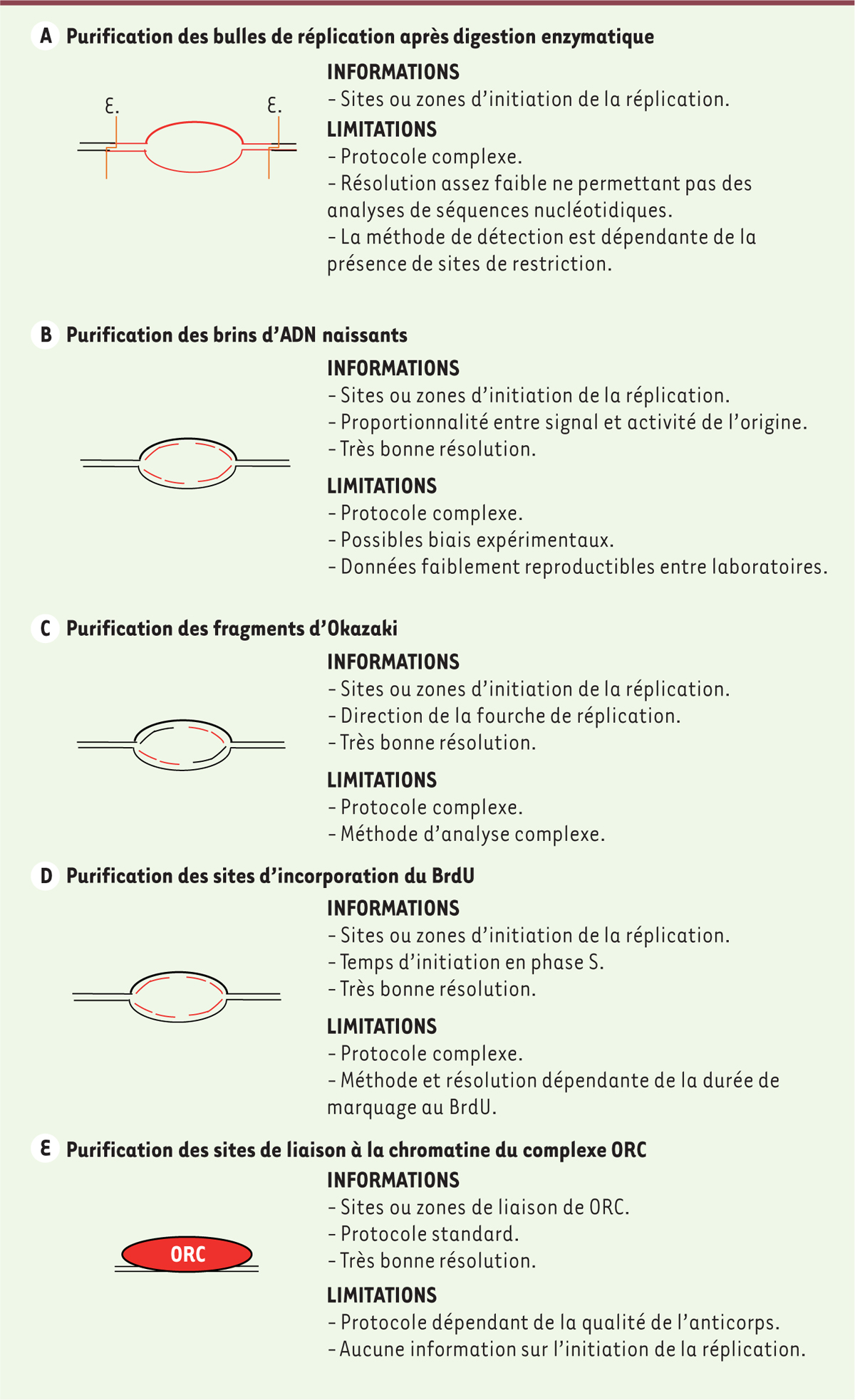
Description des méthodologies mises au point pour l’identification des origines de réplication à l’échelle du génome humain entier. A. Purification des bulles de réplication (d’après [7]). Après digestion enzymatique avec l’enzyme EcoRI (une enzyme de restriction qui coupe les sites G^AATTC, notée E.) de l’ADN génomique, les fragments d’ADN sont déposés sur une colonne d’agarose. Du fait de leur nature circulaire, les bulles de réplication sont retenues sur la colonne et éluées plus tardivement que le reste des brins d’ADN. Les ADN circulaires sont ensuite préparés et analysés par séquençage haut débit. B. Purification des brins d’ADN naissants (d’après [4, 5, 8]). Après purification, l’ADN génomique est dénaturé et analysé sur une colonne de fractionnement par la taille. Les fractions correspondant aux tailles 1 à 1,5 kilobases, et contenant les brins naissants, sont purifiées. Les échantillons sont alors traités à la lambda-endonucléase pour sélectionner les brins naissants et éliminer les fragments d’ADN (provenant de cassures lors de la préparation des échantillons). Ils sont ensuite préparés et analysés par séquençage haut-débit. C. Purification des fragments d’Okazaki (d’après [9]). Après un marquage à la 5-éthynyl-2’-désoxyuridine (EdU), un analogue de la thymidine, l’ADN est isolé des cellules puis dénaturé. Après passage sur une colonne de fractionnement, les fragments d’Okazaki sont purifiés grâce à l’EdU, l’ARN hydrolysé à la soude, et les ADN simples brins obtenus sont analysés par séquençage haut-débit. D. Purification des sites d’incorporation de la bromodésoxyuridine (BrdU)/Edu (d’après [13]). Après un marquage à la BrdU/EdU, l’ADN est isolé des cellules puis dénaturé. Les fragments d’ADN contenant l’EdU (ou le BrdU) sont purifiés et analysés par séquençage haut-débit. E. Purification des sites de liaison du complexe ORC (origin recognition complex) (d’après [6, 10]). Après fixation au formaldéhyde et capture des complexes ADN/protéine, la chromatine est extraite des cellules et fractionnée par sonication. Les brins de chromatine liés par ORC sont ensuite purifiés sur une colonne de billes d’anticorps dirigés contre la protéine ORC2. Après purification, les ADN sont analysés par séquençage haut-débit.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




