Figure 1.
Télécharger l'image originale
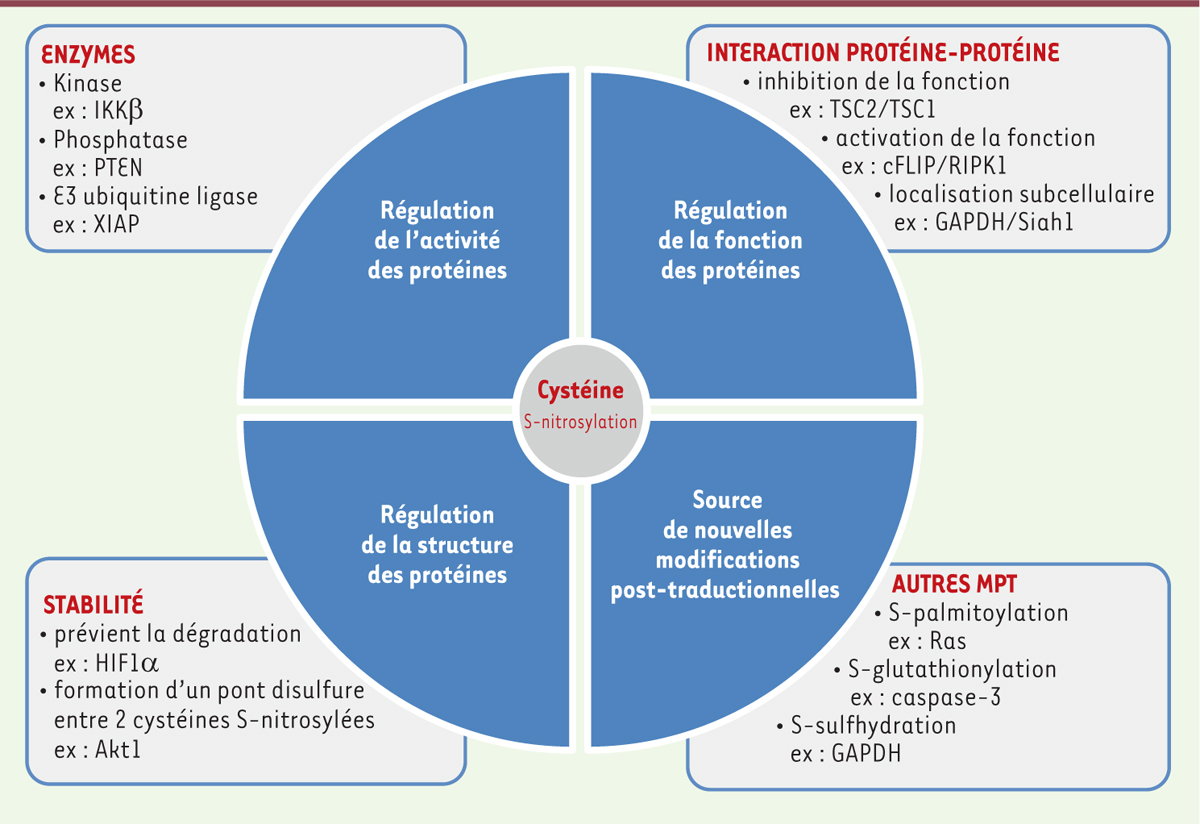
La cystéine au cœur de la S-nitrosylation. S-nitrosylation : régulation de l’activité des protéines. De nombreuses enzymes impliquées dans la MPT (modification post-traductionelle) des protéines possèdent un résidu cystéine dans leur site catalytique. La réactivité du monoxyde d’azote (NO) avec un tel résidu abroge la plupart du temps leur activité. Les enzymes de types kinases (par exemple IKKβ [inhibitor of NF-kB kinase beta]), phosphatases (par exemple PTEN [phosphatase and tensin homolog]), E3 ubiquitine ligases (par exemple XIAP [X-linked inhibitor of apoptosis]) sont souvent la cible du NO au sein de différentes voies de signalisation contrôlant la survie ou la mort cellulaire. De ce fait, la S-nitrosylation d’une cystéine spécifique peut influencer la survenue d’un grand nombre de MPT et par conséquent altérer plusieurs voies de signalisation cellulaire aussi bien positivement que négativement. S-nitrosylation : régulation de la structure et stabilité des protéines. Les groupements thiols formant ensemble des ponts disulfures ne réagissent pas avec le NO et par conséquent ne constituent pas des sites de S-nitrosylation. Néanmoins deux sites S-nitrosylés peuvent a posteriori réagir entre eux pour former un pont disulfure (par exemple Akt1 [RAC-alpha serine/threonine-protein kinase]) contribuant ainsi à la stabilité de la structure ternaire et quaternaire de la protéine. En revanche, la S-nitrosylation peut perturber la stabilité d’une métalloprotéine qui met en jeu des résidus cystéines ayant des propriétés de liaison avec les métaux. La S-nitrosylation du facteur HIF1α (hypoxia inducible factor 1 alpha) entraîne sa stabilisation. S-nitrosylation : régulation de la fonction et localisation des protéines. La S-nitrosylation peut moduler le rôle d’une protéine lorsque les résidus cystéine ne sont impliqués ni dans le domaine catalytique d’une enzyme ni dans sa stabilité. La S-nitrosylation peut influencer la capacité d’une protéine à interagir avec un partenaire déterminé et contrecarrer sa fonction. Dans le cas de TSC (tuberous sclerosis complex)2, sa S-nitrosylation prévient son interaction avec son partenaire protéique TSC1 et ainsi sa fonction. Au contraire, la S-nitrosylation de cFLIP (cellular FLICE [FADD-like IL-1β-converting enzyme]-inhibitory protein) induit son interaction avec RIPK1 (receptor-interacting protein kinase 1) pour promouvoir sa capacité à induire la voie classique NF-kB (nuclear factor-kappa B) et/ou sa localisation subcellulaire en affectant par exemple les processus de transcription (la protéine GAPDH [glyceraldehyde-3-phosphate dehydrogenase] sous sa forme S-nitrosylée assure son interaction avec la protéine Siah1 [siah E3 ubiquitin protein ligase 1] qui induit sa translocation dans le noyau), de traduction, de dégradation des protéines ou encore de modifications post-traductionnelles. S-nitrosylation : sources de nouvelles MPT. Les propriétés du groupement thiol d’une cystéine en font un site privilégié pour de nombreuses autres MPT telles que la S-palmitoylation (par exemple Ras), la S-glutathionylation (par exemple caspase-3) ou encore la S-sulfhydration (par exemple GAPDH) [49]. Il est important de noter que la S-nitrosylation d’un site peut à la fois prévenir la formation d’une autre MPT ou au contraire servir d’intermédiaire pour la formation d’une nouvelle MPT oxidative selon l’environnement rédox dans lequel il se trouve.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




