Figure 2.
Télécharger l'image originale
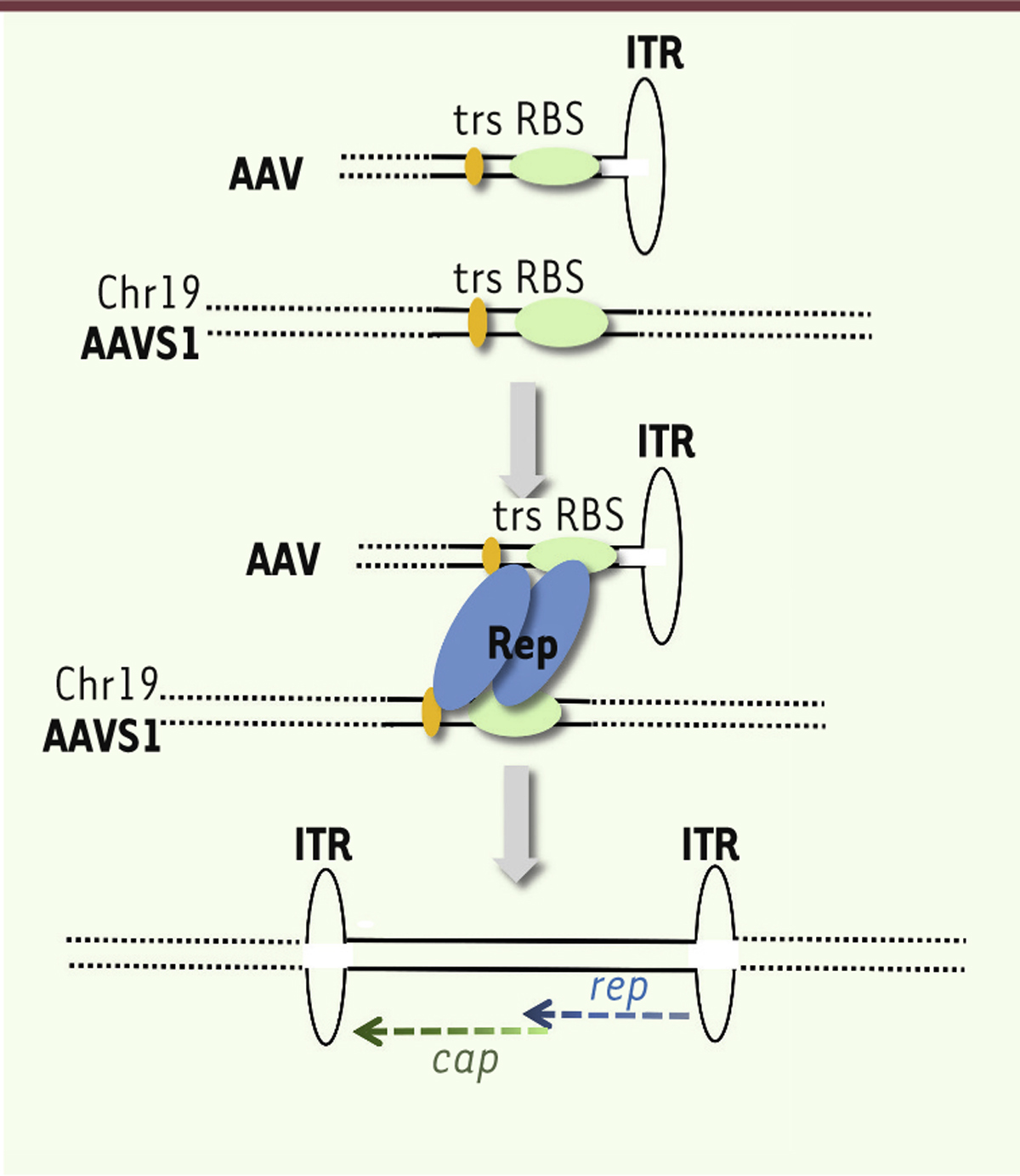
Modèle théorique d’intégration du génome de l’AAV sauvage dans le locus AAVS1. L’ITR (inverted terminal repeats) de l’AAV et le locus AAVS1 du génome cellulaire partagent un site de fixation pour les protéines Rep (ou RBS [Rep binding site]) et un site de clivage par ces mêmes protéines (ou trs, terminal resolution site). Selon le modèle d’intégration, les protéines virales Rep seraient capables de réaliser un pontage entre le génome viral et le génome cellulaire via la reconnaissance du site RBS. La fixation de ces protéines au RBS entraîne un clivage de l’ADN cellulaire et viral au niveau du site trs. Le génome viral s’intégrerait alors via un mécanisme encore mal élucidé impliquant plusieurs facteurs de réparation de l’ADN ainsi que des polymérases cellulaires. Des travaux récents ont montré que l’intégration du génome de l’AAV survenait majoritairement en orientation inverse. Il est important aussi de noter que l’intégration virale peut survenir dans une région chromosomique très large et de façon préférentielle en amont du site RBS. Il en résulte, dans la plupart des cas, une délétion partielle ou complète des ITR ainsi que des délétions et/ou des réarrangements des séquences cellulaires adjacentes. Des sites RBS consensus et des sites trs cryptiques ont été également retrouvés en grand nombre dans d’autres régions chromosomiques, ce qui explique la capacité du virus à cibler différents locus cellulaires (voir Figure 3).
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




