Figure 2.
Télécharger l'image originale
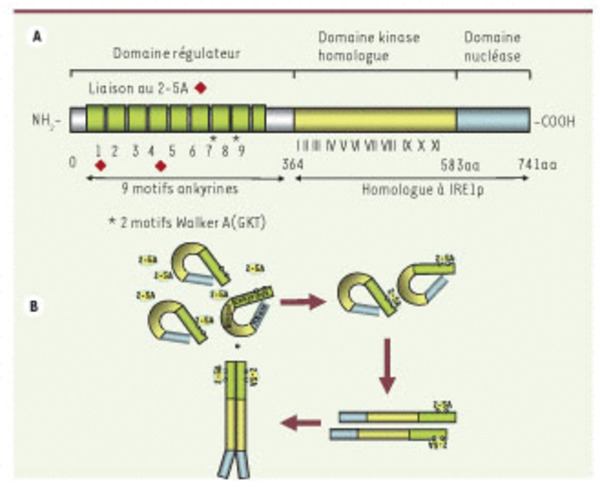
A. Structure de la RNase L. La RNase L est constituée de 3 domaines : un domaine amino-terminal régulateur qui fixe le 2-5A, un domaine central homologue à un domaine kinase et un domaine carboxy-terminal qui porte l’activité catalytique de cette enzyme. Le 2-5A interagit avec les domaines ankyrine R1-R4 (
 ) et les 2 motifs Walker A (fixation de l’ATP ou du GTP) sont situés entre les motifs ankyrines R7 et R8. La moitié carboxy-terminale de la RNase L est homologue à la nucléase IRE1p. B. Modèle d’activation de la RNase L. La fixation du 2-5A induit un changement de conformation de l’extrémité amino-terminale de la protéine, ce qui permet un démasquage du domaine nucléase carboxy-terminal. Ce changement de conformation permet son homodimérisation et/ou son interaction avec d’autres protéines et son activation.
) et les 2 motifs Walker A (fixation de l’ATP ou du GTP) sont situés entre les motifs ankyrines R7 et R8. La moitié carboxy-terminale de la RNase L est homologue à la nucléase IRE1p. B. Modèle d’activation de la RNase L. La fixation du 2-5A induit un changement de conformation de l’extrémité amino-terminale de la protéine, ce qui permet un démasquage du domaine nucléase carboxy-terminal. Ce changement de conformation permet son homodimérisation et/ou son interaction avec d’autres protéines et son activation.
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




