Figure 2.
Télécharger l'image originale
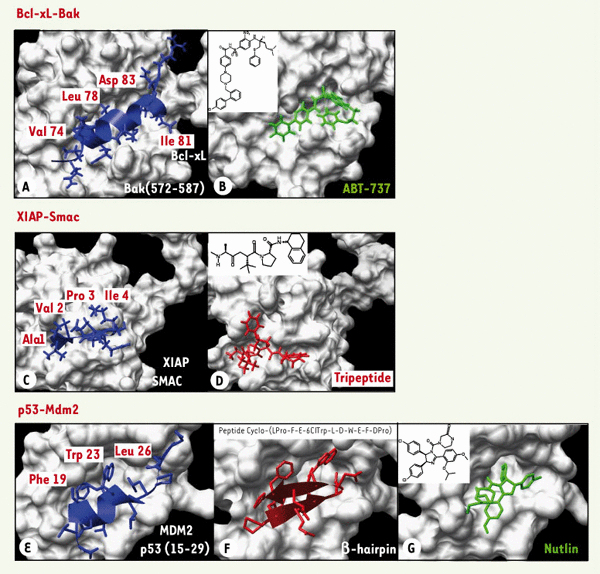
Cibler des interactions protéine-protéine : le mythe devient réalité. A, B. Interaction Bcl-xL-Bak. A. Quatre résidus de Bak représentent des hot spots pour son interaction avec Bcl-xL (pdb 1BXL, [29]). B. Une molécule chimique (ABT-737) inhibe l’interaction Bcl-xL-Bak (IC50 = 1 nM, pdb 1YSI, [31]) en mimant ces résidus critiques pour l’interaction. C, D. Interaction XIAP-Smac. C. La protéine Smac interagit avec XIAP via 4 résidus essentiels aminoterminaux, Ala1, Val2, Pro3, Ile4 (pdb 1G3F, [35]). D. Un tripeptide non naturel optimisé inhibe l’interaction avec une IC50 de 5 nM (pdb 1TFT, [39]). E, F, G. Interaction p53-Mdm2. E. La liaison du domaine de transactivation de p53 à la protéine Mdm2 (pdb 1YCR, [4]) fait intervenir principalement 3 résidus hot spots (Phe19, Trp23, Leu26). F. Un peptide mimétique contraint occupe la même poche de Mdm2 (pdb 2AXI, [40]) et inhibe l’interaction avec une IC50 de 140 nM. G. Des petites molécules chimiques, les nutlins, miment les hot spots de p53 et inhibent l’interaction avec une IC50 de 100 nM (pdb 1RVI [8]). Les images graphiques ont été produites grâce au logiciel UCSF Chimera package [41].
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




