| Numéro |
Med Sci (Paris)
Volume 23, Mars 2007
Protéomique clinique en oncologie
|
|
|---|---|---|
| Page(s) | 13 - 18 | |
| Section | M/S revues | |
| DOI | https://doi.org/10.1051/medsci/2007231s13 | |
| Publié en ligne | 15 mars 2007 | |
Les enjeux du préanalytique dans l’analyse du transcriptome et du proteome sanguins
Application à la recherche clinique: l’expérience du GOELAMS
Stakes of pre-analytical parameters in blood transcriptomic and proteomic analysis. Application to clinical research: the GOELAMS trial
1
Département HITC, UPRES EA3889, CHU Pontchaillou, 35033 Rennes, France
2
Inserm U318, CHU Michallon, 38043 Grenoble, France
3
Institut de Recherche en Biothérapie, CHU Saint Eloi, 34295 Montpellier, France
4
Laboratoire d’Immunologie, CHU de Nancy, 54000 Nancy, France
*
celine.pangault@univ-rennes1.fr
De nombreux paramètres liés au patient et aux conditions de prélèvement d’un échantillon biologique peuvent influer sur les propriétés de cet échantillon. Ces paramètres préanalytiques sont particulièrement importants à maîtriser dans le contexte d’études cliniques multicentriques. Nous rapportons ici les résultats préliminaires d’une étude conduite par le groupe coopératif GOELAMS (Groupe Ouest-Est des Leucémies aiguës et Autres Maladies du Sang) afin d’identifier les paramètres préanalytiques susceptibles d’affecter une étude protéomique sur sérum dans le contexte d’études cliniques concernant les lymphomes B non hodgkiniens.
Abstract
Numerous parameters related to the patients and the conditions of the sample collection can affect the properties of a biological sample. The pre-analytical parameters are especially important to deal with in the setting of multicenter clinical trials. Here, we report the preliminary results of a trial conducted by the cooperative group GOELAMS (Groupe Ouest-Est des Leucémies aiguës et Autres Maladies du Sang) to define the preanalytical parameters that affect the proteomic analysis of serum samples in patients with B-cell non Hodgkin lymphoma included in clinical trials.
© 2007 médecine/sciences - Inserm / SRMS
Cette dernière décennie a été marquée par d’importantes avancées en biologie soutenues par des progrès technologiques tournés vers les nanosciences. Ces derniers ne doivent pas faire oublier l’importance des étapes préalables à l’analyse qui permettent de garantir robustesse et fiabilité des résultats obtenus. En recherche clinique, nombre de paramètres liés au patient (âge, sexe, pathologie) et aux conditions de prélèvement (heure, tube, type d’anticoagulant, séparation, conservation) peuvent influencer les propriétés des échantillons et impacter les résultats. Ces paramètres, regroupés sous le terme de « préanalytique », sont largement pris en compte dans la routine diagnostique mais sont encore insuffisamment maîtrisés en recherche clinique. Dans une étude menée dans le cadre du groupe coopératif GOELAMS (Groupe Ouest-Est des Leucémies aiguës et Autres Maladies du Sang), nous avons mis en place une étude à la recherche de biomarqueurs sanguins dans les lymphomes B non hodgkiniens et nous avons tout particulièrement pris en compte cette phase préanalytique en explorant diverses pistes avec des solutions adaptées à l’étude multicentrique et à l’organisation des soins en France.
Du prélèvement à la réalisation de l’analyse : le préanalytique
Le préanalytique se définit comme l’ensemble des processus qui se déroulent préalablement à l’analyse biologique proprement dite. Pendant cette étape, de nombreux facteurs peuvent influencer la qualité des échantillons et les résultats biologiques obtenus avec ces échantillons.
Les paramètres identifiés et répertoriés [1, 2] - depuis le design de l’étude, jusqu’au choix de la méthode de collection et de l’analyse proprement dite - sont liés à l’individu lui-même, par exemple son âge, son sexe, l’existence ou non de situations pathologiques, et à l’échantillon, par exemple ses conditions de transport et/ou de conservation. Ces paramètres sont particulièrement importants à identifier et surtout à tracer.
Au cours d’études multicentriques, la plupart des étapes réalisées en amont de l’analyse biologique proprement dite font intervenir différents intervenants, lors de tâches successives, et sur des lieux multiples et le plus souvent éloignés géographiquement
Comment minimiser les facteurs d’influence ?
Dans le cadre d’études multicentriques, il est particulièrement important de mettre en œuvre des outils permettant de tracer les différentes étapes de la vie d’un échantillon, depuis son prélèvement chez le patient jusqu’à la réalisation des analyses. Des procédures adaptées doivent permettre d’obtenir différentes informations :
Avant que le prélèvement ne parvienne au laboratoire, il est nécessaire de collecter un certain nombre d’informations sur le patient telles que âge, sexe, grossesse, alimentation, poids, taille, pathologie, activité corporelle, prise de médicament, et sur le prélèvement (horaire de prélèvement, type d’anticoagulant, nature du tube).
Il faut ensuite collecter des informations sur le transport dont la méthode est conditionnée par la stabilité des différents constituants de l’échantillon. Des éléments tels que la lumière ou la température peuvent influencer fortement la qualité ou les propriétés des échantillons, d’où l’importance de la rapidité des transports, d’une durée de stockage limitée et surtout de l’utilisation de tubes dédiés avec réactifs de conservation ou agents de séparation (mécaniques ou de type gel).
Au laboratoire, en amont de l’analyse, il faut consigner l’heure d’arrivée et/ou de traitement du prélèvement, l’aspect du prélèvement à l’arrivée (hémolyse in vitro, sérum/plasma lipémique, coagulation), les manipulations éventuelles (centrifugation, séparation) et les conditions de stockage.
La traçabilité de ces éléments est assurée par la mise en place de procédures détaillées et d’une fiche de suivi du prélèvement qui accompagnera celui-ci tout au long des étapes préanalytiques.
Dans le domaine de la biologie médicale, la qualité est définie comme l’adéquation entre les moyens mis en œuvre et les informations attendues. En interne, un ensemble de procédures doit être mis en œuvre en vue : (1) d’identifier les biais potentiels ; (2) de limiter leur survenue ; et (3) de permettre un contrôle de la qualité des résultats des analyses au fur et à mesure de leur exécution.
Recherche de biomarqueurs dans les lymphomes B diffus à grandes cellules
Parmi les lymphomes B malins non hodgkiniens (LNH), les lymphomes de haut grade de type B diffus à grandes cellules ou DLBCL (diffuse large B cell lymphoma) représentent 30 % à 40 % des nouveaux diagnostics en hématologie. Ils forment un groupe très hétérogène de maladies tant sur le plan clinique et pronostique que biologique. Le développement des analyses génomiques a permis l’amélioration de nos connaissances sur la physiopathologie de ces maladies, permettant d’identifier parmi les DLBCL différents types de profils d’expression génique, chacun de ces profils pouvant être rattaché à un pronostic distinct [3, 4]. Cependant, ces approches ont fait appel à des explorations larges sur le tissu tumoral d’origine, qui n’est cependant pas toujours facilement disponible pour ces études exhaustives.
Il apparaît donc particulièrement intéressant d’évaluer l’impact de ces pathologies au niveau du sang qui est d’un accès plus facile. Toute modulation de l’équilibre physiologique peut générer une réponse entraînant une modification d’expression de certains gènes et protéines, dont l’expression dans le sang circulant reflétera la réponse de l’organisme face à l’agression et permettra d’identifier des signatures spécifiques d’une situation pathologique donnée et éventuellement de détecter de nouvelles cibles thérapeutiques [2, 5].
Le groupe coopératif GOELAMS a permis la mise en place, au cours du premier semestre 2005, de deux essais thérapeutiques portant respectivement sur les DLBCL disséminés (essai GOELAMS 075) et sur les DLBCL localisés (GOELAMS 0203). Au total, 740 patients seront inclus dans ces deux études par 49 centres investigateurs répartis sur le territoire national français. Les données cliniques et biologiques seront recueillies pour chaque patient dans un cahier électronique standardisé (e-CRF) puis transférées vers un système de gestion de base de données alimenté en temps réel grâce à un navigateur internet.
Des études biologiques spécifiques sont associées à ces protocoles thérapeutiques dont un des objectifs est la recherche, par des analyses de transcriptome et de protéome, de biomarqueurs sanguins diagnostiques et pronostiques dans ces hémopathies. Au cours de la mise en place de ces études, nous avons choisi d’apporter un soin particulier à la traçabilité des étapes préanalytiques afin de connaître les éventuels biais générés au cours de tels larges essais multicentriques. Il était donc nécessaire de proposer des procédures simples à mettre en œuvre par les cliniciens et leurs équipes.
Nous avons ainsi préparé des sets de prélèvement constitués chacun d’une boîte contenant les différents tubes dédiés aux analyses biologiques sur le sang des patients inclus dans ces protocoles 0203 ou 075 (Figure 1). Dans ces kits sont également inclus un système de prélèvement adapté de type Vacutainer®, une feuille de suivi des prélèvement reprenant les procédures nécessaires au bon déroulement de ces étapes préanalytiques et utilisée pour la traçabilité des transports, et enfin un bordereau prépayé pour l’acheminement du set depuis le centre investigateur jusqu’au laboratoire prenant en charge la collection des échantillons. Outre un tube K2-EDTA destiné à l’extraction ADN, ce set contient deux tubes PAXgeneTM (Preanalytix, Qiagen) destinés aux analyses de transcriptome sur sang total et un tube BDTM P100 (Becton Dickinson) spécialement adapté à l’analyse protéomique du plasma. Ce tube contient un anticoagulant K2-EDTA associé à des agents stabilisateurs des protéines et présente un système de séparation mécanique qui, après centrifugation, se positionne à l’interface plasma/cellules. Afin d’optimiser au laboratoire la prise en charge de ces prélèvements, un circuit dédié a été organisé et pris en charge par un technicien de recherche clinique chargé, sous la responsabilité d’un biologiste, du recueil des informations et de la gestion du prélèvement à toutes les étapes de sa mise en collection.
 |
Figure 1. Set de prélèvement sanguin pour les protocoles GOELAMS 0203 et 075. |
Résultats préliminaires - Qualité des échantillons
Nous disposons de résultats préliminaires obtenus sur 71 patients (n = 29 pour le protocole 0203 et n = 42 pour le 075). La moyenne d’âge est de 49,6 ans et le sex ratio H/F de 0,9. Dès leur réception, les tubes EDTA et PAXgeneTM ont été congelés à - 20°C. Les tubes BDTM P100 ont subi une centrifugation à 4000 rpm pendant 10 minutes puis le plasma a été aliquoté et congelé rapidement à - 80°C.
Les délais d’acheminement au laboratoire des sets ont été en moyenne de 32 heures [1-96 heures], avec 11 sets arrivés dans les 2 heures après le prélèvement (J0) et 60 sets dans un temps supérieur à 20 heures (J1), avec un pic de fréquence entre 24 et 30 heures. Ainsi, au total, 67 % des kits parviennent dans un délai inférieur à 30 heures. Les délais les plus longs étaient liés au transport depuis des centres extérieurs souvent éloignés de plusieurs centaines de kilomètres. Une cohorte de 20 plasmas témoins (2 tubes P100 par témoin) a été constituée dont la moitié a été centrifugée dans les 2 heures après le prélèvement et l’autre moitié après 24 heures afin de reproduire les conditions préanalytiques retrouvées chez les patients.
Des anomalies de type coagulation, hémolyse ou défaut de positionnement du système de séparation plasma/cellules ont été répertoriées sur les différents prélèvements (Tableau I). Au total, plus de 82 % des échantillons ne présentaient pas d’anomalies apparentes avant cryopréservation du plasma sur les tubes BDTM P100. Aucune anomalie n’a été identifiée sur les tubes EDTA et PAXgeneTM.
Artéfacts de coagulation, hémolyse et anomalie de positionnement du système de separation dans les tubes de prélèvement BDTM P100 selon le délai entre la prise de sang et la centrifugation des tubes au laboratoire (J1 : moins de 2 heures de délai ; J2 : plus de 24 heures de délai) pour 71 prélèvements réalisés chez des patients et 20 chez des témoins
Les ARN sur sang total ont été extraits selon les recommandations du fabricant à partir de tubes congelés depuis quelques semaines à plus de 6 mois. Nous avons obtenu en moyenne 5 μg d’ARN par tube PAXgeneTM, soit 10 μg par patient. La qualité des ARN extraits a été évaluée par la réalisation d’un profil électrophorétique RNA Nanochip sur un système 2100 Bioanalyzer® (Agilent) (Figure 2) et le calcul du RNA Integrity Number (RIN) par le logiciel 2100 Expert. Le RIN moyen obtenu a été de 7,6 ± 0,67 (6,4-9,6) montrant une qualité satisfaisante pour une analyse de transcriptome sur de tels échantillons de sang total.
 |
Figure 2. Profils électrophorétiques sur nanochips Bioanalyzer 2100® pour différents échantillons représentatifs (RIN moyen de l’ensemble des échantillons = 7,6). RIN : RNA integrity number |
Résultats préliminaires - Analyse protéomique
L’analyse des profils protéiques a été réalisée par une approche SELDI TOF MS (surface enhanced laser desorption/ionisation time of flight, Ciphergen), qui combine la chromatographie de surface à la spectrométrie de masse. Cette technique permet la séparation, la détection et l’analyse de protéines, directement à partir de l’échantillon biologique (ici, du plasma), avec une sensibilité de l’ordre du femtomole. Différentes surfaces de propriétés biochimiques spécifiques sont utilisées dans cette étude. Pour chacune de ces surfaces, (appelée puce à protéines) un tampon de liaison et de lavage spécifique est nécessaire : puce Q10, échangeuses d’anions (tris 0,1M pH8) ; puce CM10, échangeuses de cations (acétate de sodium 0,1M pH4) ; puce IMAC, affines aux métaux, chargées avec du cuivre (phosphate de sodium 0,1M pH7 + NaCl 0,5M) ; puce H4, hydrophobes (acétonitrile 10 % + TFA 0,1 %).
Cinquante-sept patients atteints de lymphomes de haut grade ont été analysés (protocole 0203 n=23 et protocole 075 n=34) et 20 témoins sains appariés en sexe et âge. Après dénaturation (urée 9M + Chaps 2 %), les échantillons de plasma ont été déposés sur les puces au moyen du robot Biomek® 2000 (Beckman). Après plusieurs étapes de lavage et le dépôt d’acide sinapinique (SPA), la puce à protéine a été introduite dans le spectromètre de masse PCS-4000. Sous l’impulsion du rayonnement laser (à 3000 et 5000nJ), les protéines liées à la puce ont été désorbées/ionisées et analysées en terme de masse sur charge (m/z) d’après leur temps de vol pour atteindre le détecteur. Les spectres de masse obtenus ont ensuite été calibrés, normalisés et alignés afin de limiter les biais dus à l’opérateur, à la surface chromatographique ou à l’instrument. L’analyse statistique a été réalisée au moyen du logiciel CiphergenExpress 3.0 selon le principe de la classification hiérarchique (analyse statistique multivariée) : les profils protéiques les plus similaires en terme d’intensité de pics sont groupés afin d’en extraire la meilleure combinaison de marqueurs capables de discriminer les groupes patients (075 et 0203) et témoin ainsi que d’autres marqueurs tels que l’influence du délai d’acheminement, de l’âge, du sexe et de l’hémolyse.
L’influence du délai entre collecte et aliquotage des plasmas a d’abord été étudiée sur la cohorte des témoins et a montré une variabilité des profils (p < 0,05, Figure 3A) avec une nette dégradation de certaines protéines après 24 heures (Figure 3B). Une analyse réalisée sur les plasmas de l’ensemble des échantillons (patients + témoins) montre de même une influence du délai avant congélation (Figure 3C).
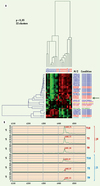  |
Figure 3. Influence du délai d’acheminement des tubes de sang sur le profil protéique plasmatique.A. Clustering hiérarchique montrant une variabilité des profils selon le délai entre le prélèvement sanguin et la congélation des échantillons de plasma témoin (J0 < 2 heures ; J1 = 24 heures). B. Exemple de la protéine de 6 440 Da retenue sur une puce CM10 échangeuse de cations pour les plasmas témoins. C. Exemple de la protéine de 12 577 Da retenue sur une puce Q10 échangeuse d’anions pour les plasmas témoins et patients (p = 0,0000005). |
Nous n’avons pas observé de différence entre les profils protéiques du plasma des patients en fonction de leur âge et l’influence du sexe n’a été retrouvée que dans le protocole 075 avec 43 clusters présentent des p significatifs (p<0,05). Une analyse (non supervisée) menée sur les plasmas de patients montre que les quatre échantillons hémolysés forment un groupe homogène (p<0,03). Les dix clusters les plus significatifs permettant d’établir cette carte correspondant, comme attendu, à l’hémoglobine. Une analyse menée sur les profils des 57 patients et 20 témoins montre l’existence de nombreux clusters differentiels entre les profils protéiques du plasma des patients et ceux des témoins ainsi qu’entre les protocoles 0203 et 075. Parmi les biomarqueurs candidats, nous avons ainsi pu exclure les protéines montrant une instabilité selon différents critères préanalytiques et les études sont actuellement en cours pour identifier des protéines d’intérêt.
Conclusions
La qualité des résultats des analyses biologiques ne dépend pas uniquement de la qualité de la technique mise en œuvre.La préparation de l’échantillon doit répondre à des règles préétablies et la traçabilité des étapes préanalytiques est indispensable à l’interprétation de la phase analytique. Notre expérience au sein du groupe GOELAMS sur les protocoles récemment mis en place dans les lymphomes B agressifs nous a permis de valider des étapes préanalytiques dans des études multicentriques à grande échelle. Nous avons mis en œuvre un circuit de prélèvement auquel nous avons associé une forte traçabilité nous permettant d’identifier et mesurer d’éventuels biais dans les analyses biologiques spécifiques sur les échantillons de patients. La connaissance de l’impact de ces éléments préanalytiques sur les resultants d’analyse nous permet désormais de concentrer nos efforts sur des biomarqueurs candidats, c’est-à-dire dont la variation n’est pas liée à des facteurs extrinsèques à l’échantillon mais à l’existence d’une pathologie, à ses caractéristiques propres et à son retentissement systémique
Remerciements
Ce travail a reçu le soutien financier de la Société Becton Dickinson (pour la constitution des sets de tubes de prélèvements avec les tubes PAXgeneTM et BDTM P100), du Cancéropôle Rhône-Alpes et du CHU de Grenoble (pour les analyses du protéome), ainsi que de la Société Roche (pour les études du transcriptome). Nous remercions également le CHU de Rennes et en particulier le personnel technique du Centre de Ressources Biologiques (CRB) pour leur aide à la constitution des collections d’échantillons.
Références
- Rai AJ, Gelfand CA, Haywood BC, et al. HUPO plasma proteome project specimen collection and handling: towards the standardization of parameters for plasma proteome samples. Proteomics 2005; 5 : 3262–77. [Google Scholar]
- Whitney AR, Diehn M, Popper SJ, et al. Individuality and variation in gene expression patterns in human blood. Proc Natl Acad Sci USA 2003; 100 : 1896–901. [Google Scholar]
- Shipp MA, Ross KN, Tamayo P, et al. Diffuse large B-cell lymphoma outcome prediction by gene-expression profiling and supervised machine learning. Nat Med 2002; 8 : 68–74. [Google Scholar]
- Rosenwald A, Wright G, Chan WC, et al. The use of molecular profiling to predict survival after chemotherapy for diffuse large-B-cell lymphoma. N Engl J Med 2002; 346 : 1937–47. [Google Scholar]
- Liew CC, Ma J, Tang HC, et al. The peripheral blood transcriptome dynamically reflects system wide biology: a potential diagnostic tool. J Lab Clin Med 2006; 147 : 126–32. [Google Scholar]
Liste des tableaux
Artéfacts de coagulation, hémolyse et anomalie de positionnement du système de separation dans les tubes de prélèvement BDTM P100 selon le délai entre la prise de sang et la centrifugation des tubes au laboratoire (J1 : moins de 2 heures de délai ; J2 : plus de 24 heures de délai) pour 71 prélèvements réalisés chez des patients et 20 chez des témoins
Liste des figures
 |
Figure 1. Set de prélèvement sanguin pour les protocoles GOELAMS 0203 et 075. |
| Dans le texte | |
 |
Figure 2. Profils électrophorétiques sur nanochips Bioanalyzer 2100® pour différents échantillons représentatifs (RIN moyen de l’ensemble des échantillons = 7,6). RIN : RNA integrity number |
| Dans le texte | |
  |
Figure 3. Influence du délai d’acheminement des tubes de sang sur le profil protéique plasmatique.A. Clustering hiérarchique montrant une variabilité des profils selon le délai entre le prélèvement sanguin et la congélation des échantillons de plasma témoin (J0 < 2 heures ; J1 = 24 heures). B. Exemple de la protéine de 6 440 Da retenue sur une puce CM10 échangeuse de cations pour les plasmas témoins. C. Exemple de la protéine de 12 577 Da retenue sur une puce Q10 échangeuse d’anions pour les plasmas témoins et patients (p = 0,0000005). |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




