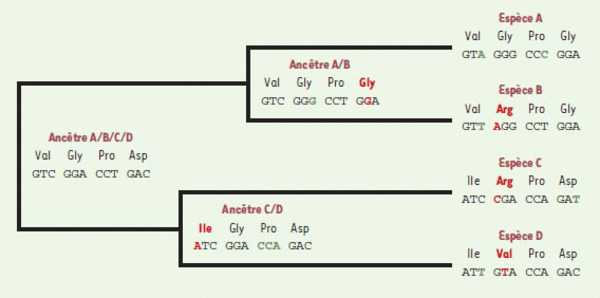
Figure 3.
Télécharger l'image originale
Estimation de la sélection au niveau moléculaire. Dans cet exemple simplifié, on considère quatre espèces actuelles (A, B, C, D) et quatre codons. Après avoir inféré les relations phylogénétiques entre ces espèces, on infère les séquences chez les trois ancêtres communs. Ensuite, pour chaque codon, on compte le nombre de substitutions synonymes (s), c’est-à-dire qui ne change pas l’acide aminé (indiqué en vert) et non-synonymes (ns), c’est-à-dire qui change l’acide aminé (indiqué en rouge). Dans le cas du premier codon, il y a eu trois mutations synonymes (s = 3) et une mutation non-synonyme (ns = 1). Comme on s’intéresse à la fréquence des changements, il faut corriger par le nombre potentiel de mutations synonymes (Ns) et non-synonymes (Nns). Par exemple, pour la valine (codé par GTN), il y a trois mutations synonymes possibles et six non-synonymes, alors que, pour le tryptophane (codé uniquement par UGG), il n’y a aucune mutation synonyme. Grâce à des méthodes plus ou moins sophistiquées, on calcule Ks (s/Ns) et Kns (ns / Nns). Ks est un estimateur du taux de mutation, Kns est un estimateur du taux de changement des acides aminés. Pour le premier codon, Kns est inférieur à Ks, ce qui indique une sélection négative qui empêche la fixation de certains changements d’acides aminés. Pour le deuxième codon, Kns est supérieur à Ks, ce qui indique une sélection positive qui favorise des changements d’acides aminés.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




