| Numéro |
Med Sci (Paris)
Volume 21, Numéro 8-9, Août–Septembre 2005
|
|
|---|---|---|
| Page(s) | 722 - 729 | |
| Section | M/S revues | |
| DOI | https://doi.org/10.1051/medsci/2005218-9722 | |
| Publié en ligne | 15 août 2005 | |
Protéomique clinique : vers la détection précoce des cancers ?
Clinical proteomics: towards early detection of cancers
Laboratoire de Biologie cellulaire et hormonale, Inserm U.540 Hôpital Arnaud de Villeneuve, 191, avenue du Doyen Giraud, 34295 Montpellier Cedex 5, France
Bien que des progrès considérables aient été réalisés ces dernières années dans la comprehension des processus tumoraux, les retombées cliniques pour le diagnostic précoce des tumeurs demeurent limitées. L’avènement récent de nouveaux outils de protéomique, faisant appel à des techniques de biopuces à protéines, a totalement modifié les perspectives dans ce domaine et permis l’émergence du concept de profil d’expression protéique, véritable signature moléculaire de la tumeur. Sur la base de ce profil, la detection des cancers pendant la phase asymptomatique pourrait constituer une réelle avancée pour le patient et sa prise en charge thérapeutique. Cet article présente la plate-forme protéomique SELDI-TOF et ses premières applications en cancérologie. Toutefois, si ces applications laissent présager de multiples développements en cancérologie clinique, de nombreuses ameliorations restent à faire avant leur implantation en routine hospitalière.
Abstract
A key challenge in clinical proteomic of cancer is the identification of biomarkers that would allow early detection, diagnosis and monitor progression of the disease to improve long-term survival of patients. Recent advances in proteomic instrumentation and computational methodologies offer unique chance to rapidly identify these new candidate markers or pattern of markers. The combination of retentate affinity chromatography and surfaced-enhanced laser desorption/ionization time-of-flight (SELDI-TOF) mass spectrometry is one of the most interesting new approaches for cancer diagnostic using proteomic profiling. This review aims to summarize the results of studies that have used this new technology method for the early diagnosis of human cancer. Despite promising results, the use of the proteomic profiling as a diagnostic tool brought some controversies and technical problems and still requires some efforts to be standardized and validated.
© 2005 médecine/sciences - Inserm / SRMS
En dépit d’avancées significatives, nos connaissances sur la physiopathologie des cancers sont encore parcellaires et les retombées pratiques de ces recherches dans le domaine du dépistage, du diagnostic, du pronostic ou des traitements sont encore trop limitées. La compréhension des différentes étapes permettant le passage d’une lésion localisée vers une tumeur invasive est particulièrement importante car elle conditionne la prévention et la prise en charge des cancers invasifs dont la mortalité reste élevée (Figure 1). C’est pourquoi, il est nécessaire d’élaborer de nouvelles stratégies pour identifier les protéines impliquées dans des mécanismes physiologiques ou pathologiques en ligne directe avec l’initiation et/ou la progression tumorale. Ces approches se focalisent non plus sur l’identification d’un marqueur unique, mais sur plusieurs marqueurs co-exprimés, débouchant ainsi sur l’établissement d’un profil d’expression ou d’une carte d’identité pour chaque tumeur. Une telle signature moléculaire associée aux données cliniques et anatomopathologiques devrait permettre le dépistage des stades précoces de cancer avant -l’apparition des signes cliniques. Récemment, les progrès en nanotechnologies fondés sur des méthodes de chromatographie liquide ou de surface ont permis l’émergence de nouvelles techniques d’analyse protéomique appliquées aux pathologies humaines [1–6]. Dans cet article, nous ne détaillerons pas l’ensemble de ces technologies. En revanche, nous nous focaliserons sur une plate-forme qui associe la chromatographie d’affinité de surface (ProteinChip®) et la spectrométrie de masse et qui nous semble, à ce jour, la mieux adaptée aux contraintes liées à la protéomique clinique.
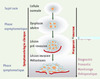 |
Figure 1. Diagramme de la progression tumorale. Il est généralement admis que l’oncogenèse suit un schéma progressif au cours duquel s’accumulent des altérations morphologiques et moléculaires aboutissant à un dérèglement du comportement de la cellule avec des processus de prolifération et d’invasion (franchissement de la membrane basale, envahissement loco-régional, métastase). Par définition, les marqueurs tumoraux sont les indicateurs biologiques de ces changements moléculaires survenant lors du processus tumoral. Ils sont généralement le reflet d’altérations génétiques comme des mutations, des pertes d’hétérozygotie ou des remaniements chromosomiques qui, au final, vont affecter un certain nombre de gènes actifs et leurs produits. La caractérisation moléculaire et fonctionnelle de l’ensemble de ces altérations et de leurs effets permet d’envisager l’établissement d’une signature moléculaire des tumeurs à un stade précis du processus tumoral. En théorie, ces marqueurs peuvent apporter des éléments d’information importants pour le dépistage d’un cancer pendant la phase asymptomatique, pour son diagnostic et son pronostic, mais aussi pour la surveillance thérapeutique à un stade plus avancé de la maladie. |
Marqueurs biologiques sériques et détection des cancers
Par définition, les marqueurs tumoraux ne sont pas statiques et leur expression varie au cours du temps en fonction de l’état général du patient, du processus tumoral ou des traitements (Figure 1). En pratique, bien qu’un grand nombre de marqueurs présomptifs ait été identifié, peu d’entre eux ont été promus et recommandés pour une utilisation diagnostique dans la population générale (Tableau I). Leur utilisation est actuellement limitée le plus souvent à l’établissement du bilan initial du cancer (valeur de référence), à l’évaluation de l’efficacité thérapeutique et à la surveillance d’une récidive. La performance d’un test diagnostic est fondée sur sa capacité à classer correctement les individus dans les différents sous-groupes cliniques (sujets sains versus sujets malades). Le test idéal devrait être totalement négatif chez les sujets en bonne santé (l00 % de spécificité) et totalement positif pour un type de tumeur donnée (100 % de sensibilité). Par ailleurs, ces marqueurs devraient montrer une corrélation nette entre leur concentration sérique et la taille de la tumeur. Actuellement, il existe peu de marqueurs possédant une spécificité et une sensibilité suffisantes pour avoir une utilité en clinique dans le diagnostic des stades précoces des cancers. Seuls l’antigène prostatique spécifique (PSA) et la thyrocalcitonine sont utilisés en routine, respectivement pour le dépistage et le diagnostic des cancers de la prostate et des formes familiales de cancers médullaires de la thyroïde. Ces deux seuls exemples illustrent la difficulté d’identifier des marqueurs de lésions cancéreuses pré-invasives qui sont, par définition, des tumeurs très localisées ne franchissant pas la membrane basale. Ces marqueurs correspondent généralement à des protéines sécrétées directement par les cellules cancéreuses ou par le microenvironnement tumoral dans un espace vasculaire de diffusion à proximité de la tumeur. Toute la difficulté consiste à les identifier et à les doser dans la circulation générale.
Marqueurs utilisés dans le dépistage (D), le diagnostic (Dg) et/ou la surveillance (S) de cancers.
Le SELDI-TOF, un nouvel outil technologique en protéomique clinique ?
Récemment, des avancées méthodologiques en spectrométrie de masse ont ouvert des perspectives intéressantes pour l’identification de nouveaux marqueurs tumoraux. Parmi celles-ci, la plate-forme SELDI-TOF (surfaced-enhanced laser desorption/ionization time-of-flight) présente de nombreux avantages par rapport aux autres techniques, notamment en raison de sa simplicité d’utilisation, de sa sensibilité et de sa capacité d’analyse à haut débit des échantillons (près de 800/jour). Cette technique permet la séparation, la détection et l’analyse de protéines, directement à partir de l’échantillon biologique avec une sensibilité de l’ordre de la femtomole (Figure 2). Elle est toutefois limitée par la gamme de masse protéique analysable, avec une très bonne détection pour les protéines inférieures à 20-30 kDa et une sensibilité moindre pour les protéines de plus hautes masses. En combinant les différentes surfaces chromatographiques disponibles, on obtient une vue d’ensemble des peptides et des protéines présents dans un échantillon donné. L’analyse différentielle et statistique de l’ensemble des données fournies par les profils protéiques des groupes témoins (sujets sains sans cancer) et pathologiques est une des étapes les plus importantes de ces études protéomiques. Pour cela, il existe une multitude d’algorithmes informatiques fondés sur des analyses statistiques multivariées (analyse discriminante, classification hiérarchique…). Ces algorithmes permettent d’analyser des données complexes et d’extraire des spectres de masse la meilleure combinaison de marqueurs capables de discriminer chacun des deux groupes. Par la suite, l’identification des marqueurs potentiels nécessite des étapes supplémentaires de purification par des méthodes électrophorétiques ou chromatographiques avant de caractériser précisément la protéine d’intérêt par spectrométrie de masse.
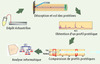 |
Figure 2. Principe de la technologie SELDI-TOF (surface-enhanced laser desorption and ionization time-of-flight). Cette plate-forme combine la chromatographie de surface à la spectrométrie de masse. En pratique, quelques microgrammes de protéines issues d’échantillons variés (liquides biologiques, cellules ou tissus) sont directement adsorbés sur une surface de 2 mm2 présentant des propriétés chromatographiques variées. En fonction de la surface chromatographique choisie, le mélange protéique subit un fractionnement qui dépend des propriétés chimiques (anionique, cationique, hydrophobique, hydrophilique, affinité aux métaux) ou biologiques des protéines (liaison à un anticorps, un peptide, un récepteur, un ligand ou un acide nucléique). Après une série d’étapes de lavage, les protéines retenues sont recouvertes d’une matrice qui va absorber l’excès d’énergie lors de l’ionisation de l’échantillon par désorption au laser et permettre l’analyse en spectrométrie de masse en temps de vol (la spectrométrie de masse en temps de vol consiste à accélérer tous les ions à la même énergie cinétique. Leurs vitesses étant inversement proportionnelles à la racine carrée de leurs masses, les ions plus légers arrivent au détecteur plus rapidement que les ions plus lourds, ce qui permet de les séparer). Au final, on obtient un profil protéique représentant les différentes protéines en fonction du rapport masse sur charge (M/Z). Ces profils sont ensuite normalisés et calibrés puis analysés de manière différentielle par des algorithmes informatiques variés afin d’établir un profil protéique spécifique. |
Applications en cancérologie
Le concept de signature tumorale ou profil protéique
Dès les premiers résultats issus de la technologie SELDI-TOF, de nombreux auteurs ont suggéré qu’un simple profil protéique spécifique pourrait être suffisant pour le diagnostic précoce de pathologies cancéreuses.Un des premiers groupes à proposer cette hypothèse a été l’équipe de Pétricoin et Liotta qui, en 2002, a réalisé une étude comparant 50 femmes atteintes de cancer de l’ovaire à différents stades et 50 femmes témoins [7]. Par une approche SELDI-TOF, les auteurs ont pu identifier un profil protéique constitué d’une combinaison de cinq marqueurs permettant de dépister, à partir d’un simple échantillon sanguin, des cancers de l’ovaire dès le stade I (stade le plus précoce) avec une sensibilité de 100 %, une spécificité de 95 % et une valeur prédictive positive de 94 %. Au moment de leur parution, ces résultats ont été présentés comme une avancée significative pour le diagnostic du cancer de l’ovaire, cancer souvent détecté à des stades avancés du processus tumoral (stade métastatique). Dans la même année, il a été décrit pour le cancer de la prostate des marqueurs diagnostiques sériques précoces présentant une meilleure spécificité que le PSA et permettant de différencier des hypertrophies bénignes des stades cancéreux (Tableau II). Selon ces études, les profils protéiques obtenus à partir de plusieurs centaines de sérum donnent une spécificité comprise entre 85 et 100 %, supérieure au 71 % du PSA. Des résultats similaires ont été obtenus par la suite pour les cancers du sein, de la vessie, du pancréas, du rein ou du foie (Tableau II), à partir d’échantillons variés comme du sérum, des biopsies tissulaires, des tissus microdisséqués, de l’urine ou d’autres fluides biologiques.
Résumé des différentes études SELDI-TOF sur la détection de marqueurs tumoraux.
L’ensemble de ces résultats a suscité un intérêt pour la protéomique clinique, avec notamment des perspectives à terme pour le diagnostic des formes précoces des cancers. Néanmoins, si ces études ont mis en avant l’intérêt de l’établissement d’une signature tumorale comme outil diagnostique, elles ont également soulevé de nombreuses critiques sur les limitations réelles ou supposées de l’approche SELDI-TOF. Ainsi, une méta-analyse réalisée à partir des données issues de cinq publications indépendantes suggère que cette approche est peu reproductible d’un laboratoire à l’autre [8]. Dans cette analyse, les profils protéiques permettant la discrimination de sérums témoins et de sérums de patients atteints de cancer de la prostate s’avèrent différents d’une publication à l’autre. En fait, il est apparu que l’établissement des profils protéiques était très dépendant des algorithmes informatiques utilisés dans les différentes études. Si ce manque de reproductibilité ne porte pas à conséquence dans la première phase de découverte des profils protéiques diagnostiques, il est clair que des étapes de validation et surtout de standardisation seront nécessaires pour les rendre utilisables en clinique à l’instar de ce qui est fait pour les tests immunologiques. Une étude récente sur le cancer de l’ovaire prend en compte ces paramètres de validation et de standardisation [9]. Cette étude multicentrique implique cinq laboratoires différents avec des protocoles standardisés permettant une validation croisée et indépendante des résultats d’un laboratoire à l’autre. Près de 500 sérums ont ainsi été analysés par SELDI-TOF lors des différentes phases de découverte et de validation des profils protéiques. Les résultats montrent qu’une combinaison de trois marqueurs permet la détection des stades précoces des cancers de l’ovaire, suggérant que l’établissement d’un profil protéique spécifique peut être considéré comme fiable et reproductible si le protocole utilisé pour le recueil des échantillons, leur analyse et l’interprétation des résultats, est parfaitement défini.
Signature tumorale versus marqueurs biologiques
Jusqu’à présent, peu d’efforts ont été faits pour l’identification précise des protéines spécifiques constituant la signature tumorale obtenue par SELDI-TOF. Bien que celle-ci ne soit pas un prérequis pour une utilité en clinique, elle présente néanmoins plusieurs intérêts. En effet, lors de la phase de découverte des biomarqueurs, l’identification des protéines candidates permet de réaliser une étape de validation supplémentaire par des méthodes indépendantes de type immunologique (ELISA), et donc d’augmenter la valeur prédictive de la signature moléculaire [9]. Par ailleurs, la caractérisation de ces marqueurs potentiels devrait permettre, à terme, de mieux comprendre les bases moléculaires de la progression tumorale. De façon étonnante, dans les quelques cas où la caractérisation des marqueurs a été effectuée, les protéines identifiées correspondent à des protéines majoritaires du sérum comme l’apolipoprotéine A1, la transthyrétine, l’α-antitrypsine ou l’α-haptoglobine [9]. Une des questions qui se pose alors, est de savoir si ces marqueurs voient leur expression altérée spécifiquement en réponse à la prolifération tumorale ou de manière non spécifique en réponse à des épiphénomènes tels que l’état général du patient ou des processus inflammatoires [10–12]. Pour certains de ces marqueurs comme l’apolipoprotéine A1 et la transthyrétine, l’altération de leur expression semble spécifique d’un type de cancer donné. En effet, leur expression est altérée spécifiquement chez des patientes atteintes de cancer de l’ovaire alors qu’elle ne varie pas dans le cas de cancer du sein, de la prostate ou du colon [9]. Dans ce cas, comment rendre compte de cette spécificité tissulaire pour des protéines ubiquitaires du sérum ? Une des explications possibles est fondée sur l’observation selon laquelle certains des marqueurs identifiés par SELDI-TOF sont des formes clivées de protéines sériques de plus haut poids moléculaire [9]. Liotta et al. ont suggéré que l’abondance de ces marqueurs clivés était le reflet direct d’événements pathologiques [13, 14]. Dans cette hypothèse, le microenvironnement tumoral serait capable de produire une signature protéique unique détectable dans le sang, une fois les protéines passées dans la circulation générale. Plusieurs études soutiennent cette hypothèse et montrent que l’équilibre entre des protéases et leurs inhibiteurs cellulaires est modifié dans le sérum et le tissu des patients en réponse à la prolifération tumorale. Ainsi, différentes familles de protéases, comme les métallo-protéases ou les kallikréines plasmatiques et tissulaires, voient leur expression augmenter ou diminuer dans différents cancers [15–17]. Ces modifications de l’expression auraient alors un retentissement direct sur la capacité de ces protéases à cliver des protéines sériques et à produire ainsi une signature moléculaire spécifique. Bien que séduisante, cette hypothèse a fait l’objet d’une controverse récente sur la capacité de l’approche SELDI-TOF à détecter des protéines très faiblement exprimées dans le sérum [18, 19]. C’est le cas, par exemple, du PSA qui fait partie des protéases dont l’expression augmente dans les cancers de la prostate [17] et qui n’a cependant jamais été identifiée dans les différentes études par SELDI-TOF de ce cancer. Cette limitation ne remet cependant pas en cause l’intérêt du SELDI comme plate-forme de protéomique clinique. Il s’agirait non pas d’une limitation de la technologie SELDI, mais plutôt d’une limitation des techniques de préparation de l’échantillon avant toute analyse protéomique quelle qu’elle soit. La Figure 3 illustre l’obstacle majeur auquel est confrontée l’analyse protéomique du sérum, à savoir la dynamique d’expression des protéines sériques. Il existe plusieurs méthodes pour augmenter la concentration relative des protéines faiblement représentées dans le sérum, telles que le pré-fractionnement, la chromatographie, la précipitation sélective ou la déplétion des protéines les plus majoritaires [20–23]. Pour être efficaces, ces techniques doivent enrichir les protéines peu représentées, sans pour autant introduire de biais supplémentaire dans l’analyse protéomique ultérieure. La détection de cette fraction du sérum, véritable face cachée de l’iceberg, est un des défis technologiques qu’il faudra relever pour assurer des développements importants dans le diagnostic des formes précoces de cancers.
 |
Figure 3. Dynamique d’expression des protéines sériques. L’albumine et les immunoglobulines à elles seules représentent près de 90 % des protéines totales du sérum. Le rapport entre ces protéines majoritaires et les protéines les plus faiblement représentées peut alors atteindre 106 à 109. La présence de protéines majoritaires rend la détection des marqueurs diagnostiques classiques comme le PSA (prostate specific antigen) ou l’ACE (antigène carcino-embryonnaire) très difficile malgré une bonne sensibilité de la technique SELDI-TOF. |
Conclusions
Les données présentées dans cet article illustrent l’intérêt du concept de signature moléculaire tumorale comme nouvel outil de diagnostic des cancers. Même si des progrès restent à faire, ces résultats laissent présager de nombreux développements de la protéomique clinique en pathologie cancéreuse. Ainsi, les tests biologiques du futur pourraient permettre de détecter des lésions pré-cancéreuses dans la population générale à partir d’une combinaison de marqueurs déterminés. De tels développements pourraient avoir un impact très important en clinique, notamment en améliorant le diagnostic précoce des cancers et en permettant de mieux caractériser des groupes à risques évolutifs pour leur proposer des thérapeutiques préventives adaptées.
Références
- Lescuyer P, Chevallet M, Rabilloud T. Concepts and therapeutic perspectives of proteomics. Med Sci (Paris) 2004; 20 : 587–92. [Google Scholar]
- Lim MS, Elenitoba-Johnson KS. Proteomics in pathology research. Lab Invest 2004; 84 : 1227–44. [Google Scholar]
- Chong BE, Lubman DM, Rosenspire A, Miller F. Protein profiles and identification of high performance liquid chromatography isolated proteins of cancer cell lines using matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Commun Mass Spectrom 1998; 12 : 1986–93. [Google Scholar]
- O’Neil KA, Miller FR, Barder TJ, Lubman DM. Profiling the progression of cancer: separation of microsomal proteins in MCF10 breast epithelial cell lines using nonporous chromatophoresis. Proteomics 2003; 3 : 1256–69. [Google Scholar]
- Zhu K, Kim J, Yoo C, et al. High sequence coverage of proteins isolated from liquid separations of breast cancer cells using capillary electrophoresis-time-of-flight MS and MALDI-TOF MS mapping. Anal Chem 2003; 75 : 6209–17. [Google Scholar]
- Issaq HJ, Veenstra TD, Conrads TP, Felschow D. The SELDI-TOF MS approach to proteomics : protein profiling and biomarker identification. Biochem Biophys Res Commun 2002; 292 : 587–92. [Google Scholar]
- Petricoin EF, Ardekani AM, Hitt BA, et al. Use of proteomic patterns in serum to identify ovarian cancer. Lancet 2002; 359 : 572–7. [Google Scholar]
- Diamandis EP. Mass spectrometry as a diagnostic and a cancer biomarker discovery tool : opportunities and potential limitations. Mol Cell Proteomics 2004; 3 : 367–78. [Google Scholar]
- Zhang Z, Bast RC, Jr., Yu Y, et al. Three biomarkers identified from serum proteomic analysis for the detection of early stage ovarian cancer. Cancer Res 2004; 64 : 5882–90. [Google Scholar]
- Diamandis EP. Proteomic patterns in serum and identification of ovarian cancer. Lancet 2002; 360 : 170 ; author reply 170–1. [Google Scholar]
- Diamandis EP. Analysis of serum proteomic patterns for early cancer diagnosis : drawing attention to potential problems. J Natl Cancer Inst 2004; 96 : 353–6. [Google Scholar]
- Coussens LM, Werb Z. Inflammation and cancer. Nature 2002; 420 : 860–7. [Google Scholar]
- Mehta AI, Ross S, Lowenthal MS, et al. Biomarker amplification by serum carrier protein binding. Dis Markers 2003; 19 : 1–10. [Google Scholar]
- Liotta LA, Ferrari M, Petricoin E. Clinical proteomics: written in blood. Nature 2003; 425 : 905. [Google Scholar]
- Mok SC, Chao J, Skates S, et al. Prostasin, a potential serum marker for ovarian cancer: identification through microarray technology. J Natl Cancer Inst 2001; 93 : 1458–64. [Google Scholar]
- Matrisian LM, Sledge GW, Jr., Mohla S. Extracellular proteolysis and cancer: meeting summary and future directions. Cancer Res 2003; 63 : 6105–9. [Google Scholar]
- Yousef GM, Diamandis EP. The new human tissue kallikrein gene family : structure, function, and association to disease. Endocrinol Rev 2001; 22 : 184–204. [Google Scholar]
- Petricoin E, 3rd, Liotta LA. Counterpoint : the vision for a new diagnostic paradigm. Clin Chem 2003; 49 : 1276–8. [Google Scholar]
- Diamandis EP. Point : proteomic patterns in biological fluids : do they represent the future of cancer diagnostics ? Clin Chem 2003; 49: 1272–5. [Google Scholar]
- Solassol J, Marin P, Demettre E, et al. Proteomic detection of prostate-specific antigen using a serum fractionation procedure : potential implication for new low-abundance cancer biomarkers detection. Anal Biochem 2005; 338 : 26–31. [Google Scholar]
- Merrell K, Southwick K, Graves SW, et al. Analysis of low-abundance, low-molecular-weight serum proteins using mass spectrometry. J Biomol Tech 2004; 15 : 238–48. [Google Scholar]
- Wang YY, Cheng P, Chan DW. A simple affinity spin tube filter method for removing high-abundant common proteins or enriching low-abundant biomarkers for serum proteomic analysis. Proteomics 2003; 3 : 243–8. [Google Scholar]
- Govorukhina NI, Keizer-Gunnink A, Van der Zee AG, et al. Sample preparation of human serum for the analysis of tumor markers. Comparison of different approaches for albumin and gamma-globulin depletion. J Chromatogr A 2003; 1009 : 171–8. [Google Scholar]
- Petricoin EF, 3rd, Ornstein DK, Paweletz CP, et al. Serum proteomic patterns for detection of prostate cancer. J Natl Cancer Inst 2002; 94 : 1576–8. [Google Scholar]
- Kozak KR, Amneus MW, Pusey SM, et al. Identification of biomarkers for ovarian cancer using strong anion-exchange ProteinChips : potential use in diagnosis and prognosis. Proc Natl Acad Sci USA 2003; 100 : 12343–8. [Google Scholar]
- Ye B, Cramer DW, Skates SJ, et al. Haptoglobin-alpha subunit as potential serum biomarker in ovarian cancer : identification and characterization using proteomic profiling and mass spectrometry. Clin Cancer Res 2003; 9 : 2904–11. [Google Scholar]
- Adam BL, Qu Y, Davis JW, et al. Serum protein fingerprinting coupled with a pattern-matching algorithm distinguishes prostate cancer from benign prostate hyperplasia and healthy men. Cancer Res 2002; 62 : 3609–14. [Google Scholar]
- Qu Y, Adam BL, Yasui Y, et al. Boosted decision tree analysis of surface-enhanced laser desorption/ionization mass spectral serum profiles discriminates prostate cancer from noncancer patients. Clin Chem 2002; 48 : 1835–43. [Google Scholar]
- Banez LL, Prasanna P, Sun L, et al. Diagnostic potential of serum proteomic patterns in prostate cancer. J Urol 2003; 170 : 442–6. [Google Scholar]
- Lehrer S, Roboz J, Ding H, et al. Putative protein markers in the sera of men with prostatic neoplasms. BJU Int 2003; 92 : 223–5. [Google Scholar]
- Hlavaty JJ, Partin AW, Shue MJ, et al. Identification and preliminary clinical evaluation of a 50.8-kDa serum marker for prostate cancer. Urology 2003; 61 : 1261–5. [Google Scholar]
- Cazares LH, Adam BL, Ward MD, et al. Normal, benign, preneoplastic, and malignant prostate cells have distinct protein expression profiles resolved by surface enhanced laser desorption/ionization mass spectrometry. Clin Cancer Res 2002; 8 : 2541–52. [Google Scholar]
- Wellmann A, Wollscheid V, Lu H, et al. Analysis of microdissected prostate tissue with ProteinChip arrays: a way to new insights into carcinogenesis and to diagnostic tools. Int J Mol Med 2002; 9 : 341–7. [Google Scholar]
- Koopmann J, Zhang Z, White N, et al. Serum diagnosis of pancreatic adenocarcinoma using surface-enhanced laser desorption and ionization mass spectrometry. Clin Cancer Res 2004; 10 : 860–8. [Google Scholar]
- Rosty C, Christa L, Kuzdzal S, et al. Identification of hepatocarcinoma-intestine-pancreas/pancreatitis-associated protein I as a biomarker for pancreatic ductal adenocarcinoma by protein biochip technology. Cancer Res 2002; 62 : 1868–75. [Google Scholar]
- Vlahou A, Laronga C, Wilson L, et al. A novel approach toward development of a rapid blood test for breast cancer. Clin Breast Cancer 2003; 4 : 203–9. [Google Scholar]
- Li J, Zhang Z, Rosenzweig J, et al. Proteomics and bioinformatics approaches for identification of serum biomarkers to detect breast cancer. Clin Chem 2002; 48 : 1296–304. [Google Scholar]
- Becker S, Cazares LH, Watson P, et al. Surfaced-enhanced laser desorption/ionization time-of-flight (SELDI-TOF) differentiation of serum protein profiles of BRCA-1 and sporadic breast cancer. Ann Surg Oncol 2004; 11 : 907–14. [Google Scholar]
- Pusztai L, Gregory BW, Baggerly KA, et al. Pharmacoproteomic analysis of prechemotherapy and postchemotherapy plasma samples from patients receiving neoadjuvant or adjuvant chemotherapy for breast carcinoma. Cancer 2004; 100 : 1814–22. [Google Scholar]
- Paweletz CP, Trock B, Pennanen M, et al. Proteomic patterns of nipple aspirate fluids obtained by SELDI-TOF : potential for new biomarkers to aid in the diagnosis of breast cancer. Dis Markers 2001; 17 : 301–7. [Google Scholar]
- Sauter ER, Shan S, Hewett JE, et al. Proteomic analysis of nipple aspirate fluid using SELDI-TOF-MS. Int J Cancer 2004; 17 : 17. [Google Scholar]
- Kuerer HM, Coombes KR, Chen JN, et al. Association between ductal fluid proteomic expression profiles and the presence of lymph node metastases in women with breast cancer. Surgery 2004; 136 : 1061–9. [Google Scholar]
- Zhukov TA, Johanson RA, Cantor AB, et al. Discovery of distinct protein profiles specific for lung tumors and pre-malignant lung lesions by SELDI mass spectrometry. Lung Cancer 2003; 40 : 267–79. [Google Scholar]
- Xiao X, Liu D, Tang Y, et al. Development of proteomic patterns for detecting lung cancer. Dis Markers 2003; 19 : 33–9. [Google Scholar]
- Rogers MA, Clarke P, Noble J, et al. Proteomic profiling of urinary proteins in renal cancer by surface enhanced laser desorption ionization and neural-network analysis : identification of key issues affecting potential clinical utility. Cancer Res 2003; 63 : 6971–83. [Google Scholar]
- Won Y, Song HJ, Kang TW, et al. Pattern analysis of serum proteome distinguishes renal cell carcinoma from other urologic diseases and healthy persons. Proteomics 2003; 3 : 2310–6. [Google Scholar]
- Soltys SG, Le QT, Shi G, et al. The use of plasma surface-enhanced laser desorption/ionization time-of-flight mass spectrometry proteomic patterns for detection of head and neck squamous cell cancers. Clin Cancer Res 2004; 10 : 4806–12. [Google Scholar]
- Wadsworth JT, Somers KD, Stack BC Jr, et al. Identification of patients with head and neck cancer using serum protein profiles. Arch Otolaryngol Head Neck Surg 2004; 130 : 98–104. [Google Scholar]
- Melle C, Ernst G, Schimmel B, et al. Biomarker discovery and identification in laser microdissected head and neck squamous cell carcinoma with ProteinChip(R) technology, two-dimensional gel electrophoresis, tandem mass spectrometry, and immunohistochemistry. Mol Cell Proteomics 2003; 2 : 443–52. [Google Scholar]
- Melle C, Kaufmann R, Hommann M, et al. Proteomic profiling in microdissected hepatocellular carcinoma tissue using ProteinChip technology. Int J Oncol 2004; 24 : 885–91. [Google Scholar]
- Wilson LL, Tran L, Morton DL, Hoon DS. Detection of differentially expressed proteins in early-stage melanoma patients using SELDI-TOF mass spectrometry. Ann NY Acad Sci 2004; 1022 : 317–22. [Google Scholar]
- Vlahou A, Schellhammer PF, Mendrinos S, et al. Development of a novel proteomic approach for the detection of transitional cell carcinoma of the bladder in urine. Am J Pathol 2001; 158 : 1491–502. [Google Scholar]
- Krieg RC, Fogt F, Braunschweig T, et al. ProteinChip array analysis of microdissected colorectal carcinoma and associated tumor stroma shows specific protein bands in the 3.4 to 3.6 kDa range. Anticancer Res 2004; 24 : 1791–6. [Google Scholar]
- Shiwa M, Nishimura Y, Wakatabe R, et al. Rapid discovery and identification of a tissue-specific tumor biomarker from 39 human cancer cell lines using the SELDI ProteinChip platform. Biochem Biophys Res Commun 2003; 309 : 18–25. [Google Scholar]
- Chen YD, Zheng S, Yu JK, Hu X. Artificial neural networks analysis of surface-enhanced laser desorption/ionization mass spectra of serum protein pattern distinguishes colorectal cancer from healthy population. Clin Cancer Res 2004; 10 : 8380–5. [Google Scholar]
- Wong YF, Cheung TH, Lo KW, et al. Protein profiling of cervical cancer by protein-biochips: proteomic scoring to discriminate cervical cancer from normal cervix. Cancer Lett 2004; 211 : 227–34. [Google Scholar]
Liste des tableaux
Marqueurs utilisés dans le dépistage (D), le diagnostic (Dg) et/ou la surveillance (S) de cancers.
Résumé des différentes études SELDI-TOF sur la détection de marqueurs tumoraux.
Liste des figures
 |
Figure 1. Diagramme de la progression tumorale. Il est généralement admis que l’oncogenèse suit un schéma progressif au cours duquel s’accumulent des altérations morphologiques et moléculaires aboutissant à un dérèglement du comportement de la cellule avec des processus de prolifération et d’invasion (franchissement de la membrane basale, envahissement loco-régional, métastase). Par définition, les marqueurs tumoraux sont les indicateurs biologiques de ces changements moléculaires survenant lors du processus tumoral. Ils sont généralement le reflet d’altérations génétiques comme des mutations, des pertes d’hétérozygotie ou des remaniements chromosomiques qui, au final, vont affecter un certain nombre de gènes actifs et leurs produits. La caractérisation moléculaire et fonctionnelle de l’ensemble de ces altérations et de leurs effets permet d’envisager l’établissement d’une signature moléculaire des tumeurs à un stade précis du processus tumoral. En théorie, ces marqueurs peuvent apporter des éléments d’information importants pour le dépistage d’un cancer pendant la phase asymptomatique, pour son diagnostic et son pronostic, mais aussi pour la surveillance thérapeutique à un stade plus avancé de la maladie. |
| Dans le texte | |
 |
Figure 2. Principe de la technologie SELDI-TOF (surface-enhanced laser desorption and ionization time-of-flight). Cette plate-forme combine la chromatographie de surface à la spectrométrie de masse. En pratique, quelques microgrammes de protéines issues d’échantillons variés (liquides biologiques, cellules ou tissus) sont directement adsorbés sur une surface de 2 mm2 présentant des propriétés chromatographiques variées. En fonction de la surface chromatographique choisie, le mélange protéique subit un fractionnement qui dépend des propriétés chimiques (anionique, cationique, hydrophobique, hydrophilique, affinité aux métaux) ou biologiques des protéines (liaison à un anticorps, un peptide, un récepteur, un ligand ou un acide nucléique). Après une série d’étapes de lavage, les protéines retenues sont recouvertes d’une matrice qui va absorber l’excès d’énergie lors de l’ionisation de l’échantillon par désorption au laser et permettre l’analyse en spectrométrie de masse en temps de vol (la spectrométrie de masse en temps de vol consiste à accélérer tous les ions à la même énergie cinétique. Leurs vitesses étant inversement proportionnelles à la racine carrée de leurs masses, les ions plus légers arrivent au détecteur plus rapidement que les ions plus lourds, ce qui permet de les séparer). Au final, on obtient un profil protéique représentant les différentes protéines en fonction du rapport masse sur charge (M/Z). Ces profils sont ensuite normalisés et calibrés puis analysés de manière différentielle par des algorithmes informatiques variés afin d’établir un profil protéique spécifique. |
| Dans le texte | |
 |
Figure 3. Dynamique d’expression des protéines sériques. L’albumine et les immunoglobulines à elles seules représentent près de 90 % des protéines totales du sérum. Le rapport entre ces protéines majoritaires et les protéines les plus faiblement représentées peut alors atteindre 106 à 109. La présence de protéines majoritaires rend la détection des marqueurs diagnostiques classiques comme le PSA (prostate specific antigen) ou l’ACE (antigène carcino-embryonnaire) très difficile malgré une bonne sensibilité de la technique SELDI-TOF. |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




