Figure 1.
Télécharger l'image originale
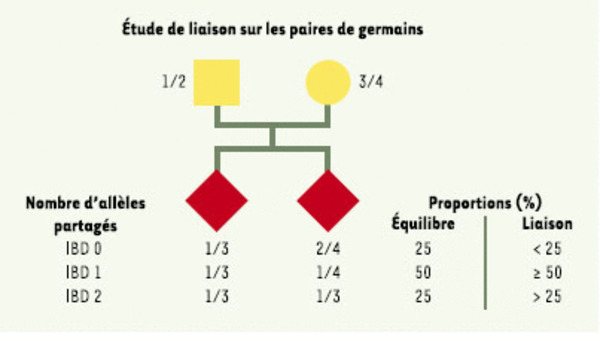
Analyse de liaison non paramétrique. Cette méthode permet d’estimer la liaison génétique entre un marqueur et le gène responsable d’un phénotype dont on ne connaît pas les paramètres de transmission, comme c’est le cas dans l’autisme. Pour ce type d’étude, il est nécessaire d’avoir accès à des familles ayant au moins deux enfants atteints. On regarde dans ces familles le pourcentage d’allèles (chaque individu possède deux allèles à chaque locus, numérotés de 1 à 4) partagés par les individus malades (IBD identity by descent). Ce type d’analyse est souvent conduit sur un nombre élevé de paires de germains atteints (affected sib-pairs). En l’absence de liaison à un locus donné, c’est-à-dire à l’équilibre, 50% des allèles seront partagés dans les fratries, avec 25 % des enfants ayant deux allèles en communs (IBD2), 50 % n’en ayant qu’un (IBD1) et 25 % ne partageant aucun allèle (IBD0). Pour chaque locus étudié, un rapport de vraisemblance d’observer de telles familles sous une hypothèse de liaison et d’indépendance génétique (LOD score) est calculé. Un excès de partage significatif indique une liaison génétique entre le marqueur étudié et l’autisme.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




