Figure 1.
Télécharger l'image originale
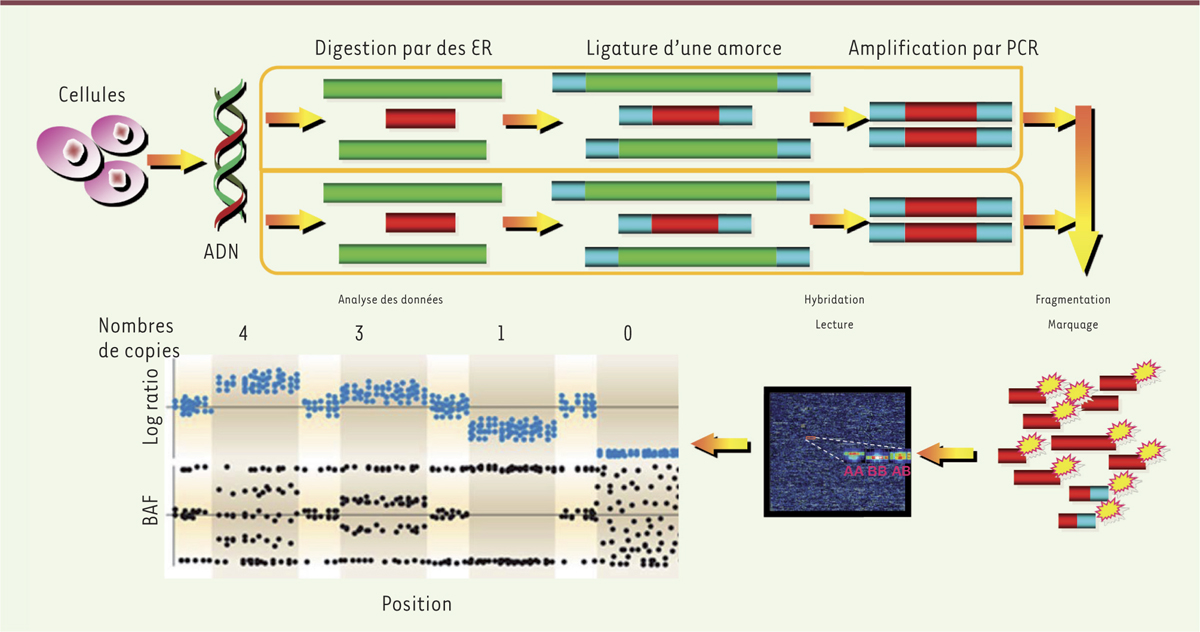
La technique de SNP-array. Cette technique permet de déterminer le variant de séquence (single nucleotide polymorphism) et le nombre de copies d’une région génétique. L’ADN d’un patient est isolé à partir des cellules sanguines ; les deux brins (allèles) sont ensuite digérés par des enzymes de restriction (ER), puis amplifiés par PCR (polymerase chain reaction) et regroupés. Des sondes marquées par des fluorochromes sont hybridées sur les puces « SNP-array » du génome humain. La quantification du signal d’hybridation de chaque sonde se fait par une lecture avec un laser ; aboutissant après numérisation à une quantification du nombre de copies pour chaque région étudiée (exprimée sous forme d’un rapport logarithmique). Cette technique offre l’avantage d’une haute résolution (identification de zones < 150 kb) et elle permet une analyse pangénomique (figure modifiée d’après Alkan et al. [10]).
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




