Figure 1.
Télécharger l'image originale
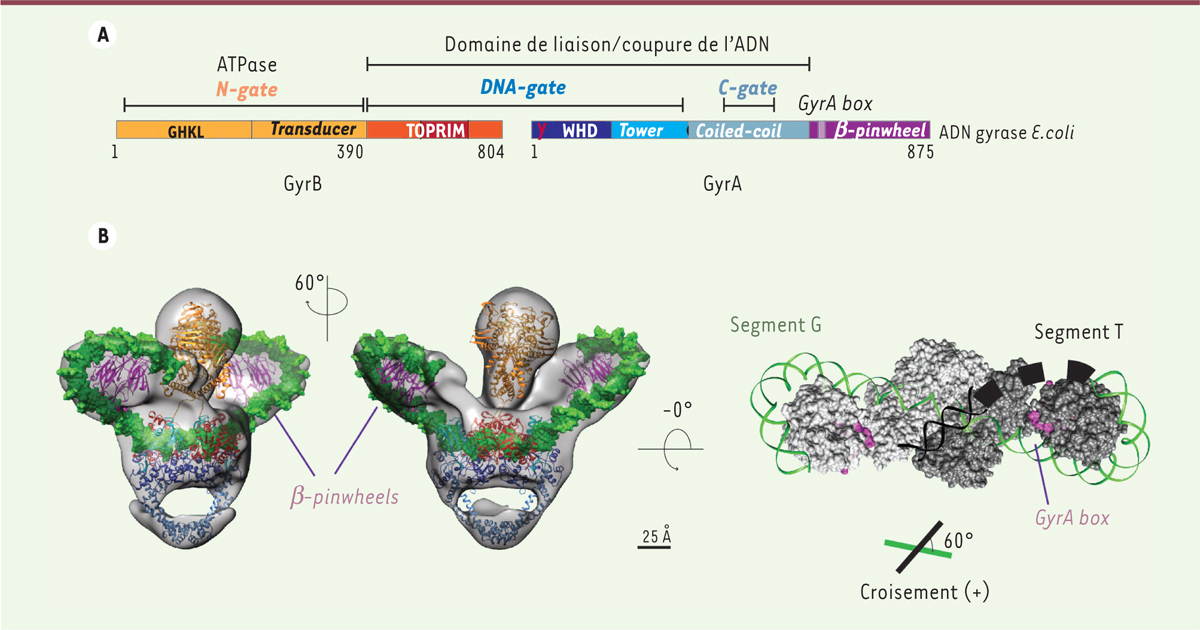
Organisation des domaines catalytiques de l’ADN gyrase (numérotation selon la référence de l’ADN gyrase de E. coli). A. Dans la sous-unité B, le domaine ATPase est formé des deux régions GHKL (site de liaison de l’ATP commun aux protéines gyrase, Hsp90, histidine kinase, MutL) et transducer formant la porte N (ou N-gate). Le domaine de liaison/clivage de l’ADN (ou DNA-gate) est composé du domaine TOPRIM de la sous-unité B, et des domaines WHD (winged helix domain portant la tyrosine conservée catalytique) et Tower de la sous-unité A. La troisième porte ou « C-gate » est constituée par deux domaines coiled-coil. Les domaines carboxy-terminaux CTD possèdent un repliement b-pinweel avec le motif conservé GyrA box. B. Reconstruction tridimensionnelle en cryo-microscopie électronique du complexe de l’ADN gyrase avec un ADN de 155 pb en complexe avec l’ADPNP et la ciprofloxacine. Deux vues de la densité électronique (surface grise) sont représentées avec les modèles moléculaires des domaines placés en densité. Les couleurs pour chaque domaine correspondent à celles du schéma ci-dessus, l’ADN est représenté en surface verte. Le schéma de droite représente une vue de dessus de la surface du complexe sans le domaine ATPase et avec une extrapolation de la trajectoire de l’ADN à l’interface de l’enzyme (segment T en noir). La GyrA box (violet) fait face au croisement positif d’ADN formé par les segments T et G. Le sens de ce croisement est inversé en surenroulement négatif après transport du segment T à travers la coupure du segment G.
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




