Figure 2.
Télécharger l'image originale
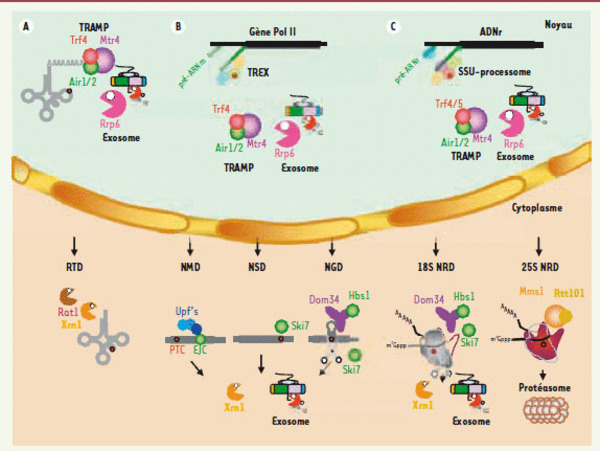
Les voies majeures de la surveillance ARN. Chacune des altérations en cis- (dans la séquence de l’ARN) ou en trans (liées à l’absence de liaison, ou à la perte, d’un facteur d’assemblage ou d’une protéine constitutive de la RNP) est représentée par un point rouge. A. ARNt noyau : les précurseurs d’ARNt hypomodifiés sont polyadénylés à leur extrémité 3’ par le complexe TRAMP4. Le complexe TRAMP et la queue de poly(A) agissent comme un signal de recrutement et de stimulation de l’exosome qui va mener à une digestion exoribonucléolytique 3’->5’ du substrat.Cytoplasme : les ARNt hypomodifiés sont dégradés par la voie du Rapid tRNA decay ou RTD qui implique leur digestion 5’->3’ par les exoRNases Xrn1 et Rat1. B. ARNm noyau : l’assemblage cotranscriptionnel d’une mRNP est symbolisé (gène en noir ; transcrit naissant en vert). Les défauts d’assemblage entraînent soit la séquestration des pré-mRNP dans des foci spatialement liés aux sites de transcription, soit la polyadénylation par TRAMP4 de leurs constituants ARN et leur dégradation rapide par l’exosome. TREX : transport-export.Cytoplasme : un ARNm est schématisé avec soit un stop prématuré (annoté PTC) dans la phase ouverte de lecture (cas du NMD), soit pas de stop (cas du NSD), soit une tige-boucle, symbolisant un ralentissement du ribosome dans sa progression (cas du NGD). Lors du NMD, un complexe de surveillance constitué des protéines Upf initie la dégradation menée aussi bien par Xrn1, suite au décoiffage (mais ne nécessitant pas de déadénylation préalable), que par l’exosome, suite à une déadénylation accélérée. Chez les mammifères, les protéines Upf sont assistées du complexe EJC pour exon junction complex, une signature de l’épissage, qui servirait de plateforme d’assemblage aux protéines Upf’s. C’est en partie la distance entre le PTC et l’EJC (mammifères) ou entre le PTC et la queue de poly(A) liée par la protéine Pab1 (levure), qui jouerait un rôle déterminant dans la reconnaissance du codon stop prématuré. Notons que chez les insectes, le NMD nécessite un clivage endonucléolytique initiateur, ce qui le rapproche d’un point de vue du mécanisme du NGD (voir ci-dessous). Dans le NSD, c’est Ski7, une protéine homologue respectivement aux facteurs d’élongation et de terminaison de la traduction eEF1A et eRF3 qui, agissant comme un véritable mime moléculaire, reconnaîtrait le ribosome bloqué sur l’ARNm (dont le site A est vide) et recruterait l’exosome. Il n’y a pas de déadénylation préliminaire dans le NSD. Xrn1 et la dégradation 3’->5’ contribuent au NSD. Chez les procaryotes, les polypeptides synthétisés à partir d’ARNm dépourvus de codon stop sont étiquetés pour la dégradation par l’ajout d’une séquence lue sur un ARNtm [47]. Dans le NGD, les protéines partenaires Dom34 et Hbs1 sont recrutées sur un messager induisant un ralentissement dans la progression du ribosome, ce qui enclenche au moins un clivage endonucléolytique par une protéine qui demeure inconnue [48] ; les fragments générés sont dégradés par Xrn1 et l’exosome. Dom34 et Hbs1 sont homologues respectivement aux facteurs de terminaison de la traduction eRF1 et eRF3 et, dans ce cas, le modèle de reconnaissance du site A vide du ribosome serait également basé sur le principe du mime moléculaire. C. ARNr noyau : les défauts d’assemblage du préribosome entraînent la polyadénylation de leurs ARN par les complexes TRAMP5 (ARN destiné à la petite sous-unité) ou TRAMP4 (grande sous-unité) suivie de leur dégradation par l’exosome. Le SSU-processome qui est un complexe macromoléculaire indispensable à la formation de la petite sous-unité du ribosome est représenté. Il résulte de l’assemblage hiérarchisé de plusieurs modules protéiques autonomes organisés autour de l’ARNsno U3 qui, lui, interagit par appariement Watson-Crick avec l’extrémité 5’ de l’ARN naissant. Cytoplasme : les petites sous-unités défectueuses du ribosome (suite à l’introduction par l’expérimentateur d’altérations en cis- dans des positions ultra-conservées du site de décodage) sont dégradées par le mécanisme de 18S NRD, qui rappelle à beaucoup d’égards le mécanisme de NGD. Les grandes sous-unités non fonctionnelles (résultant de l’introduction du même type de mutation dans le centre peptidyl transférase) sont également reconnues et dégradées. Le processus dénommé 25S NRD est toutefois distinct d’un point de vue du mécanisme mis en jeu. Dans ce cas, des composants protéiques associés avec le ribosome, dont la nature moléculaire reste à préciser, sont ubiquitinylés par le complexe Mms1-Rtt101 et les ribosomes modifiés dirigés pour la dégradation vers le protéasome.
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




