Figure 3.
Télécharger l'image originale
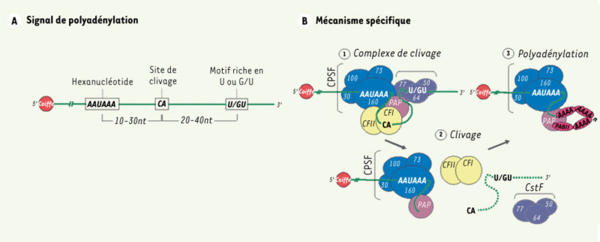
Polyadénylation. A. Signal de polyadénylation. La formation de la queue de polyadénosines est dirigée par le signal de polyadénylation composé de trois séquences. On retrouve ainsi un hexanucléotide hautement conservé, AAUAAA, situé à 10-30 bases du site de clivage, un motif riche en U ou en G/U (downstream element : DSE) positionné 20-40 bases en aval du site de clivage et le site de clivage, lui-même constitué du dinucléotide CA. La position exacte de ce dernier site est déterminée par la distance existant entre l’hexanucléotide et la séquence riche en U ou en G/U. L’étendue de la distance est également une indication de la longueur que peut atteindre la queue poly(A) pour chaque ARNm. Celle-ci varie normalement de 200 à 300 résidus adénosines. B. Mécanisme spécifique. 1. Afin de permettre une polyadénylation complète, un minimum de six facteurs polymériques doivent être recrutés dans le noyau. On retrouve tout d’abord le facteur protéique CPSF (cleavage/polyadenylation specific factor) qui participe à la fois à la réaction de clivage et à celle de polyadénylation. Il est constitué de quatre sous-unités nommées selon leur poids moléculaire respectif : CPSF-160, CPSF-100, CPSF-73 et CPSF-30. Le facteur CstF, constitué des sous-unités CstF-77, CstF-64 et CstF-50, est pour sa part engagé dans l’activité de clivage sans que sa présence ne soit obligatoire lors de la polyadénylation. Les facteurs CFI et CFII (cleavage factors I and II) sont essentiels pour le processus de clivage puisqu’ils possèdent chacun une activité endonucléolitique. L’extrémité carboxyterminale (CTD) de l’ARN polymérase II (Pol II) fait également partie de la machinerie de polyadénylation. L’enzyme responsable de la synthèse de la queue poly(A), la poly(A) polymérase (PAP), est généralement requise pour obtenir un complexe de clivage actif. Notons que son activité de polymérisation est strictement exclusive à l’incorporation d’adénosines sur n’importe quel substrat d’ARN. Cependant, après le clivage du substrat, l’élongation de la queue poly(A) peut avoir lieu mais seulement en présence du complexe protéique CPSF. Pour augmenter la vitesse d’élongation tout en régulant la longueur de la queue poly(A), la protéine de liaison à la queue poly(A), PAB II (poly(A)-binding protein II), interagit avec ce segment d’adénosines. 2. Le processus de maturation nucléaire de l’extrémité 3’ de l’ARN pré-m débute par la formation du complexe de clivage. Pour ce faire, il y a liaison de CPSF et CstF à l’hexanucléotide et à la séquence riche en U ou en G/U. CFI et II sont ensuite recrutés au site de clivage. Le complexe est complété par la triple association de PAP à CPSF, CstF et CFI. La coupure de l’ARN entraîne la dégradation éventuelle du produit de clivage en 3’ par des exonucléases cellulaires de même que la libération des facteurs CstF, CFI et CFII. L’activité de polyadénylation de PAP débute alors très lentement à l’extrémité 3’ de l’ARN pré-m jusqu’à l’arrivée de PAB II. Ainsi, à chaque groupe de 10 résidus adénosines nouvellement synthétisés, PAB II s’y apparie, causant l’accélération de la processivité de la polymérase. Une fois la longueur maximale de la queue poly(A) atteinte, l’activité de PAP ralentit et s’arrête complètement avec la dissociation du complexe PAP-CPSF.
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




