| Numéro |
Med Sci (Paris)
Volume 41, Numéro 6-7, Juin-Juillet 2025
Chroniques génomiques
|
|
|---|---|---|
| Page(s) | 611 - 614 | |
| Section | Forum | |
| DOI | https://doi.org/10.1051/medsci/2025097 | |
| Publié en ligne | 7 juillet 2025 | |
Séquençage de l’ADN
La fin d’un quasi-monopole
DNA sequencing: the end of a near-monopoly
Biologiste, généticien et immunologiste, Président d’Aprogène (Association pour la promotion de la Génomique), Marseille, France
Abstract
For the last decade, the field of DNA sequencing has been dominated by a single company, Illumina, with very limited competition. However, a new method with superior performance and lower cost has recently been announced by Roche, a major pharmaceutical and diagnostics company, and promises to have a large impact, especially (but not exclusively) for medical applications.
© 2025 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l’utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l’utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.

Vignette (© Bertrand Jordan).
Un acteur dominant dans un paysage complexe
Le séquençage « en masse » de l’ADN est devenu rapide et financièrement accessible, avec l’apparition de techniques et d’appareils globalement désignés par le sigle NGS (nextgeneration sequencing) à partir de 2005, quelques années après l’obtention de la première séquence nucléotidique du génome humain, qui avait mobilisé de très nombreux laboratoires et coûté plus d’un milliard de dollars. Après des débuts qui ont vu s’affronter nombre de techniques proposées par des firmes aujourd’hui disparues (454, Solexa, SOLiD, Helicos, Complete Genomics), le marché s’est stabilisé autour d’un acteur ultra-dominant, l’entreprise Illumina, dont les machines peuvent actuellement lire une centaine de génomes humains par semaine1 à un coût inférieur à mille dollars par génome. Il s’agit d’une entreprise importante : Illumina emploie près de neuf mille personnes, et son chiffre d’affaires dépasse neuf milliards de dollars ; on estime que 90 % des séquences d’ADN lues chaque année dans le monde proviennent de ses machines. Quelques autres firmes subsistent sans vraiment menacer le market leader : Pacific Biosciences, qui permet d’obtenir des séquences plus longues et de très bonne qualité, mais à un coût cinq ou dix fois plus élevé, Oxford Nanopore, exploitant comme son nom l’indique le passage de l’ADN à travers des nanopores (systèmes très compacts et bon marché, lectures très longues, mais débit limité et taux d’erreurs plus élevé), et enfin Ion Torrent, avec un système aux capacités modestes, mais qui a su se faire une place pour des applications de diagnostic. Il s’agit là d’entreprises bien plus petites qu’Illumina, financièrement fragiles, et dont l’avenir n’est pas assuré. Mentionnons enfin des start-up plus récentes et prometteuses : Ultima Genomics qui revendique un génome à 80 dollars, Element Avita, Singular Genomics, qui ont chacune produit et placé quelques machines, mais qui font figure d’outsider, et une firme chinoise, MGI (aussi appelée BGI, et qui a repris, pour les États-Unis, le nom de Complete Genomics, dont elle exploite la technologie) – mais cette dernière, bien implantée en Extrême-Orient, a pour des raisons politiques peu de chances de percer à l’Ouest. Une nouvelle arrivée va sans doute bouleverser ce paysage : la multinationale Roche a récemment annoncé le lancement d’un système de séquençage « ultra-performant » susceptible de remettre en question le leadership d’Illumina.
Roche et le séquençage nucléotidique : vingt ans de réflexion…
Roche, première entreprise pharmaceutique mondiale et qui emploie plus de cent mille personnes, s’intéresse depuis longtemps au séquençage de l’ADN et à ses applications pour le diagnostic. En 2007, elle avait racheté 454, le premier acteur du NGS. C’est grâce à cette technique que furent réalisées les premières séquences de l’ADN de Néandertal [1] ainsi que la lecture du génome de Jim Watson [2], qui avait coûté environ un million de dollars – mille fois moins que celle du Programme Génome, mais mille fois plus qu’aujourd’hui. La compétition des systèmes Illumina (plus productif) et Ion Torrent (mieux adapté aux applications cliniques) devait amener Roche à abandonner la commercialisation du système 454 en 2013. La firme continuait néanmoins à s’intéresser au séquençage de l’ADN, avec un intérêt particulier pour l’emploi de nanopores (objet durant quelques années d’une collaboration avec IBM) et l’acquisition d’entreprises dans cet objectif. Deux d’entre elles, Stratos (acquise en 2020) et Genia (acquise en 2014) ont fourni la base du nouveau système annoncé par Roche en février 2025.
Une combinaison efficace
Le système de séquençage annoncé par Roche n’a pas encore de nom commercial (il devrait être disponible en 2026) et s’appelle pour le moment SBX (pour sequencing by expansion). On en trouve une description détaillée dans un blog très bien informé2 ainsi que sur le site internet de la firme3. La méthode associe deux techniques mises au point par des entreprises acquises par Roche, qui ont ensuite été perfectionnées et combinées. La première est celle des « expandomères », développée par Stratos, qui repose sur une copie de l’ADN aboutissant à une molécule dans laquelle les bases sont largement espacées (Figure 1).
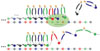 |
Figure 1 Principe des « expandomères ». En haut, copie de l’ADN employant des nucléotides qui comportent une grande séquence en boucle ; en bas, ouverture des boucles (par un traitement à l’acide, astérisque jaune) et obtention d’un expandomère dans lequel les bases sont largement espacées (image extraite de la documentation SBX Roche3). Naturellement, on perd au passage le repérage des bases modifiées – du moins dans la version actuelle de la méthode. |
L’expandomère sera ensuite séquencé par un système de nanopores dont les performances sont largement améliorées par la séparation ainsi introduite entre les bases (voir plus loin). La difficulté technique est de disposer d’une polymérase capable d’incorporer ces nucléotides hautement anormaux et bien plus volumineux qu’un nucléotide normal : une vingtaine de kilodaltons au lieu d’environ 500 daltons. Il a fallu en quelque sorte « construire » cette polymérase à partir d’un enzyme existant, ce qui a requis un très gros travail de mutagénèse dirigée faisant appel aux techniques les plus sophistiquées d’ingénierie des protéines. Le résultat est une polymérase pouvant copier un brin d’ADN en une suite d’expandomères avec un taux d’erreur inférieur à 1 % – et capable de synthétiser des séquences dont la longueur dépasse 1 000 nucléotides. Une performance remarquable, qui va grandement faciliter la lecture ultérieure de la séquence. Cette dernière est confiée à un système développé par Genia et qui repose sur des nanopores formés sur une puce CMOS (complementary metal oxide semi-conductor). Cette dernière comporte huit millions d’éléments, chacun associé à un nanopore, et l’ADN (sous forme d’expandomère) est tiré à travers le nanopore par de brèves impulsions électriques (Figure 2).
 |
Figure 2 Passage d’un expandomère dans un nanopore et lecture de la séquence. L’élément figuré par le triangle jaune assure le déplacement de l’ADN à travers le pore en réponse à une brève impulsion électrique (image extraite de la documentation SBX Roche3). |
Pour percevoir tout ce qu’apporte l’utilisation des expandomères, il faut comprendre la difficulté du séquençage par nanopores tel qu’il est habituellement pratiqué. On imagine souvent que chaque base est lue au passage dans le nanopore et reconnue grâce à son signal caractéristique, mais la réalité est plus compliquée. La dimension du nanopore est très supérieure à celle d’une base, et le signal provient de l’ensemble des quatre ou cinq bases présentes à l’intérieur du pore à un moment donné : il faut donc interpréter très finement ce signal pour en déduire la séquence, ce qui explique à la fois le taux d’erreurs relativement élevé et la nécessité d’une informatique performante. Ce problème n’existe plus avec les expandomères puisque les bases sont très espacées, passent une par une dans le nanopore, et de fait, les données brutes issues de ce système sont pratiquement lisibles à l’œil nu. Le fait que l’expandomère soit obtenu par synthèse d’un nouvel ADN permet aussi d’inclure une bonne différenciation entre les bases. Le résultat est un système capable, selon les données dévoilées par Roche lors de sa présentation, de lire sept génomes humains en une heure. Il faut certes ajouter la durée de la préparation des expandomères (deux heures), mais il s’agit tout de même de performances exceptionnelles. Et le coût de l’appareil, qui contrairement à la plupart de ses concurrents ne comporte pas de système optique ultra-sophistiqué, devrait être très compétitif (il n’est pas encore connu). Quant au coût de fonctionnement, le fait que la puce CMOS soit réutilisable devrait contribuer à le limiter, et Roche vise le génome à cent dollars sans pour autant avoir annoncé des tarifs précis.
Un changement de paysage
Le système SBX, dont je viens d’indiquer les grandes lignes, est donc très prometteur, et semble susceptible d’accélérer considérablement le séquençage d’ADN tout en diminuant son coût. Et il ne s’agit pas d’une énième technologie révolutionnaire annoncée par une petite start-up aux reins fragiles et à la durée de vie incertaine : la firme Roche est une entité puissante capable d’investir sur le long terme. Elle vise à coup sûr le marché des applications cliniques, prévoyant un avenir dans lequel le séquençage du génome d’un patient fera partie des examens de routine au même titre qu’un scanner ou une IRM (imagerie par résonance magnétique) – et pour un coût du même ordre. Illumina ou Oxford nanopore se tournent aussi vers ce marché – mais l’entreprise Roche, largement implantée dans le secteur du diagnostic et connaissant bien ses impératifs, bénéficie d’un avantage décisif. À moins d’un bug majeur (et peu probable), on peut donc prévoir que Roche fera bientôt partie des « Grands » du séquençage d’ADN, avec un fort impact médical, mais aussi pour les applications en recherche (voir le blog déjà mentionné pour un état des lieux complet)4. En tout cas, que de chemin parcouru depuis les premières autoradiographies de Fred Sanger… [3].
Liens d’intérêt
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Robinson K. Roche Xpounds on new sequencing technology. Omics ! Omics ! Feb. 20, 2025 https://omicsomics.blogspot.com/2025/02/roche-xpounds-on-new-sequencing.html
Robinson K. Roche Ripple Predictions. Omics ! Omics ! Feb.20, 2025 https://omicsomics.blogspot.com/2025/02/roche-ripple-predictions.html
Références
- Green RE, Krause J, Ptak SE, et al. Analysis of one million base pairs of Neanderthal DNA. Nature. 2006 ; 444 : 330–6 [CrossRef] [PubMed] [Google Scholar]
- Wheeler DA, Srinivasan M, Egholm M, et al. The complete genome of an individual by massively parallel DNA sequencing. Nature. 2008 ; 452 : 872–6. [CrossRef] [PubMed] [Google Scholar]
- Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci USA 1977 ; 74 : 5463–7. [CrossRef] [Google Scholar]
Liste des figures
 |
Figure 1 Principe des « expandomères ». En haut, copie de l’ADN employant des nucléotides qui comportent une grande séquence en boucle ; en bas, ouverture des boucles (par un traitement à l’acide, astérisque jaune) et obtention d’un expandomère dans lequel les bases sont largement espacées (image extraite de la documentation SBX Roche3). Naturellement, on perd au passage le repérage des bases modifiées – du moins dans la version actuelle de la méthode. |
| Dans le texte | |
 |
Figure 2 Passage d’un expandomère dans un nanopore et lecture de la séquence. L’élément figuré par le triangle jaune assure le déplacement de l’ADN à travers le pore en réponse à une brève impulsion électrique (image extraite de la documentation SBX Roche3). |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




