Figure 2.
Télécharger l'image originale
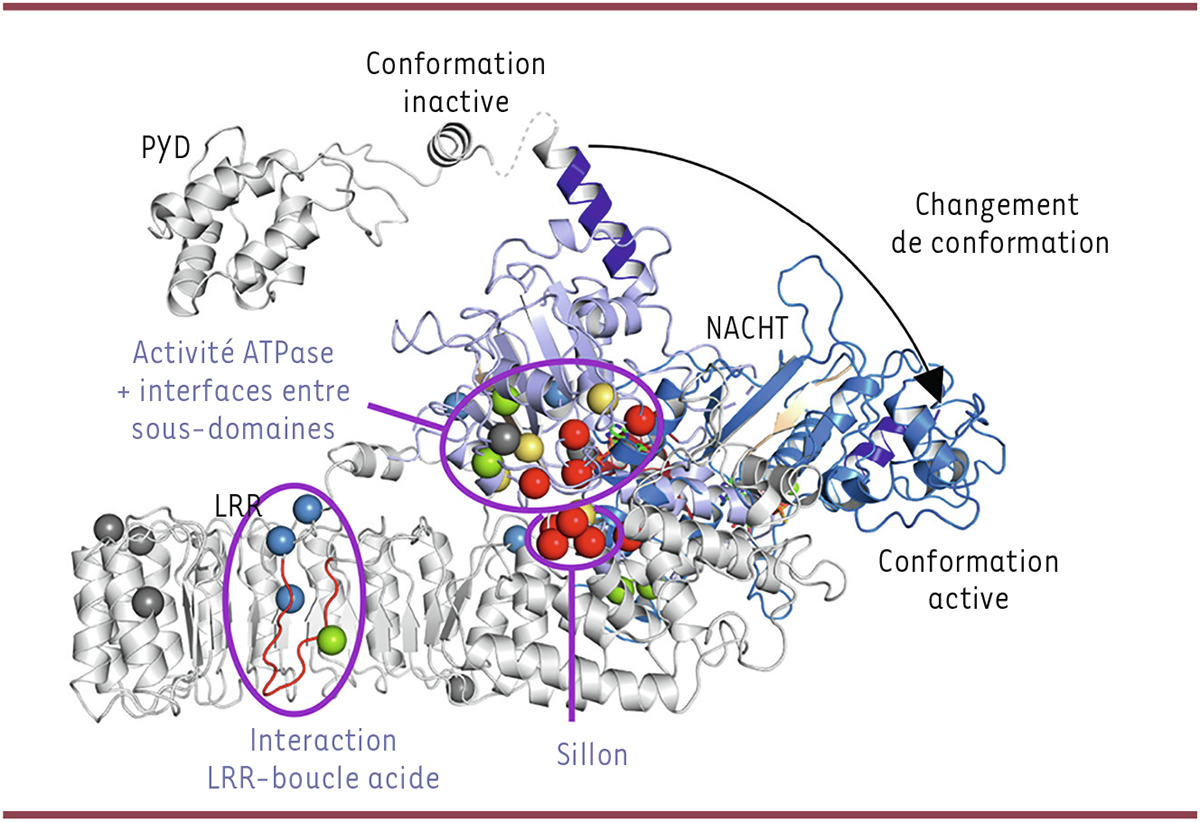
Identification des structures impliquées dans l’activation de NLRP3 par l’analyse de ses variants naturels. Les mutations gain-de-fonction de NLRP3 sont concentrées au site ATPase de la protéine, aux interfaces entre sous-domaines impliqués dans le changement conformationnel responsable de son activation, dans un sillon accessible à la surface de la protéine, sans fonction connue, et à l’interface entre le domaine leucine-rich repeats (LRR) et une boucle acide (en rouge). La mise en évidence de ces structures clés pour le contrôle de l’activation de NLRP3 permettra d’orienter les recherches mécanistiques et de les cibler pharmacologiquement. Les sphères colorées indiquent les mutations gain-de-fonction de NLRP3 responsables d’une activité constitutive de la protéine (en rouge), d’une activité dépendante indifféremment de l’amorçage ou du signal d’activation (en vert), d’une activité dépendante strictement de l’amorçage (en jaune) ou strictement du signal d’activation (en bleu), ainsi que les variants sans gain de fonction (en gris). PYD : domaine pyrine ; NACHT : domaine nommé d’après les autres protéines qui le possèdent (NAIP, CIITA, HET-E et TEP1).
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




