Figure 1.
Télécharger l'image originale
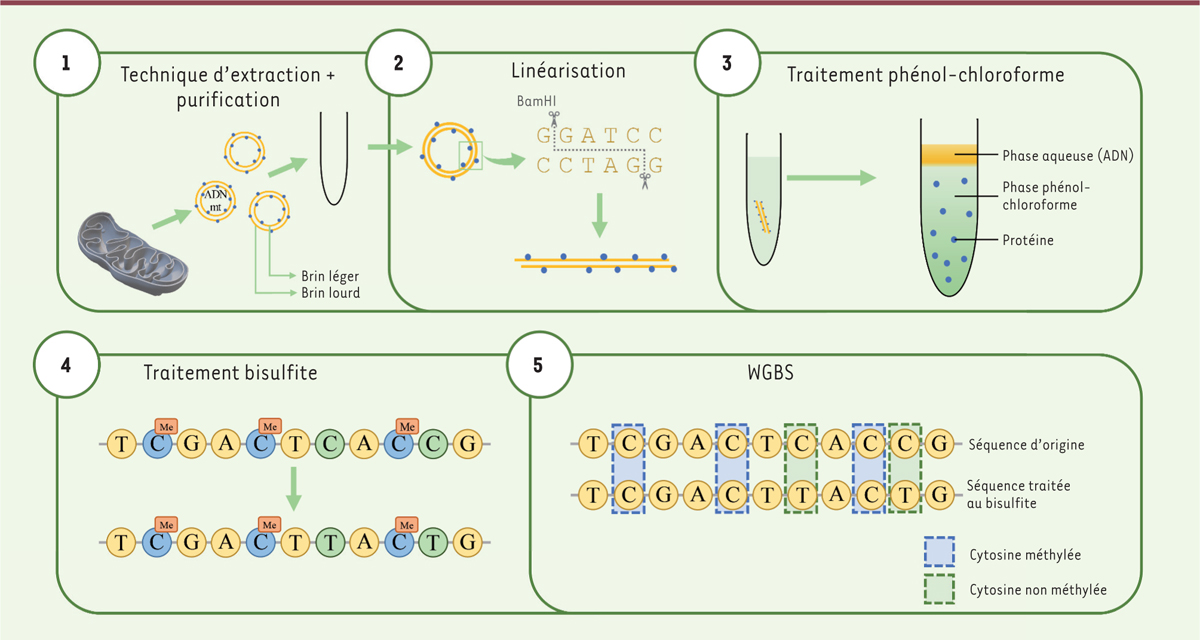
Démarche optimale pour détecter la méthylation des cytosines dans l’ADNmt. 1. Séparation de l’ADNmt et de l’ADNn pour éviter les contaminations (en isolant d’abord les mitochondries, par exemple) et extraction de l’ADNmt. 2. Linéarisation de l’ADNmt par des enzymes de restriction (par exemple BamHI) pour que le bisulfite de sodium ait accès à toutes les cytosines. 3. Traitement au phénol-chloroforme pour enlever toutes les protéines liées à l’ADNmt. 4. Traitement au bisulfite de sodium pour la conversion des cytosines non méthylées. 5. WGBS afin d’établir le patron et le taux de méthylation de l’ADNmt sans discrimination entre les cytosines dans un contexte CpG et non CpG.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




