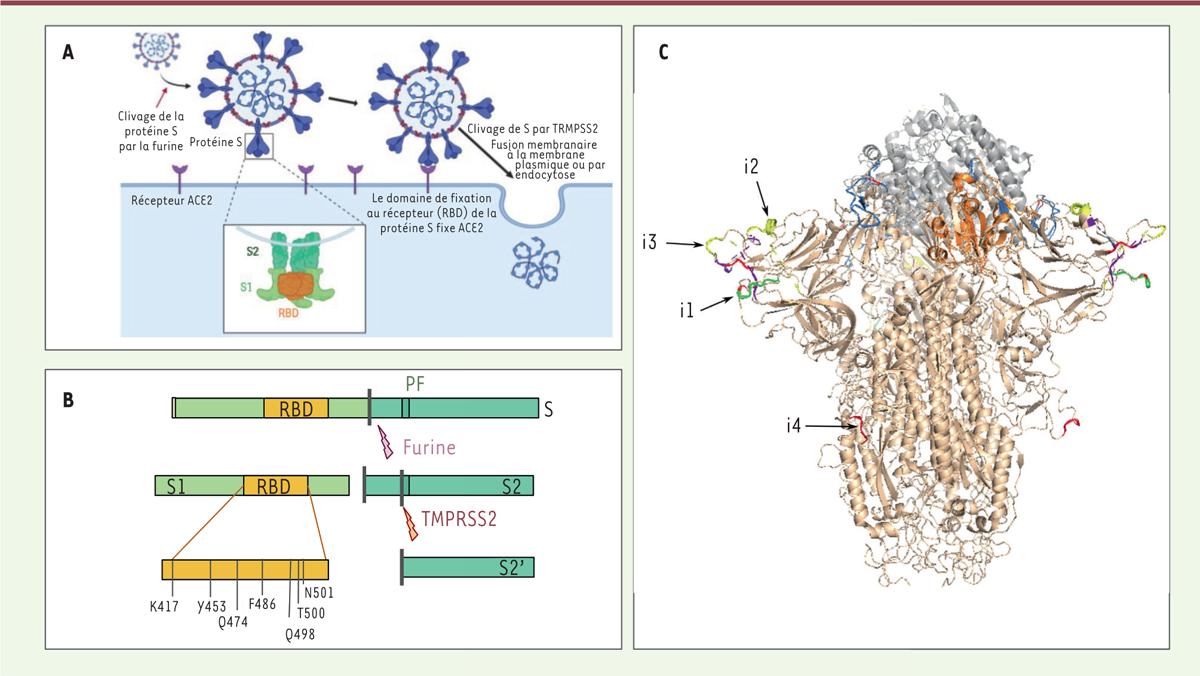
Figure 3.
Télécharger l'image originale
Structure et fonctions de la protéine S (spicule, spike en anglais). A. Représentation schématique de l’infection des cellules par le SARS-CoV-2 après fixation de la protéine S au récepteur ACE2. B. La protéine S subit deux étapes de maturation par clivage protéolytique (par les protéases furine puis TMPRSS2) nécessaires à son activation et à la libération du peptide de fusion. C.. Structure de la protéine S fixée au récepteur ACE2. La structure de la protéine S de SARS-CoV-2 (en beige) est obtenue grâce au logiciel SWISSMODEL sur la base de la structure 6acc de SARS-CoV (disponible dans Protein Data Bank [PDB]), et alignée sur la structure d’un domaine RBD (en orange) interagissant avec ACE2 (en gris) issue du modèle 6m0j (disponible dans PDB). Les sites d’insertion sont indiqués en couleur. Les résidus sont colorés en fonction de l’ordre de conservation des insertions, en passant du rouge (insertion présente uniquement chez SARS-CoV-2), au jaune, vert, bleu clair puis indigo (insertion présente chez la majorité des sarbécovirus [sous-genre de coronavirus regroupant les virus apparentés à celui du SRAS]).
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




