Figure 2.
Télécharger l'image originale
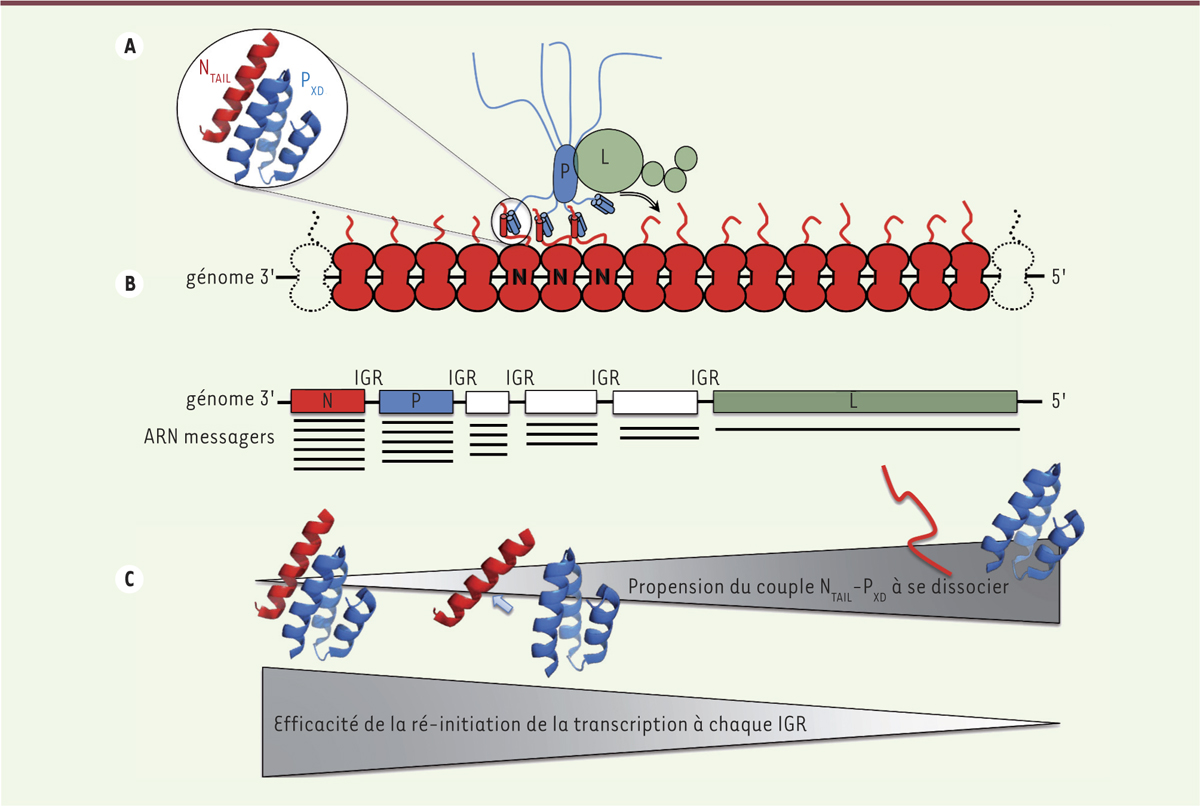
Illustration schématique de l’activité de transcription de la polymérase virale. A. représentation schématique du déplacement de la polymérase virale (L, en vert) en complexe avec la phosphoprotéine (P, en bleu) le long de la nucléocapside, faite du génome viral encapsidé par la nucléoprotéine N (en rouge). La protéine P est représentée sous forme de tétramère, avec sa région centrale responsable de l’oligomérisation montrée en ovale allongé. Les lignes ondulées représentent la région N-terminale de P ainsi que la région flexible reliant la partie centrale de P à son domaine X (PXD). Les hélices au sein de PXD et de NTAIL sont représentées sous forme de cylindre. La liaison du complexe L-P à la nucléocapside repose sur l’interaction entre le domaine X de P (PXD) et le domaine NTAIL de N. B. organisation schématique du génome du virus de la rougeole avec ses six gènes, séparés par des régions inter-géniques (IGR). En dessous de chaque gène sont représentés, dans leur abondance relative, les ARN messagers respectivement transcrits. C. l’efficacité avec laquelle la polymérase virale ré-initie la transcription à chaque région intergénique (IGR) dépend de l’affinité du couple NTAIL -PXD. Une propension plus prononcée du complexe NTAIL -PXD à la dissociation (de la gauche vers la droite) est ainsi corrélée à une plus faible efficacité de ré-initiation de la transcription.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




