Figure 2.
Télécharger l'image originale
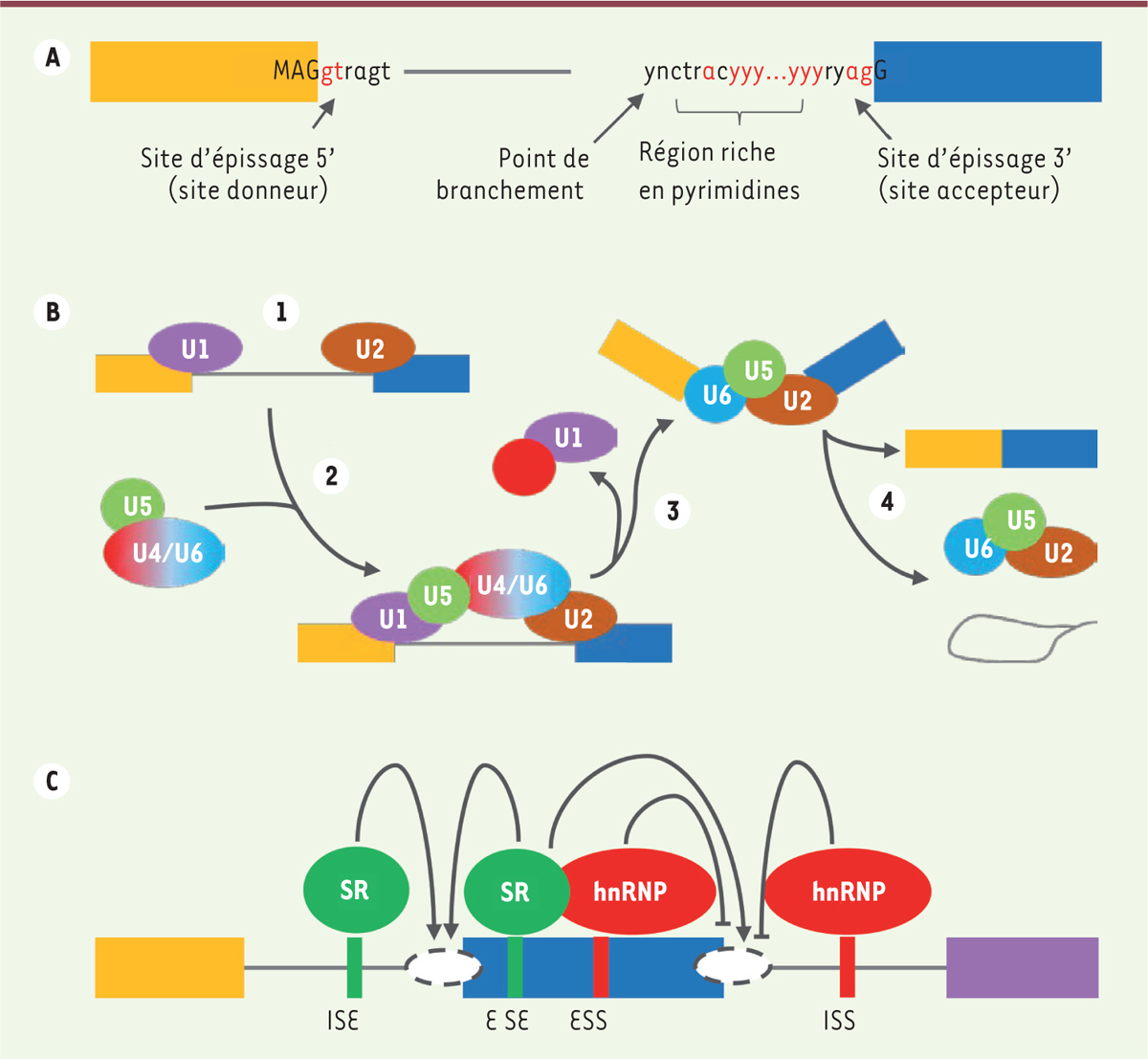
L’épissage. A. Signaux de l’épissage reconnus par les complexes ribonucloprotéiques du splicéosome. Le code IUPAC (International Union of Pure and Applied Chemistry) est utilisé pour décrire la séquence nucléotidiques (M : A ou C ; N : A, C, G ou T ; R : A ou G ; Y : C ou T). B. La réaction d’épissage. (1) Le complexe U1 s’associe au site donneur et U2 se fixe sur le point de branchement. (2) Les complexes U4/U5/U6 interviennent : le complexe U6 se fixe sur le complexe U2 tandis que le complexe U5 va se fixer sur l’extrémité 3’ de l’exon (proche du site donneur d’épissage) à proximité du complexe U1. (3) Les complexes U1 et U4 sont libérés, le complexe U5 glisse sur l’intron et le complexe U6 se fixe sur le site donneur qui est coupé. (4) L’extrémité 3’ de l’intron est coupée, ce qui libère l’intron (forme d’un « lasso ») et les exons sont liés. Enfin, le complexe se dissocie. C. La modulation de la reconnaissance des sites d’épissage par les protéines régulatrices de l’épissage. Il est souvent admis que les protéines SR (serine/arginine-rich) se fixent sur les séquences activatrices ESE (exonic splicing enhancer) ou ISE (intronic splicing enhancer). À l’inverse, les hnRNP (heterogeneous nuclear ribonucleoprotein) sont généralement connues pour se fixer sur les séquences inhibitrices ESS (exonic splicing silencer) ou ISS (intronic splicing silencer).
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




