Figure 1.
Télécharger l'image originale
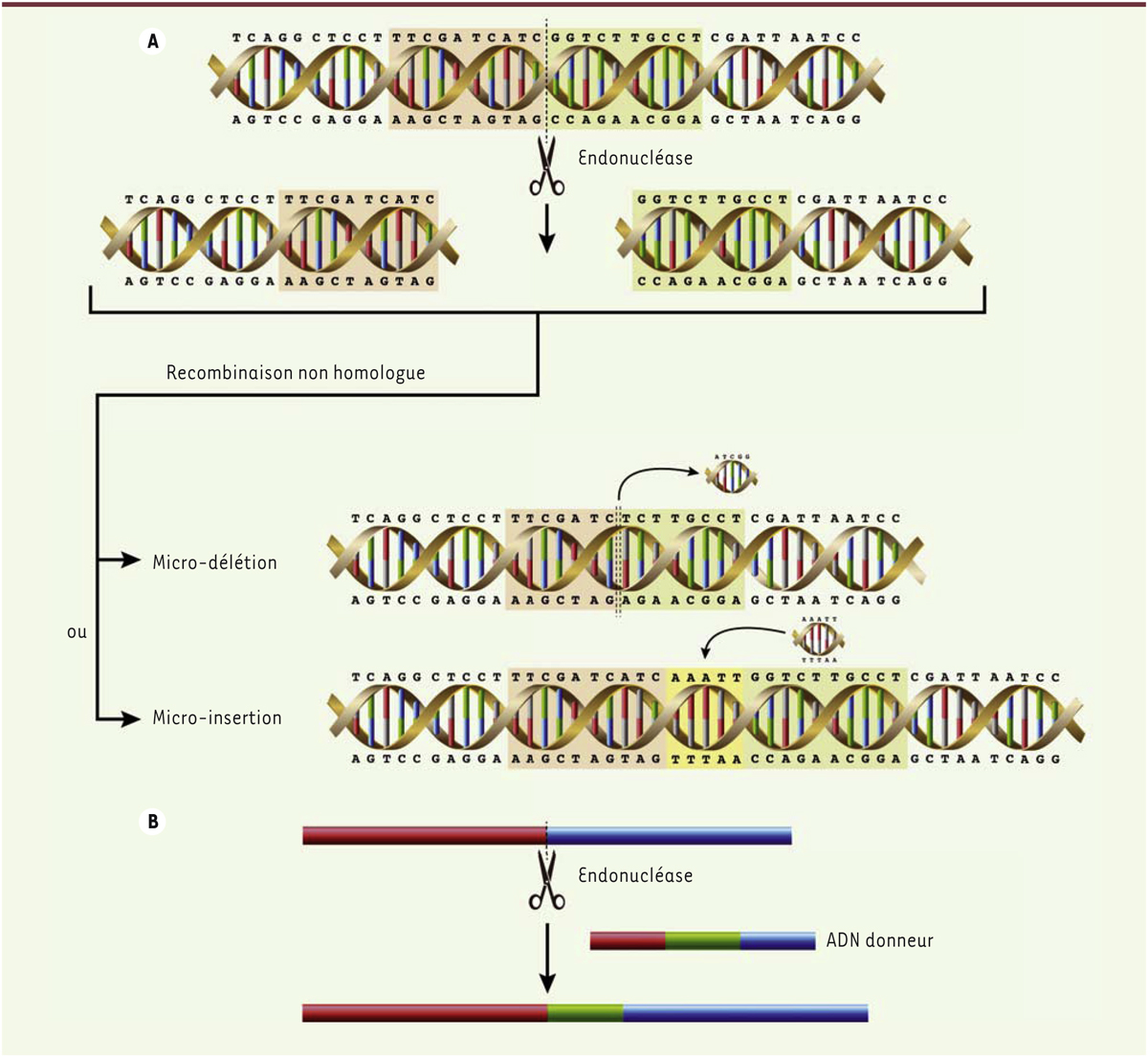
Principes de l’édition du génome à l’aide de nucléases. Plusieurs sortes d’endonucléases (méganucléases, nucléases à doigts de zinc, nucléases TAL effecteur et la combinaison d’un ARN guide et d’une Cas9) peuvent être conçues et produites par des méthodes de biologie moléculaire pour cibler une séquence choisie d’ADN afin d’y induire des coupures touchant les deux brins. Ces coupures peuvent être réparées soit par recombinaison non-homologue (A) , soit par recombinaison homologue (B) . Pour la recombinaison homologue, la présence d’une séquence d’ADN donneur est nécessaire. Cet ADN donneur doit contenir des séquences de nucléotides homologues (en rouge et bleu) à celles qui précèdent et suivent le site de coupure. Dans le schéma ces séquences sont courtes mais elles peuvent contenir jusqu’à 500 nucléotides. Entre les deux séquences homologues, l’ADN donneur peut contenir une séquence de nucléotides (en vert) plus ou moins longue qui permet de modifier quelques nucléotides du gène ciblé, d’y introduire un ou plusieurs exons manquants, ou même d’introduire un gène complet. La recombinaison non-homologue induit une réparation moins précise qui peut résulter soit en une micro-délétion, soit en une micro-insertion (en jaune) d’un nombre variable de nucléotides. Ces micro-insertions ou micro-délétions, si elles ne sont pas des multiples de trois nucléotides, changent le cadre de lecture du gène résultant soit en un gène non fonctionnel soit dans le rétablissement d’un gène fonctionnel lorsque le gène ciblé ne l’était pas.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




