Figure 5.
Télécharger l'image originale
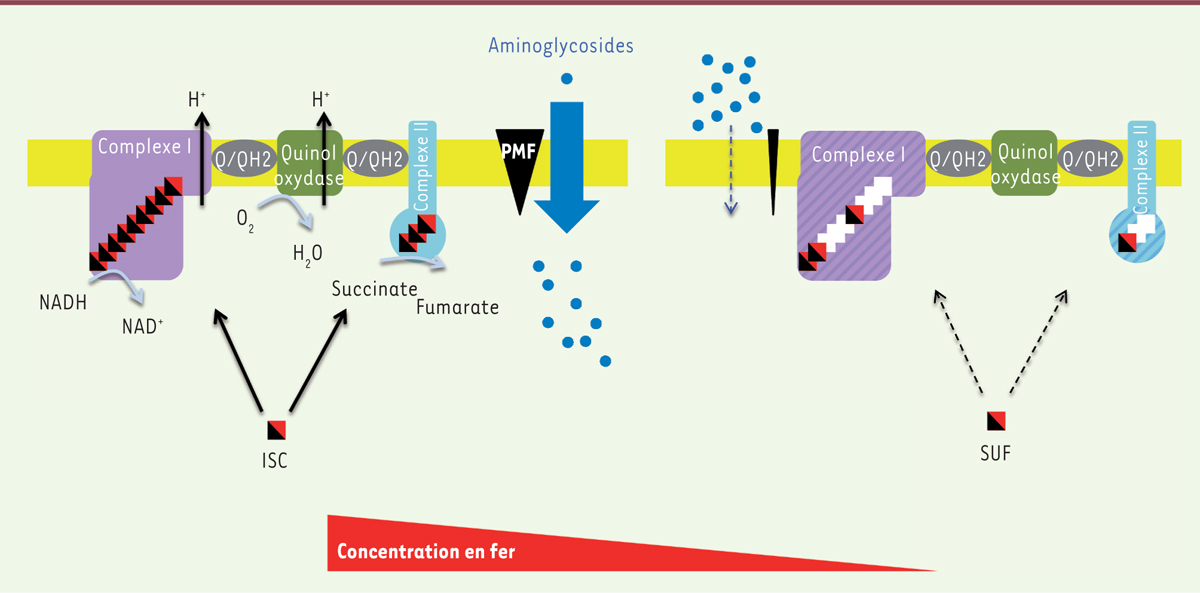
Modèle illustrant comment la biogenèse des centres Fe-S peut moduler la sensibilité d’E. coli aux aminoglycosides. Les aminoglycosides (points bleus) pénètrent dans la cellule (flèche bleue) par un mécanisme dépendant de la force proton motrice (PMF) (triangle noir). Cette dernière est générée par l’activité des complexes respiratoires (violet et bleu clair) connus pour dépendre de protéines à centres Fe-S (carré noir et rouge). En conditions limitantes en fer, E. coli utilise la voie de biogenèse SUF (droite) qui induit une maturation moins efficace des complexes I et II (centres Fe-S manquants représentés par un carré blanc) et, de surcroît, la synthèse des complexes I et II est réduite (schématisée par des hachures). La synthèse de la machinerie ISC, et celle des complexes I et II, sont sous le contrôle inhibiteur de l’ARNnc RyhB qui est lui-même sous le contrôle répresseur de Fur. En conditions limitantes en fer, la répression exercée par Fur est levée, l’ARNnc RyhB est exprimé, la synthèse de la machinerie ISC inhibée, le régulateur IscR sous sa forme apo qui active l’expression de l’opéron suf, dont la répression par Fur est également levée. Il en résulte une souche synthétisant ses centres Fe-S par la machinerie SUF. Une telle souche possède un niveau de PMF diminué et une réduction de l’efficacité d’import des aminoglycosides.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




