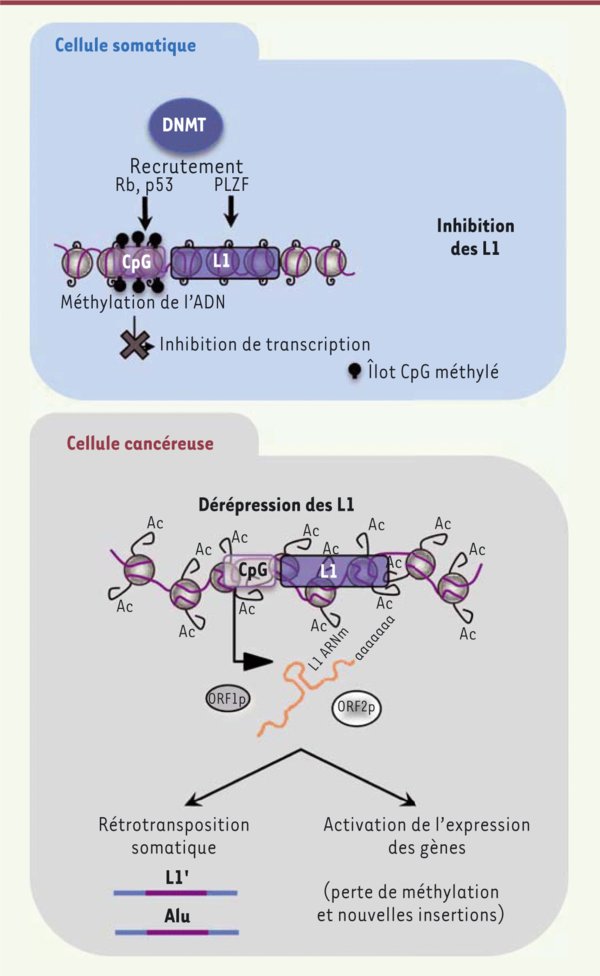
Figure 2.
Télécharger l'image originale
La méthylation de l’ADN inhibe l’expression des L1 alors que la perte de cette méthylation permet aux L1 d’être actifs. Dans les cellules somatiques normales, les protéines p53, Rb et PLZF induisent la méthylation des L1 en recrutant les protéines DNMT (DNA methyltransferase) et inhibent la rétrotransposition active des L1. Dans les cellules cancéreuses, l’hypométhylation (ou perte de méthylation), souvent associée à la perte d’expression ou de fonctions de ces protéines, est accompagnée d’une activation de la machinerie de rétrotransposition des L1. De nouveaux éléments d’insertion se produisent incluant les éléments transposables de type L1 (L1’) ou Alu. L’insertion de ces nouveaux éléments et la perte de méthylation des éléments transposables sont impliquées dans la dérégulation de l’expression génique (adapté de [12]).
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




