Figure 1.
Télécharger l'image originale
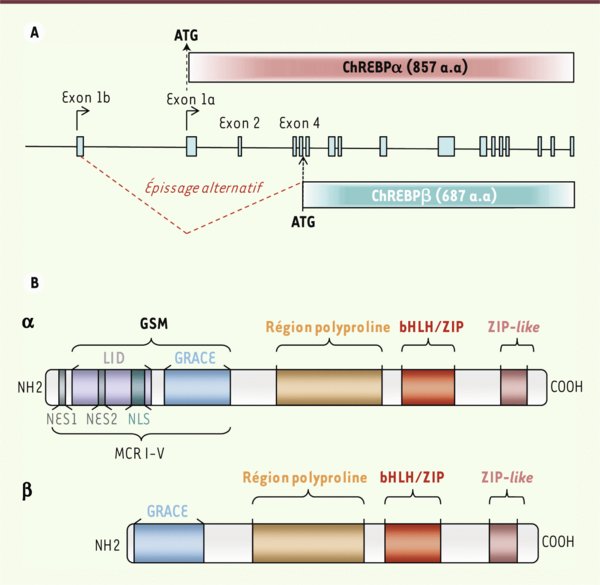
Représentations génique et protéique de ChREBP α et β. A. Epissage alternatif de ChREBPβ. ChREBPα est synthétisée à partir du site d’initiation de la traduction localisé dans l’exon 1a. ChREBPβ est transcrite à partir du promoteur situé dans l’exon 1b et par un épissage des exons 1a et 2. Le site d’initiation de la traduction est localisé dans l’exon 4 conduisant à la formation d’une protéine plus courte de 687 acides aminés. Adapté de [23]. B. Structure des protéines ChREBP α et β. La protéine ChREBPα (857 a.a) possède différents domaines dont un signal de localisation nucléaire (NLS), deux signaux d’export nucléaire (NES1 et NES2), un domaine riche en proline, un domaine bHLH/ZIP permettant l’interaction avec son partenaire d’hétérodimérisation Mlx et un domaine leucine-zipper-like. Une région très conservée et sensible au glucose (GSM) a été identifiée par analyse structure/fonction. Cette région est comprend les domaines LID (low-glucose inhibitory domain) et GRACE (glucose conserved response element). Le domaine LID est absent de la protéine courte ChREBPβ. MCR : mondo conserved region.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




