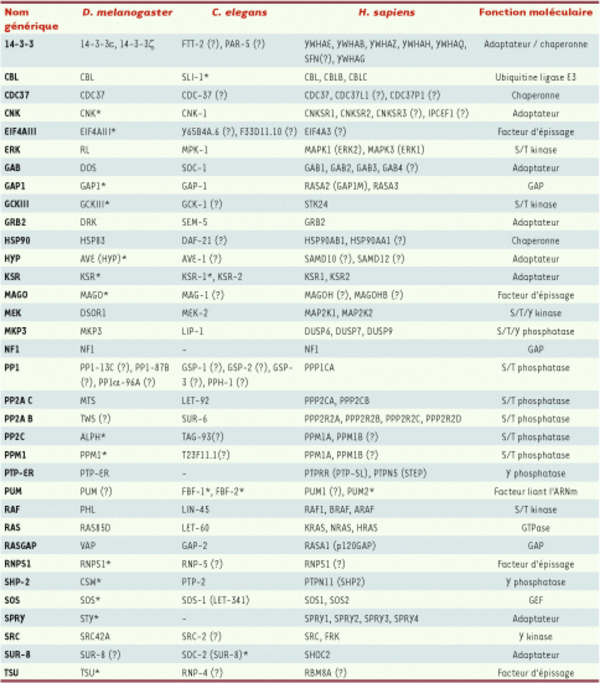
Tableau I
Composantes de l’axe RTK/RAS/ERK communes à la drosophile, au nématode et à l’humain. Les symboles génériques représentés ici sont utilisés dans l’article pour désigner les homologues des protéines à travers les espèces. Les symboles officiels correspondants pour les protéines de D. melanogaster (FlyBase), C. elegans (WormBase) et les gènes de H. sapiens (HGNC) sont listés ici. Les symboles alternatifs communément employés sont entre parenthèses. Les homologues ne se trouvant pas dans la littérature ont été identifiées à l’aide de l’outil HCOP du HGCN (www.genenames.org), Ensembl (www.ensembl.org) ou par recherche BLAST. Dans un but de simplicité, les homologues des RTK ne sont pas présentés ici. Les astérisques indiquent les protéines qui furent initialement associées à la signalisation RTK/RAS/ERK grâce à la drosophile ou au nématode. Un point d’interrogation est placé à la suite des homologues pour lesquels une fonction dans la voie MAPK n’a pas été décrite.
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.