Figure 1.
Télécharger l'image originale
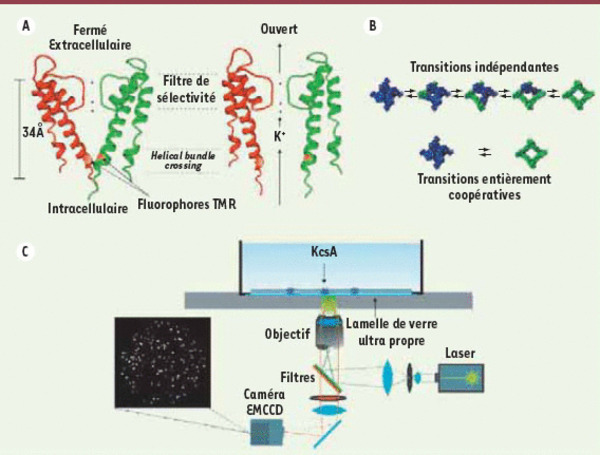
Détection de l’ouverture/fermeture du canal potassique bactérien. A. À gauche : représentation du canal potassique bactérien de Streptomyces lividans : KcsA (construit à partir de la structure cristalline de la protein data bank [pdb] ; code 1bl8). Le canal est dans l’état fermé et seulement deux des quatre sous-unités sont visibles (pour une image plus claire) selon une coupe transversale de la membrane cellulaire. Le canal est marqué sur chacune des quatre sous-unités dans la région du helical bundle crossing. Les autres sous-unités non visibles sont placées de manière symétrique (B). À droite : représentation de l’état ouvert (le canal n’a pas été cristallisé dans l’état ouvert, ce n’est donc qu’un modèle construit à partir de la représentation fermée). On peut y voir la conduction des ions K+ dans le pore. B. Illustrations de transitions d’ouverture et de fermeture du canal KcsA vu du dessus (on regarde au-dessus de la membrane cellulaire). Haut : transitions où les sous-unités sont indépendantes ; bas : transitions entièrement coopérative. C. Observation des canaux KcsA marqués au TMR dans une bicouche lipidique : un laser vert excite les fluorophores et un signal de fluorescence parvient à la caméra EMCCD, où plusieurs canaux unitaires sont reconnus individuellement.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




