Figure 1.
Télécharger l'image originale
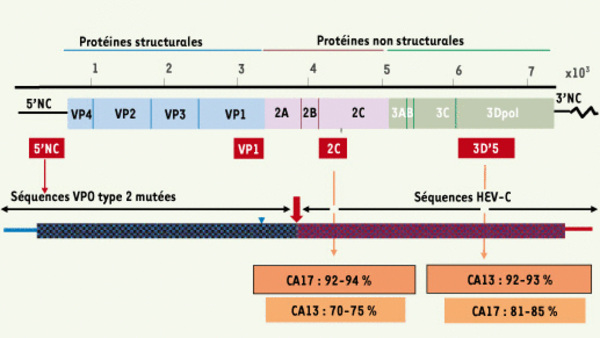
Structure génomique d’un VDPV recombinant isolé à Madagascar. L’organisation du génome des entérovirus est indiquée avec les régions 5’- et 3’-non codantes (5’ NC et 3’ NC). La phase ouverte de lecture codant les protéines de la capside du virion (VP1 à VP4) et les protéines impliquées dans la multiplication virale (2A à 3Dpol) ainsi que la taille du génome viral (× 103 nucléotides) sont représentés. La séquence nucléotidique du génome du VDPV ainsi que les séquences partielles de quatre régions génomiques (5’ NC, VP1, 2C et 3D’5) de tous les entérovirus non poliovirus co-circulant avec le VDPV ont été déterminées. Le génome du VDPV apparaît formé de séquences dérivées de la souche vaccinale de type 2 du VPO et de séquences d’HEV-C, le site de recombinaison étant localisé dans la protéine virale 2A (flèche rouge). Les séquences d’HEV-C apparaissent proches des séquences de souches de coxsackievirus A17 (CA17) dans la région 2C et de souches de coxsackievirus A13 (CA13) dans la région 3Dpol. Les pourcentages d’identité nucléotidique entre le VDPV et les souches de coxsackievirus sont indiqués.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




