Figure 1.
Télécharger l'image originale
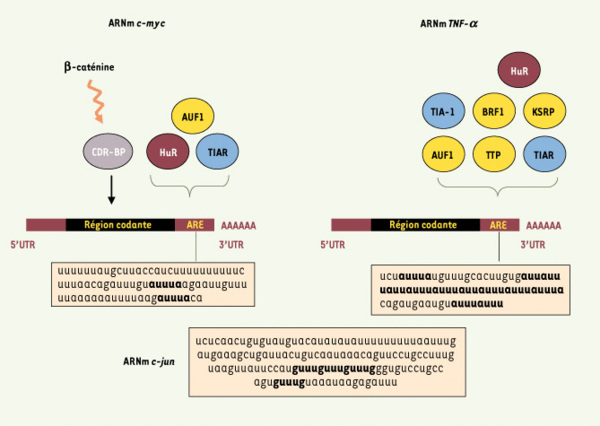
Diversité des séquences ARE et des AUBP avec lesquelles elles interagissent. La classification initiale des ARE en trois catégories [5] s’est enrichie de subdivisions de la catégorie II en 4 groupes suivant le nombre de pentamères AUUUA [4]. L’ARE de l’ARN c-myc (nt 2061-2141 NM_002467) illustre la catégorie I et celle du TNFa la catégorie II, groupe 3 (nt n° 1311-1385 du gène TNFα NM_000594). À titre de comparaison, l’ARE de c-jun, un exemple de la catégorie III qui ne contient pas de pentamère mais une région riche en uridines ainsi que plusieurs motifs GUUUG est indiqué (nt 2401-2542 du gène c-jun J04115). Deux AUUUA dispersés dans un contexte riche en uridines sont présents dans l’ARE de c-myc, alors que le pentamère se répète 8 fois, dont 6 de manière chevauchante, pour l’ARE du TNFa. Les AUBP interagissant avec ces 2 dernières séquences sont indiquées (en jaune les AUBP déstabilisantes, en rouge foncé la seule AUBP stabilisante et en bleu les régulateurs traductionnels). miR16 contient la séquence UAAAUAUU et peut donc s’apparier à plusieurs sites de l’ARE du TNFα [20]. La région codée par l’exon 3 de c-myc contient la séquence CRD reconnue par la protéine CRD-BP qui la protège d’action d’endonucléases (pour revue, voir [41]) et dont l’expression est activée par la voie β-caténine/Tcf.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




