Figure 1.
Télécharger l'image originale
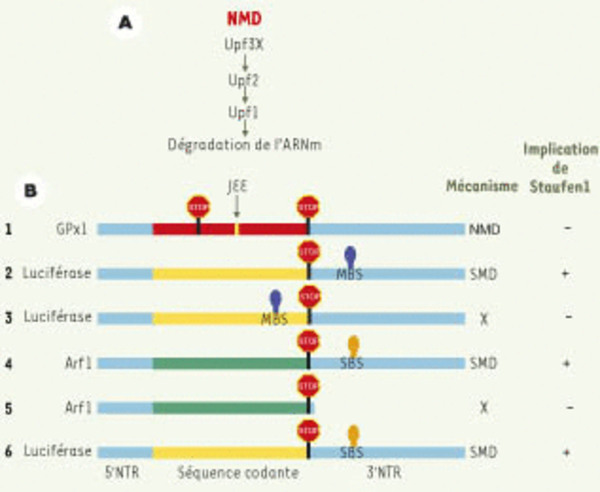
Comparaison des mécanismes NMD et SMD. A. Lors de l’épissage des transcrits primaires, un complexe protéique (EJC, exon junction complex) se dépose une vingtaine de nucléotides en amont des jonctions exon-exon (JEE). Si ce complexe composé de plusieurs protéines dont les facteurs Upf3X et Upf2 recrute le facteur Upf1, le NMD sera déclenché. B. (1) Les transcrits porteurs de codons non-sens apparus à la suite d’une erreur de transcription sont éliminés par le NMD. Staufen1 n’est pas impliqué dans ce mécanisme. (2-3) Le recrutement de Staufen1 au moyen d’une protéine de fusion Staufen1-MS2 (MS2 étant une protéine qui lie une structure spécifique de l’ARN) sur la séquence de reconnaissance MS2 (MBS, MS2-binding site) déclenche le SMD si celle-ci est placée en aval du codon normal de terminaison.(4) Staufen1, en se liant à une séquence d’ARN (SBS, Staufen1-binding site) située en aval du codon de terminaison normal sur le transcrit endogène Arf1 déclenche le SMD. (5) Cette séquence est importante puisque sa délétion protège le transcrit Arf1 de la dégradation. (6) De même, un transcrit rapporteur sur lequel a été fusionnée la séquence SBS devient sensible au SMD.
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




