Figure 2.
Télécharger l'image originale
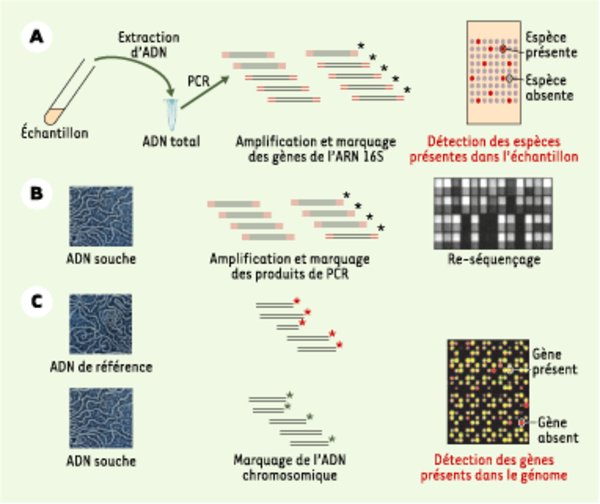
Trois applications des puces à ADN dans l’analyse microbiologique. A. Analyse d’une population bactérienne mixte. Après extraction de l’ADN, les régions codant pour l’ARN ribosomique 16S sont amplifiées pour toutes les bactéries de l’échantillon en utilisant des amorces universelles correspondant à des régions conservées dans toutes les espèces. La puce porte des oligonucléotides de séquence spécifique de chaque espèce. Après hybridation, la quantification du signal permet de détecter la présence de bactéries des différentes espèces représentées sur la puce et d’évaluer leurs quantités relatives. B. Puce Affymetrix de re-séquençage(http://www.affymetrix.com/) (http://www.affymetrix.com/). Ces puces portent plusieurs centaines de milliers d’oligonucléotides synthétisés in situ par une technique de photolithographie. Pour chaque base, quatre oligonucléotides sont synthétisés d’après une séquence de référence avec à la position centrale l’une des quatre bases, A, C, G ou T. La comparaison des signaux de fluorescence pour ces quatre oligonucléotides permet de déterminer la séquence à cette position. C. Détection de régions chromosomiques spécifiques d’un isolat. L’ADN de la souche à analyser et un ADN de référence sont marqués par des fluorophores ayant des propriétés spectrales différentes. La puce à ADN est hybridée avec un mélange des deux ADN marqués. Après analyse aux deux longueurs d’onde d’émission de fluorescence, les sondes absentes de la souche analysée n’émettront que pour la longueur d’onde du fluorophore de l’ADN de référence.
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




